一、一览

二、遗传算法流程

**种群初始:**随机生成一组N个DNA,每个DNA上都有S个基因,每个DNA都有自己的r值(适度值)。r最大的DNA为种群最优。
**选择操作:**将每个DNA的r值占总体的比例作为新一代选取的概率,再生成一组N个DNA,这些DNA都是从种群的概率选取来的。例如:上一代3号DNA的r3值占总r值为r3/r=1/6,则这一代中3号DNA就有大约1/6N个。
**交叉操作:**选择相邻的两个DNA,随机选择某段基因进行等位交换,让新的种群有更多的DNA样本。交叉率较高。
**变异操作:**随机选择DNA的某段基因进行变异。变异率较低。
三、算法介绍
3.1编码、解码
本文用一元函数模拟在指定区间求最值,函数y=f(x),x∈[lower,upper],保留Accuracy位小数点精度,可将区间划分(upper-lower)*10^Accuracy等份,因为需要用二进制编码,所以编码长度n=Log2((upper-lower)*10^Accuracy),此编码实质是将区间[lower,upper]内对应的实数x转化为一个二进制串:
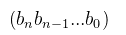
例如,保留精度6位,区间[-1,2],则串长n=22

我们知道不管是int类型还是long类型,我们可以直接转二进制,而且二进制的长度可以计算,如下:

但是,对于函数y=f(x),x∈[lower,upper],区间是未知的,说明有可能为负号,且对于实数x而言,在设计的时候为了追求精度,可能设计成单精度或双精度,那么该如何编码或解码?如何利用到上面已知的编码、解码方式?
我们知道数据很大的时候一般会把数据01化,如下公式:

这样的话就可以把实数x经过g(x)映射到[0,1]内,记为x->g(x),以就成了正数,但是还没有达到整数。
对于函数y=f(x),x∈[lower,upper],我们一般需要保留n位小数,那么我们就可以把g(x)扩大10^n 倍,为了统一编码长度,就应该扩大(upper-lower)*10^n 倍。
此时二进制串的长度=log2((upper-lower)*10^n),值x->G(x)=g(x)*(upper-lower)*10^n ,这样既保证了长度,也保证了符号。(这里的n指精度)
代码实现
 geneMinLength指二进制串长度,代码如下:
geneMinLength指二进制串长度,代码如下:

3.2种群初始化
PopulationSize为种群大小,在区间[lower,upper]上随机生成实数x,然后编码、计算适应度值,如下:

3.3选择操作
我们将r值(适应度值)所占总体的比例计算出来,然后利用累积概率随机选择DNA。r值(适应度值)所占总体的比例越大,越容易被选择。选择操作时保留精英代,下一次操作直接加入种群。

3.4交叉操作
 3.5变异操作:
3.5变异操作:
这里采用多点变异,变异时直接取反,例如:0->1,1->0

3.6适应度函数
适应度函数采用目标函数+指数函数,如下:

这里采用目标函数外,为什么还需要用指数函数?因为我们在选取时,需要适应度为正数且函数值有正负,例如在指定区间找最小值,越小的值(有正负),指数函数(0<a<1)映射的值越大,以就是权重越大,被选择的概率就越大。
指数函数:

经过一次迭代后再次回到3.3选择操作,直到迭代结束。
迭代代码如下:

这里种群大小为50,迭代200,交叉率0.75,变异率0.05作为测试。