🎓 作者:计算机毕设小月哥 | 软件开发专家
🖥️ 简介:8年计算机软件程序开发经验。精通Java、Python、微信小程序、安卓、大数据、PHP、.NET|C#、Golang等技术栈。
🛠️ 专业服务 🛠️
- 需求定制化开发
- 源码提供与讲解
- 技术文档撰写(指导计算机毕设选题【新颖+创新】、任务书、开题报告、文献综述、外文翻译等)
- 项目答辩演示PPT制作
🌟 欢迎:点赞 👍 收藏 ⭐ 评论 📝👇🏻 精选专栏推荐 👇🏻 欢迎订阅关注!
大数据实战项目
PHP|C#.NET|Golang实战项目
微信小程序|安卓实战项目
Python实战项目
Java实战项目🍅 ↓↓主页获取源码联系↓↓🍅
这里写目录标题
- 基于大数据的痴呆症预测数据可视化分析系统-功能介绍
- 基于大数据的痴呆症预测数据可视化分析系统-选题背景意义
- 基于大数据的痴呆症预测数据可视化分析系统-技术选型
- 基于大数据的痴呆症预测数据可视化分析系统-视频展示
- 基于大数据的痴呆症预测数据可视化分析系统-图片展示
- 基于大数据的痴呆症预测数据可视化分析系统-代码展示
- 基于大数据的痴呆症预测数据可视化分析系统-结语
基于大数据的痴呆症预测数据可视化分析系统-功能介绍
基于大数据的痴呆症预测数据可视化分析系统是一套集成Hadoop分布式存储、Spark大数据处理、Vue前端展示的综合性分析平台。系统采用Hadoop+Spark作为核心大数据处理引擎,通过HDFS分布式文件系统存储海量医疗数据,利用Spark SQL进行高效的数据清洗与特征工程处理。后端基于Spring Boot框架构建RESTful API服务,结合MyBatis进行数据持久化操作,前端采用Vue+ElementUI+ECharts技术栈实现交互式数据可视化界面。系统主要功能涵盖总体人口学特征分析、核心临床指标与认知功能分析、脑部影像学特征分析以及痴呆转化过程追踪等四大维度,能够对痴呆症相关的年龄、性别、教育水平、MMSE评分、CDR评定、脑体积等多维度指标进行深度挖掘分析。通过Pandas、NumPy等数据科学库进行统计计算,最终以直观的图表形式展现不同诊断状态下各项指标的分布规律与相关性,为医疗研究人员提供便捷的数据分析工具。
基于大数据的痴呆症预测数据可视化分析系统-选题背景意义
选题背景
随着全球人口老龄化趋势的加剧,痴呆症已成为影响老年人生活质量的重要疾病之一。痴呆症的早期诊断和预测对于延缓病情进展、改善患者生活质量具有重要价值。传统的痴呆症研究主要依赖小样本的临床数据分析,在数据处理效率和分析深度方面存在明显不足。现有的医疗数据分析系统大多基于传统的关系型数据库和单机处理模式,面对海量的医疗影像数据、认知评估数据以及纵向追踪数据时,往往出现处理速度慢、存储成本高、分析维度单一等问题。医疗机构迫切需要能够高效处理大规模数据集的分析工具,以便从多维度、多时间点的数据中挖掘出痴呆症发生发展的规律。大数据技术的成熟为解决这一问题提供了新的思路,Hadoop生态系统的分布式存储和Spark的内存计算能力,为处理复杂医疗数据提供了强有力的技术支撑。
选题意义
本系统的开发具有多方面的实际意义。从技术角度来看,系统验证了大数据技术在医疗数据分析领域的可行性,展示了Hadoop+Spark架构在处理结构化医疗数据方面的优势,为后续类似系统的开发提供了参考框架。从应用价值来说,系统能够帮助医疗研究人员快速完成大规模痴呆症数据的统计分析工作,提高了数据处理效率,降低了人工分析的时间成本。系统的可视化功能使复杂的医疗数据以直观的图表形式呈现,便于研究人员发现数据中的潜在规律和异常模式。从学习角度而言,本系统的开发过程涵盖了大数据存储、分布式计算、数据可视化等多个技术栈的综合运用,对于掌握现代数据处理技术具有良好的实践价值。虽然作为毕业设计项目,系统在功能完善度和性能优化方面还有提升空间,但其在整合多种技术栈、解决实际数据分析需求方面仍具有一定的参考意义,为今后从事相关技术开发工作奠定了基础。
基于大数据的痴呆症预测数据可视化分析系统-技术选型
大数据框架:Hadoop+Spark(本次没用Hive,支持定制)
开发语言:Python+Java(两个版本都支持)
后端框架:Django+Spring Boot(Spring+SpringMVC+Mybatis)(两个版本都支持)
前端:Vue+ElementUI+Echarts+HTML+CSS+JavaScript+jQuery
详细技术点:Hadoop、HDFS、Spark、Spark SQL、Pandas、NumPy
数据库:MySQL
基于大数据的痴呆症预测数据可视化分析系统-视频展示
计算机毕设推荐:痴呆症预测可视化系统Hadoop+Spark+Vue技术栈详解
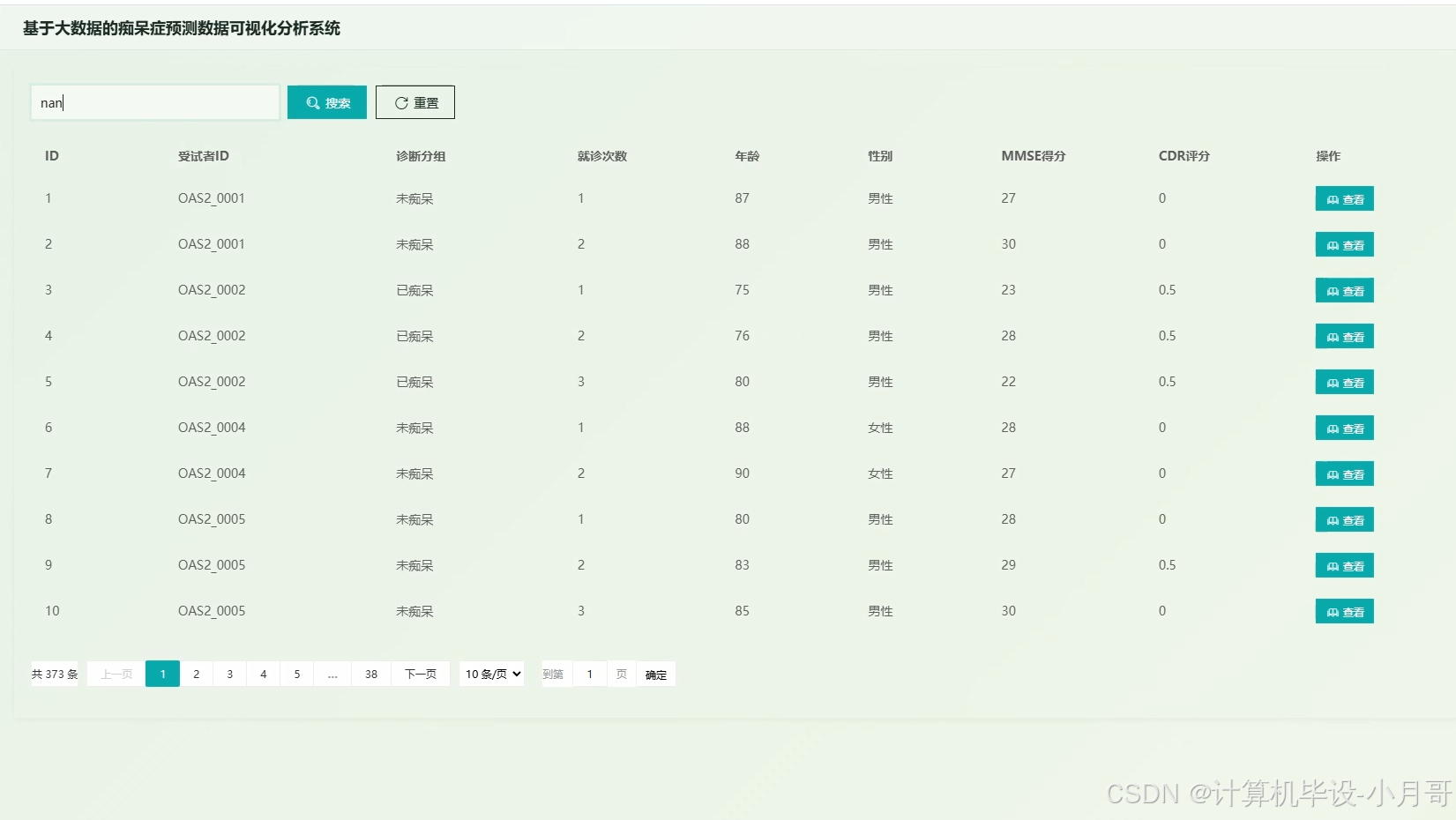
基于大数据的痴呆症预测数据可视化分析系统-图片展示
登录
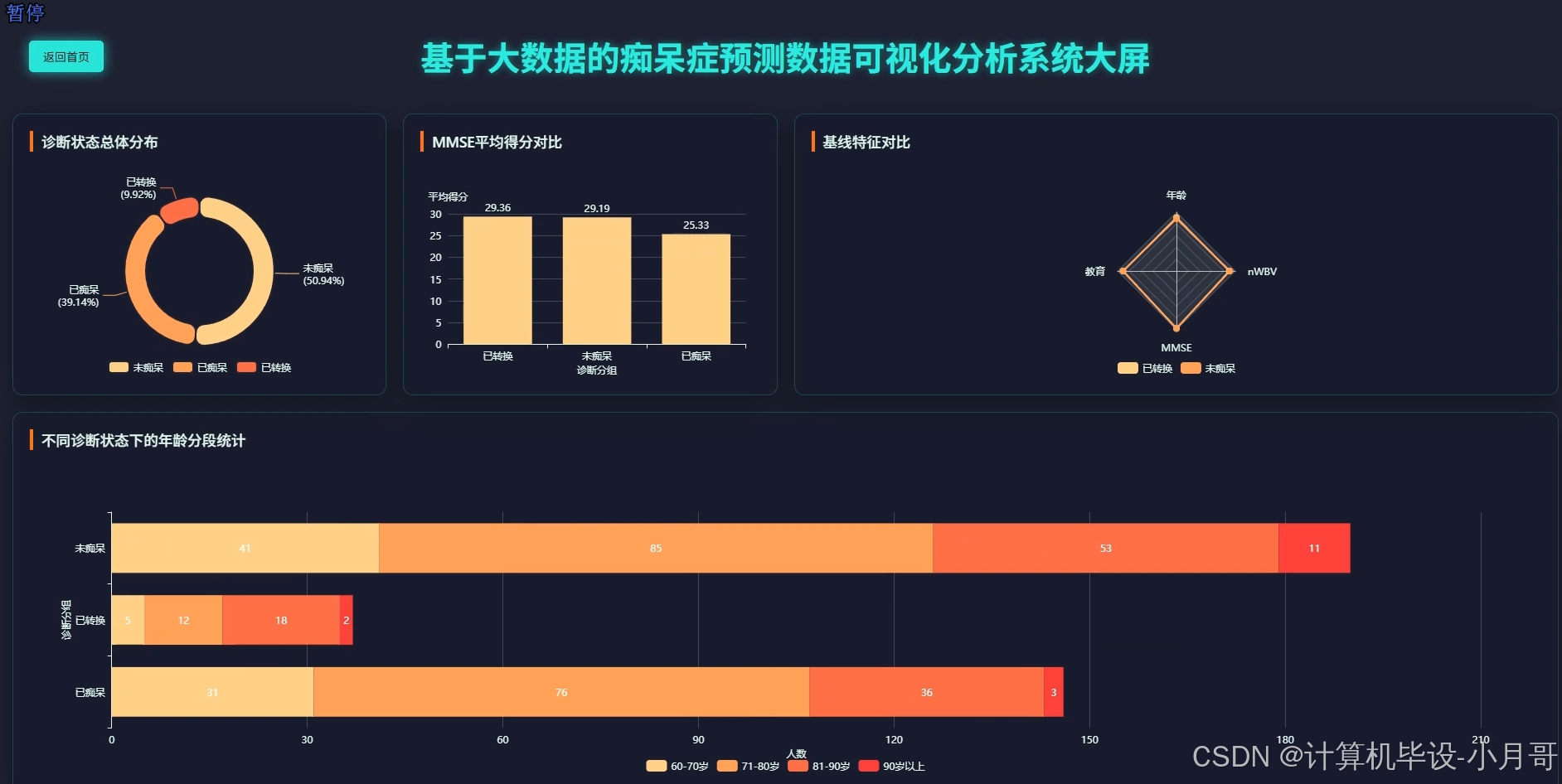
大屏上
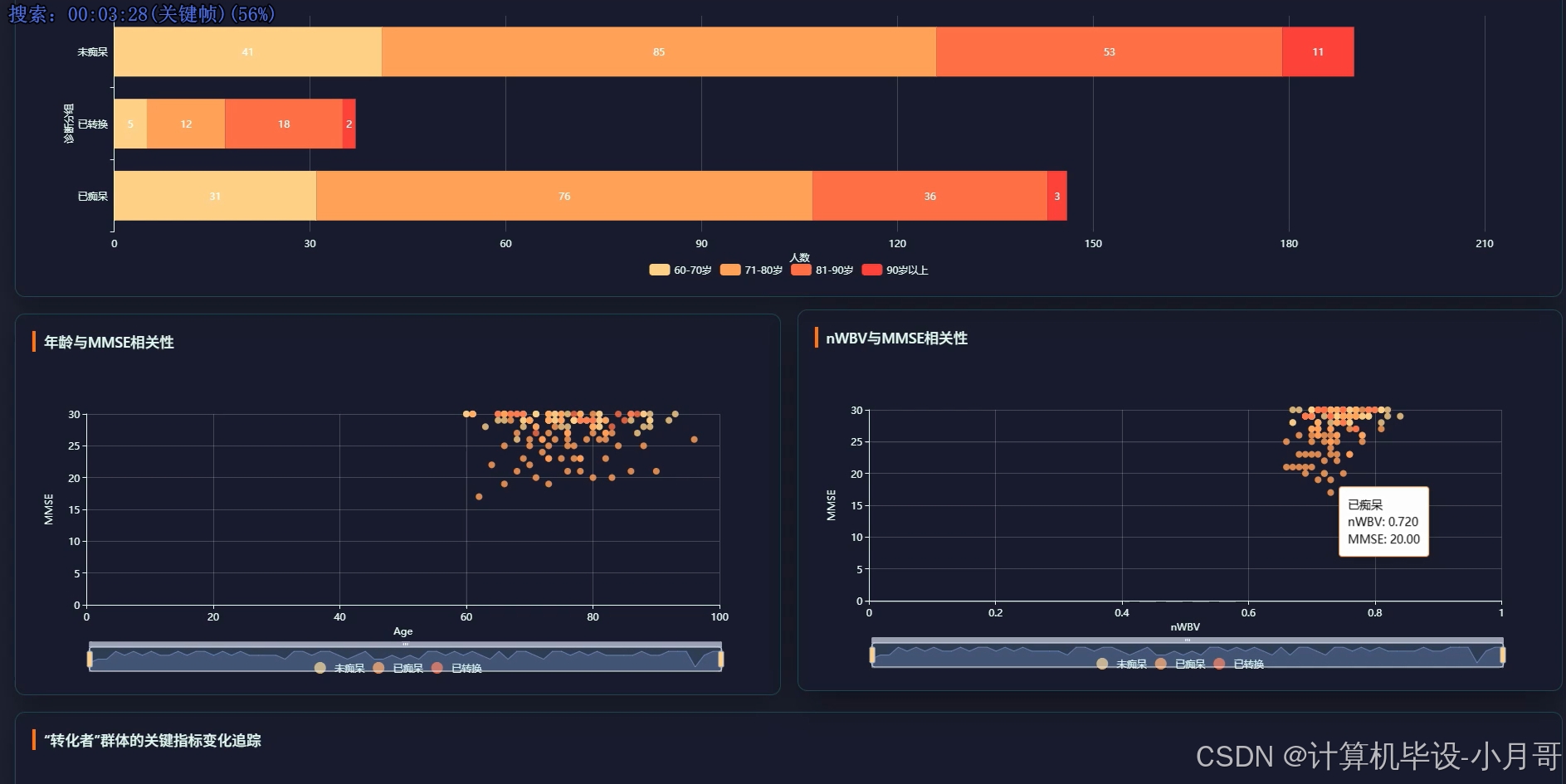
大屏中
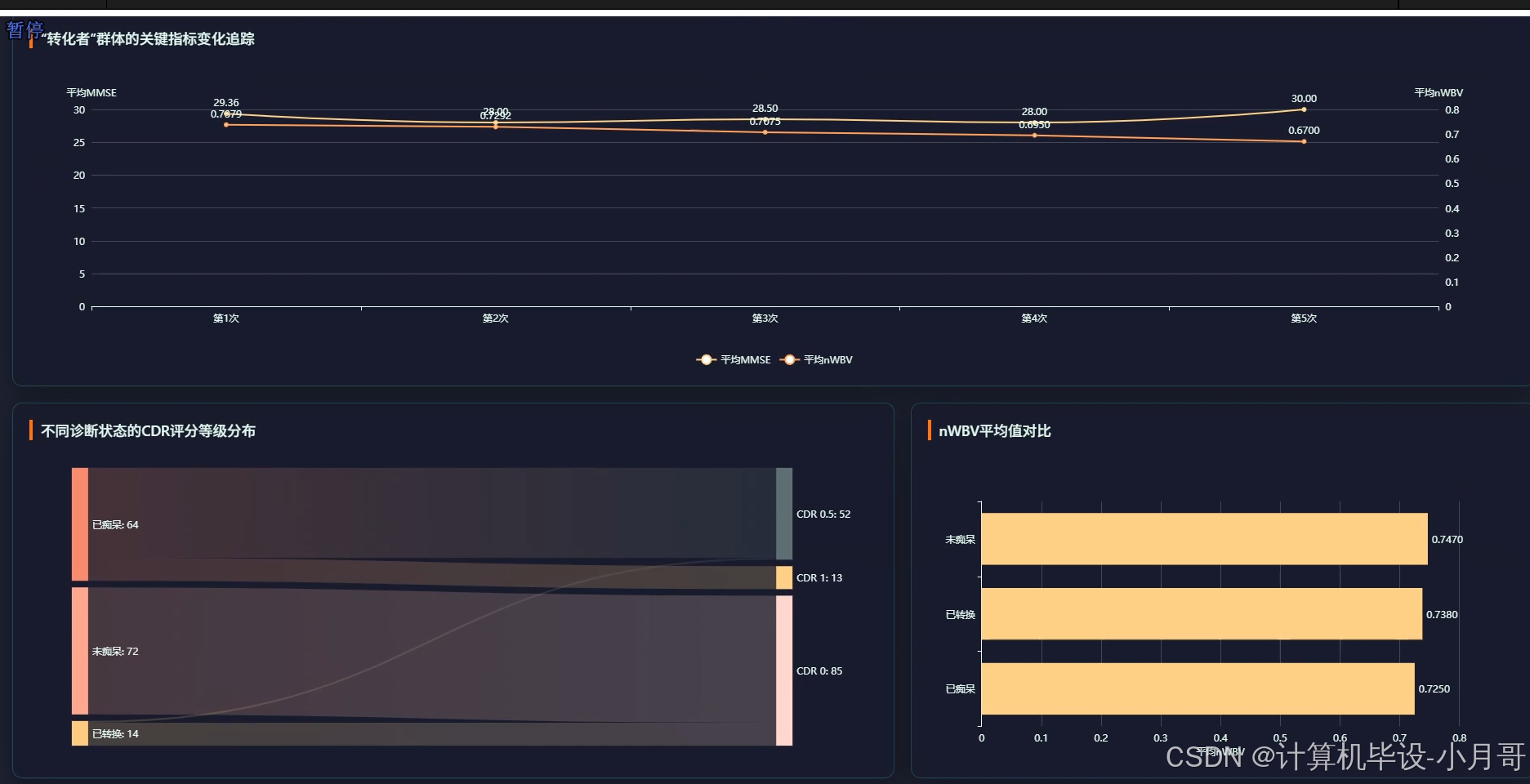
大屏下
痴呆症数据总览
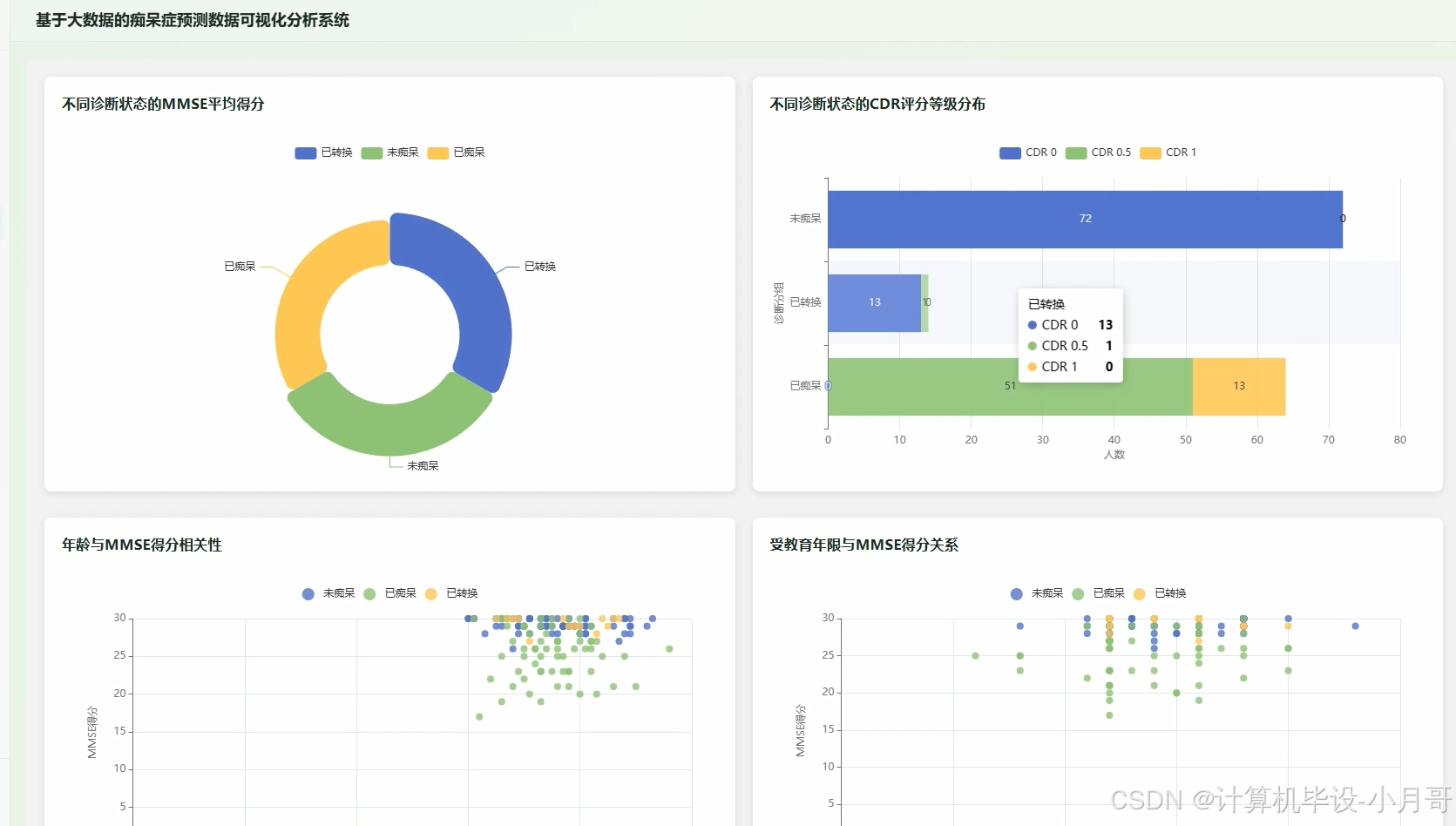
临床特征分析
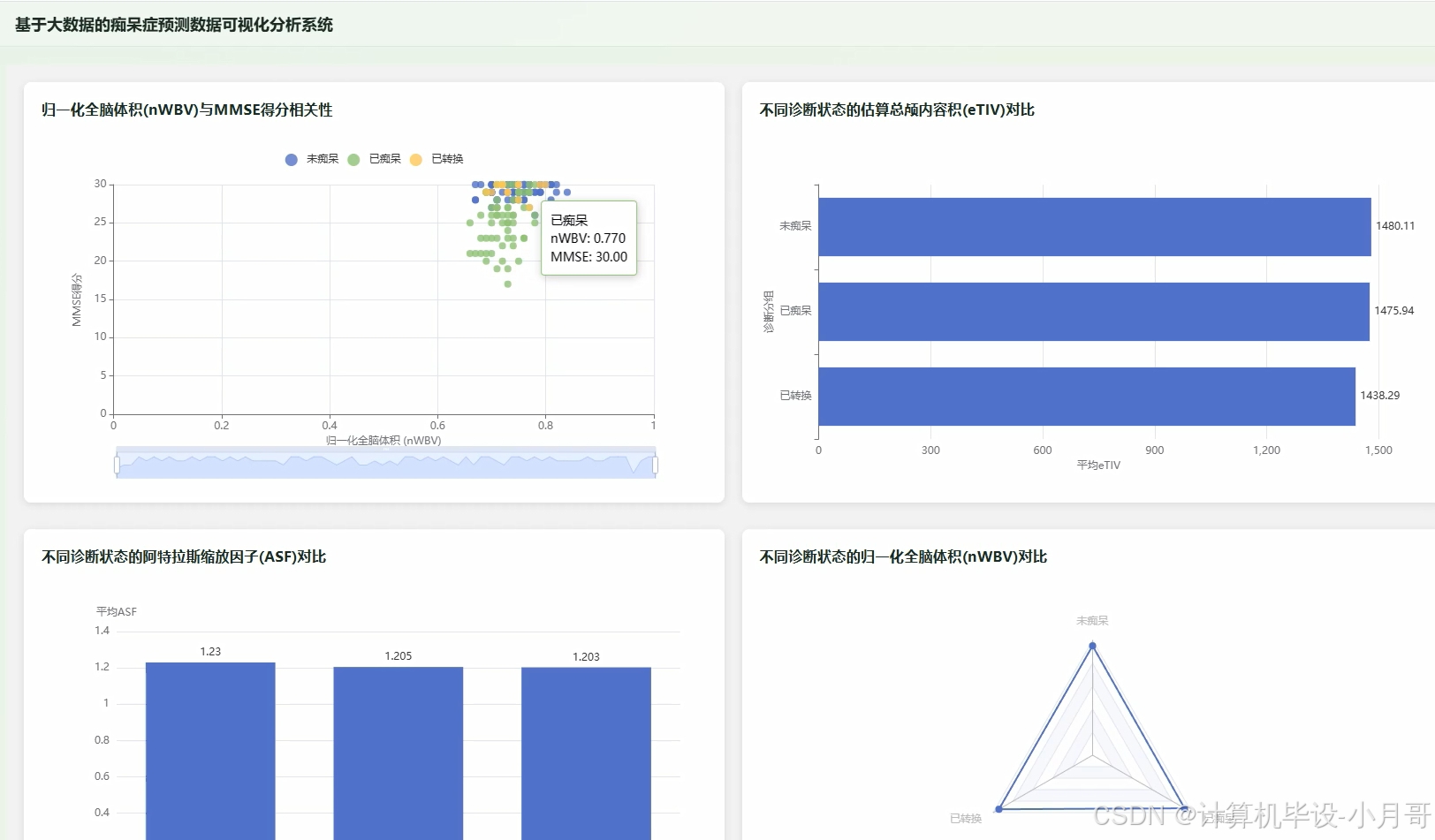
脑影像分析
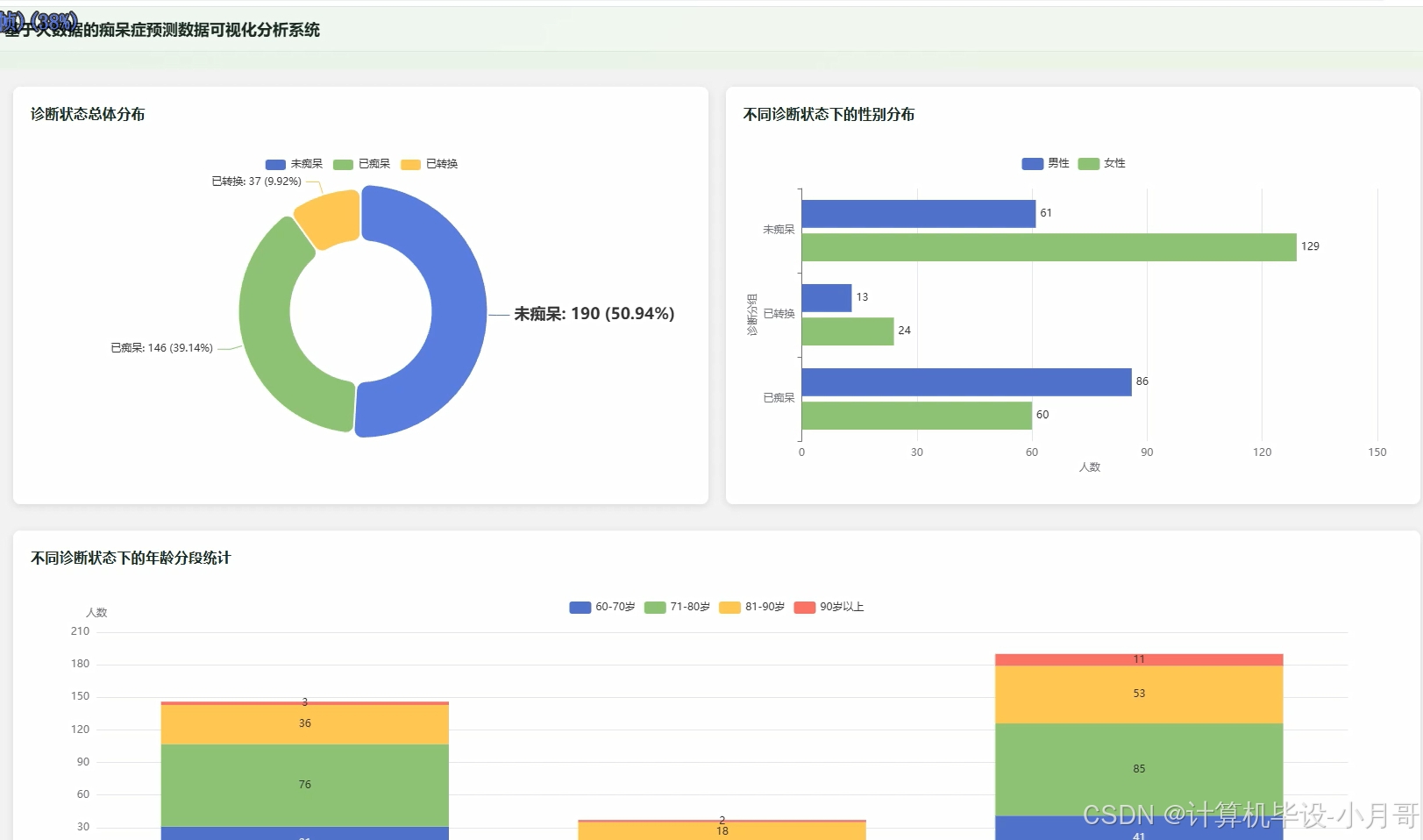
人群特征分析
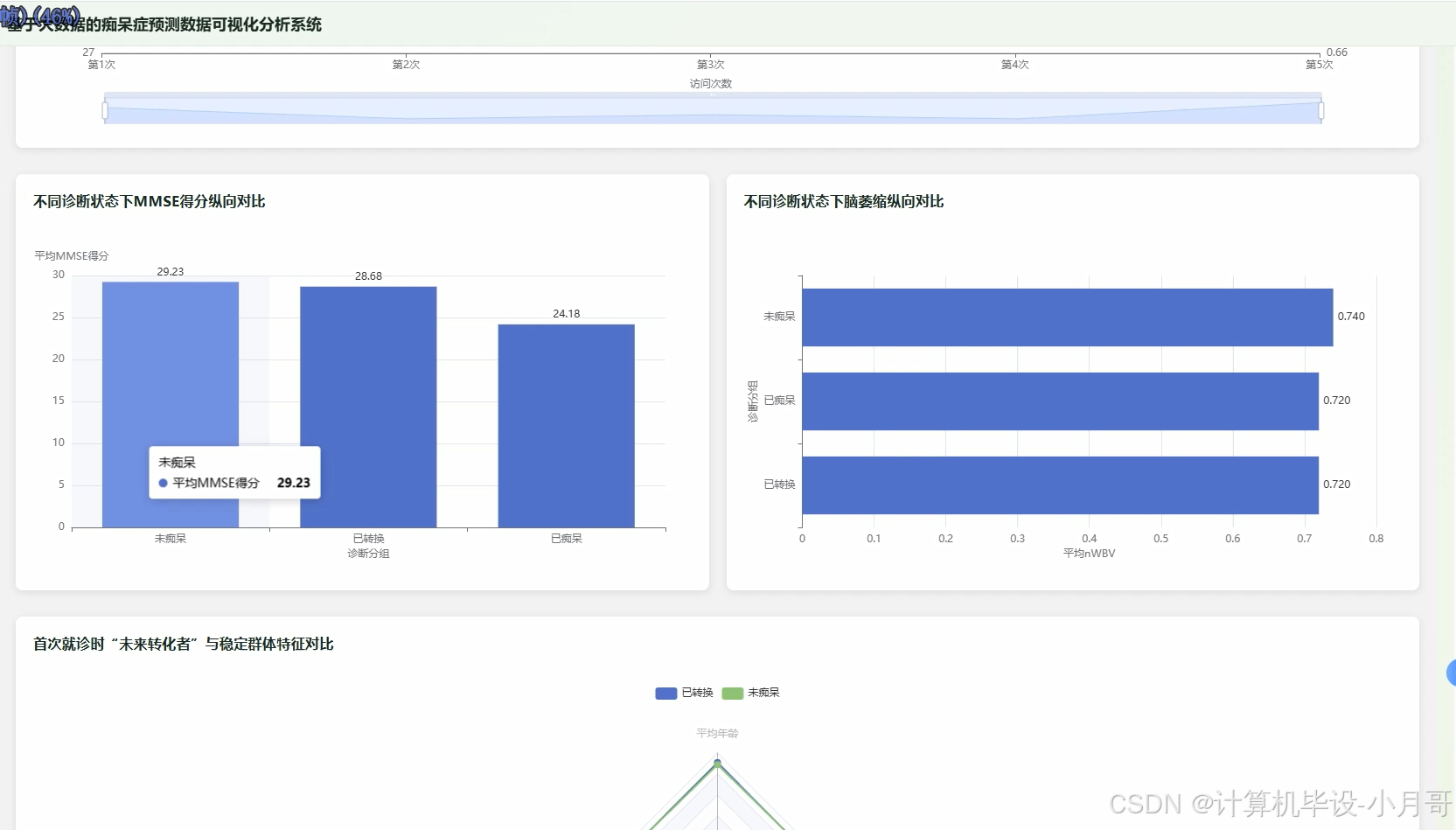
纵向追踪分析
基于大数据的痴呆症预测数据可视化分析系统-代码展示
python
from pyspark.sql import SparkSession
from pyspark.sql.functions import col, count, avg, stddev, corr, when, isnan, isnull
import pandas as pd
import numpy as np
from django.http import JsonResponse
from django.views.decorators.csrf import csrf_exempt
import json
spark = SparkSession.builder.appName("DementiaAnalysis").master("local[*]").getOrCreate()
@csrf_exempt
def demographic_analysis(request):
df = spark.read.option("header", "true").option("inferSchema", "true").csv("/data/cleaned_dementia_data.csv")
group_distribution = df.groupBy("Group").agg(count("*").alias("count")).collect()
total_count = df.count()
distribution_data = []
for row in group_distribution:
percentage = (row['count'] / total_count) * 100
distribution_data.append({
'group': row['Group'],
'count': row['count'],
'percentage': round(percentage, 2)
})
gender_group_analysis = df.groupBy("Group", "M/F").agg(count("*").alias("count")).collect()
gender_data = {}
for row in gender_group_analysis:
if row['Group'] not in gender_data:
gender_data[row['Group']] = {}
gender_data[row['Group']][row['M/F']] = row['count']
age_bins = [60, 70, 80, 90, 100]
age_binned_df = df.withColumn("age_group",
when(col("Age") < 70, "60-70").
when(col("Age") < 80, "71-80").
when(col("Age") < 90, "81-90").
otherwise("90+"))
age_distribution = age_binned_df.groupBy("Group", "age_group").agg(count("*").alias("count")).collect()
age_data = {}
for row in age_distribution:
if row['Group'] not in age_data:
age_data[row['Group']] = {}
age_data[row['Group']][row['age_group']] = row['count']
education_stats = df.groupBy("Group").agg(avg("EDUC").alias("avg_education"), stddev("EDUC").alias("std_education")).collect()
education_data = []
for row in education_stats:
education_data.append({
'group': row['Group'],
'avg_education': round(row['avg_education'], 2),
'std_education': round(row['std_education'], 2) if row['std_education'] else 0
})
ses_distribution = df.groupBy("Group", "SES").agg(count("*").alias("count")).collect()
ses_data = {}
for row in ses_distribution:
if row['Group'] not in ses_data:
ses_data[row['Group']] = {}
ses_data[row['Group']][row['SES']] = row['count']
result = {
'group_distribution': distribution_data,
'gender_analysis': gender_data,
'age_distribution': age_data,
'education_stats': education_data,
'ses_distribution': ses_data
}
return JsonResponse(result)
@csrf_exempt
def clinical_indicators_analysis(request):
df = spark.read.option("header", "true").option("inferSchema", "true").csv("/data/cleaned_dementia_data.csv")
mmse_stats = df.groupBy("Group").agg(
avg("MMSE").alias("avg_mmse"),
stddev("MMSE").alias("std_mmse"),
count("MMSE").alias("count")
).collect()
mmse_data = []
for row in mmse_stats:
mmse_data.append({
'group': row['Group'],
'avg_mmse': round(row['avg_mmse'], 2),
'std_mmse': round(row['std_mmse'], 2) if row['std_mmse'] else 0,
'count': row['count']
})
cdr_distribution = df.groupBy("Group", "CDR").agg(count("*").alias("count")).collect()
cdr_data = {}
for row in cdr_distribution:
if row['Group'] not in cdr_data:
cdr_data[row['Group']] = {}
cdr_data[row['Group']][str(row['CDR'])] = row['count']
age_mmse_correlation = df.select("Age", "MMSE", "Group").filter(col("MMSE").isNotNull()).collect()
correlation_data = []
for row in age_mmse_correlation:
correlation_data.append({
'age': row['Age'],
'mmse': row['MMSE'],
'group': row['Group']
})
overall_age_mmse_corr = df.stat.corr("Age", "MMSE")
education_mmse_correlation = df.select("EDUC", "MMSE", "Group").filter(col("MMSE").isNotNull()).collect()
educ_correlation_data = []
for row in education_mmse_correlation:
educ_correlation_data.append({
'education': row['EDUC'],
'mmse': row['MMSE'],
'group': row['Group']
})
overall_educ_mmse_corr = df.stat.corr("EDUC", "MMSE")
group_correlations = {}
for group in ['Demented', 'Nondemented', 'Converted']:
group_df = df.filter(col("Group") == group)
if group_df.count() > 1:
age_mmse_corr = group_df.stat.corr("Age", "MMSE")
educ_mmse_corr = group_df.stat.corr("EDUC", "MMSE")
group_correlations[group] = {
'age_mmse_correlation': round(age_mmse_corr, 3) if age_mmse_corr else 0,
'education_mmse_correlation': round(educ_mmse_corr, 3) if educ_mmse_corr else 0
}
result = {
'mmse_statistics': mmse_data,
'cdr_distribution': cdr_data,
'age_mmse_scatter': correlation_data,
'education_mmse_scatter': educ_correlation_data,
'overall_correlations': {
'age_mmse': round(overall_age_mmse_corr, 3) if overall_age_mmse_corr else 0,
'education_mmse': round(overall_educ_mmse_corr, 3) if overall_educ_mmse_corr else 0
},
'group_correlations': group_correlations
}
return JsonResponse(result)
@csrf_exempt
def brain_imaging_analysis(request):
df = spark.read.option("header", "true").option("inferSchema", "true").csv("/data/cleaned_dementia_data.csv")
nwbv_stats = df.groupBy("Group").agg(
avg("nWBV").alias("avg_nwbv"),
stddev("nWBV").alias("std_nwbv"),
count("nWBV").alias("count")
).collect()
nwbv_data = []
for row in nwbv_stats:
nwbv_data.append({
'group': row['Group'],
'avg_nwbv': round(row['avg_nwbv'], 4),
'std_nwbv': round(row['std_nwbv'], 4) if row['std_nwbv'] else 0,
'count': row['count']
})
etiv_stats = df.groupBy("Group").agg(
avg("eTIV").alias("avg_etiv"),
stddev("eTIV").alias("std_etiv"),
count("eTIV").alias("count")
).collect()
etiv_data = []
for row in etiv_stats:
etiv_data.append({
'group': row['Group'],
'avg_etiv': round(row['avg_etiv'], 2),
'std_etiv': round(row['std_etiv'], 2) if row['std_etiv'] else 0,
'count': row['count']
})
asf_stats = df.groupBy("Group").agg(
avg("ASF").alias("avg_asf"),
stddev("ASF").alias("std_asf"),
count("ASF").alias("count")
).collect()
asf_data = []
for row in asf_stats:
asf_data.append({
'group': row['Group'],
'avg_asf': round(row['avg_asf'], 4),
'std_asf': round(row['std_asf'], 4) if row['std_asf'] else 0,
'count': row['count']
})
nwbv_mmse_correlation = df.select("nWBV", "MMSE", "Group").filter(col("nWBV").isNotNull() & col("MMSE").isNotNull()).collect()
nwbv_mmse_data = []
for row in nwbv_mmse_correlation:
nwbv_mmse_data.append({
'nwbv': round(row['nWBV'], 4),
'mmse': row['MMSE'],
'group': row['Group']
})
overall_nwbv_mmse_corr = df.stat.corr("nWBV", "MMSE")
group_brain_correlations = {}
for group in ['Demented', 'Nondemented', 'Converted']:
group_df = df.filter(col("Group") == group)
if group_df.count() > 1:
nwbv_mmse_corr = group_df.stat.corr("nWBV", "MMSE")
group_brain_correlations[group] = {
'nwbv_mmse_correlation': round(nwbv_mmse_corr, 3) if nwbv_mmse_corr else 0
}
brain_volume_comparison = df.select("Group", "nWBV", "eTIV", "ASF").collect()
volume_comparison_data = []
for row in brain_volume_comparison:
volume_comparison_data.append({
'group': row['Group'],
'nwbv': round(row['nWBV'], 4) if row['nWBV'] else 0,
'etiv': round(row['eTIV'], 2) if row['eTIV'] else 0,
'asf': round(row['ASF'], 4) if row['ASF'] else 0
})
result = {
'nwbv_statistics': nwbv_data,
'etiv_statistics': etiv_data,
'asf_statistics': asf_data,
'nwbv_mmse_correlation': nwbv_mmse_data,
'overall_brain_correlation': {
'nwbv_mmse': round(overall_nwbv_mmse_corr, 3) if overall_nwbv_mmse_corr else 0
},
'group_brain_correlations': group_brain_correlations,
'volume_comparison': volume_comparison_data
}
return JsonResponse(result)基于大数据的痴呆症预测数据可视化分析系统-结语
🌟 欢迎:点赞 👍 收藏 ⭐ 评论 📝
👇🏻 精选专栏推荐 👇🏻 欢迎订阅关注!
大数据实战项目
PHP|C#.NET|Golang实战项目
微信小程序|安卓实战项目
Python实战项目
Java实战项目🍅 ↓↓主页获取源码联系↓↓🍅