1.miniconda3的安装
官网:Miniconda --- Conda documentation
找到对应系统、Python版本下载
wget https://mirrors.ustc.edu.cn/anaconda/miniconda/Miniconda3-latest-Linux-x86_64.sh
#wget -c https://repo.continuum.io/miniconda/Miniconda3-latest-Linux-x86_64.sh
bash Miniconda-latest-Linux-x86_64.sh2. 设置conda镜像源
依次输入以下命令设置软件镜像源,并展示镜像源地址,一般设下载地址为清华,也可以选择其他。注意,要用 bioconda 源,必须先设置 conda-forge 源,并且 conda-forge 源优先级是最高的
清华:anaconda | 镜像站使用帮助 | 清华大学开源软件镜像站 | Tsinghua Open Source Mirror
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/
conda config --set show_channel_urls yes北外镜像:
conda config --add channels https://mirrors.bfsu.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.bfsu.edu.cn/anaconda/pkgs/main/
conda config --add channels https://mirrors.bfsu.edu.cn/anaconda/pkgs/r/
conda config --add channels https://mirrors.bfsu.edu.cn/anaconda/cloud/bioconda/
conda config --add channels https://mirrors.bfsu.edu.cn/anaconda/cloud/conda-forge/
conda config --set show_channel_urls yes中科大:
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/pkgs/pro
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/pkgs/r
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/pkgs/free
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/pkgs/main
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/cloud/bioconda
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/cloud/conda-forge
conda config --set show_channel_urls yes其他一些命令:
conda config --show channels #显示镜像源
conda config --show-sources #显示镜像源
conda config --remove channels https://*** #删除镜像源3. 创建流程环境
-
创建rna流程环境,指定python版本为3,同时下载sra-tools 软件
conda create -n rna_p3 python=3 sra-tools
conda env list #查看环境
conda activate rna_p3 #进入conda 环境
conda deactivate #退出当前conda环境 -
如果想重命名环境:先新建clone再进行删除
conda create -n rna --clone rna_p3
conda remove -n rna_p3 --all
4. 软件的下载管理
conda安装包在线查询 https://anaconda.org/
conda install (-y)软件名 # 安装软件
conda install 软件名=版本号 # 安装特定版本软件
conda search 软件名 #搜索软件
conda list # 列出已安装软件
conda update 软件名 #更新软件
conda remove 软件名 #卸载软件5. 在脚本中切换conda环境
若想在脚本中切换conda环境,脚本内容如下,脚本执行完后会自动退回当前环境
source ~/miniconda3/bin/activate rna_p3
python --version
conda info --envs
#####命令
python --version
conda info --envs或者先在脚本执行source ~/miniconda3/etc/profile.d/conda.sh,再使用conda命令
6. 环境迁移:导出安装包列表成为yaml文件
如果想要迁移环境中的安装包到其他计算机上,可以导出yaml文件
conda env export --file rna_p3_env.yaml --name rna_p3
conda env create -f rna_p3_env.yaml2.安装R环境
1. 提前准备好conda的R单独环境
conda env list #查看已有环境
conda create -n R4.1.2
conda activate R4.1.2
#激活环境
source activate R4.1.2
2. 进入环境后,多添加一些conda channel
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda
conda config --set show_channel_urls yes
conda config --show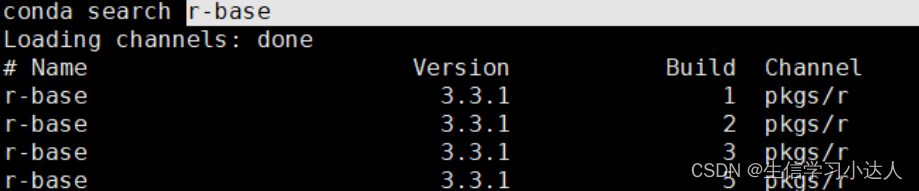
选择一款适合的版本
conda install r-base=4.1.2
which -a R将自己刚才安装的R绝对路径添加到.bashrc中的环境变量中$PATH,也可以加alias。
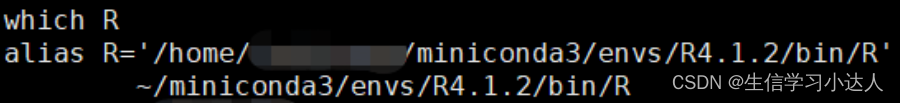
如今,R的环境已经安装好了。
3.安装R包
options()$repos #查看现有镜像file.edit(file.path("~",".Rprofile")) #打开一个文件
在文件中写入下面的内容 #多写几个镜像
options("repos"=c(CRAN="https://mirrors.pku.edu.cn/CRAN/","The Comprehensive R Archive Network","http://mirrors.aliyun.com/CRAN"))
print("已设置北大阿里云镜像")
写完后,退出()
#退出R语言
q()
#重新进行R
R重新进入R
options()$repos #查看现有镜像
- 安装BiocManager ##官网Bioconductor - Install
R4.1.2对应的BiocManager版本是3.14
在R中输入
if (!require("BiocManager",quietly = TRUE))
install.packages("BiocManager")
BiocManager::install(version ="3.14")
- 安装R包
方法一:>install.packages("package_name")
方法二:
> library(BiocManager)
> BiocManager::install("ggplot2")
OK,此时你可以安装自己想要的R包了。
#附些常见的命令,查看已安装的R包"library()"。加载包"library(packagename)"。查看已加载的包"(.packages())"。取消已加载的包"detach("package: packagename")"。
例如安装一些常见的R包:
安装:install.packages("dplyr")
加载:library(dplyr)
查看帮助文档(函数的不同参数使用说明):?dplyr