- 博主简介:努力学习的22级计算机科学与技术本科生一枚🌸
- 博主主页: @Yaoyao2024
- 往期回顾:【AlphaFold2】深入浅出,讲解Evoformer|学习笔记
- 每日一言🌼: 永远不要只看见前方路途遥远 而忘了从前的自己坚持了多久才走到这里。🌺
前言
博主刚接触AI for Biology,这个领域有很多需要掌握且重要的分析工具(eg:Phenix/Chimerax),网上教程少之又少,英文的文档对初学者来说毕竟还是难以一下子理解和掌握。故决定把使用过程中的收获和使用方法记录下来,方便这个领域的同行以及后来者参阅。
关于Phenix的命令行方法使用的文档,我目前参阅的是这两个
- https://phenix-online.org/version_docs/1.17.1-3660/overviews/validation_cryo_em.html
- https://phenix-online.org/documentation/
一、Phenix介绍
https://phenix-online.org/documentation/
phenix 是一个用于大分子晶体学研究的综合性软件包,它和 Python 之间存在紧密的联系,下面详细解释 phenix 与 Python 的关系,以及它与虚拟环境中 Python 的关联。
1. phenix 与 Python 的关系
-
基于 Python 开发 :
phenix是使用 Python 语言开发的,Python 作为一种高级、通用、解释型的编程语言,具有简洁易读的语法和丰富的库,适合用于快速开发和实现复杂的算法。phenix借助 Python 构建了用户界面、脚本接口以及各种功能模块,使得用户可以通过编写 Python 脚本来调用phenix的功能,实现自动化的晶体学数据处理和分析。 -
Python 作为脚本语言 :
phenix提供了大量的 Python 脚本和工具,用户可以使用这些脚本来完成诸如晶体结构解析、精修、建模等任务。例如,用户可以编写 Python 脚本调用phenix.refine模块来进行晶体结构的精修。 -
Python 模块和库的使用 :
phenix内部使用了许多 Python 标准库和第三方库,同时也开发了自己的 Python 模块,如libtbx等。这些模块和库为phenix提供了强大的功能支持,包括数据处理、算法实现、图形界面设计等。
2. phenix 中的 Python 与虚拟环境中 Python 的关系
-
可能相互独立 :
phenix通常会自带一个 Python 解释器和相关的 Python 环境,这个环境是为了确保phenix能够稳定运行而专门配置的。它可能包含了特定版本的 Python 以及phenix所需的所有依赖库。而虚拟环境中的 Python 是你为了开发其他项目而创建的独立 Python 环境,与phenix自带的 Python 环境相互独立。 -
可以集成使用 :在某些情况下,你可能希望将
phenix集成到你自己的虚拟环境中,以便在同一个环境中使用phenix和其他 Python 库。为了实现这一点,你需要将phenix的安装目录添加到虚拟环境的PYTHONPATH中,让虚拟环境的 Python 解释器能够找到phenix的模块和库。例如,在虚拟环境中执行以下命令:
Shell
export PYTHONPATH=/path/to/phenix/lib/python3.x/site-packages:$PYTHONPATH其中,/path/to/phenix 是 phenix 的实际安装目录,python3.x 是 phenix 所使用的 Python 版本。
- 版本兼容性问题 :需要注意的是,
phenix对 Python 版本有一定的要求,通常会指定特定的 Python 版本(如 Python 3.7)。因此,在集成phenix到虚拟环境时,要确保虚拟环境中的 Python 版本与phenix兼容,否则可能会出现模块导入错误或其他运行时问题。
综上所述,phenix 与 Python 紧密相关,它基于 Python 开发并使用 Python 作为脚本语言。phenix 自带的 Python 环境与虚拟环境中的 Python 可以相互独立,也可以通过配置进行集成使用,但需要注意版本兼容性问题。
二、phenix.map_model_cc
phenix.map_model_cc 是 Phenix 中专门用于计算 模型与电子密度图的整体及残基级别相关性(CC, Correlation Coefficient) 的工具。它比通用的 real_space_refine 或 validation_cryoem 更直接地提供 CC 计算功能 ,尤其适合快速评估模型与实验数据的匹配程度。
1. phenix.map_model_cc 的功能
-
整体 CC 计算:输出模型与密度图的全局相关系数(整体模型质量评估)。
-
残基级别 CC(
cc_per_residue):计算每个氨基酸残基的局部电子密度相关性,识别弱密度区域。 -
链级别 CC:可选计算每条链的平均 CC。
-
支持多种数据 :适用于 X 射线晶体学(
.mtz)和冷冻电镜(.mrc/.map)密度图。
2. 基本用法
命令行示例
Shell
phenix.map_model_cc model.pdb map.mrc输出结果:
-
终端打印全局 CC 值。
-
生成日志文件(
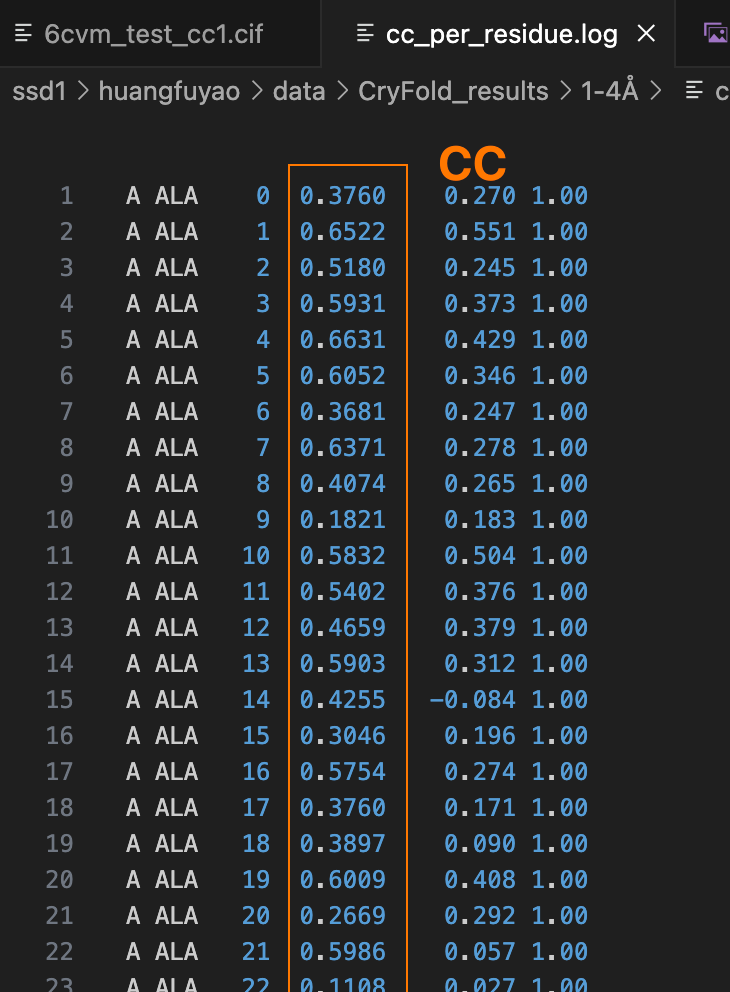
.log),包含残基级别的 CC 表格(如下示例):
Python
RESIDUE CHAIN RESID CC
ALA A 1 0.82
LEU A 2 0.76常用参数
| 参数 | 说明 |
|---|---|
output_per_residue=True |
强制输出残基级别 CC(默认已启用) |
resolution=3.0 |
指定分辨率(影响 CC 计算方式) |
output_file=cc_results.txt |
将结果保存到指定文件 |
🪧示例
powershell
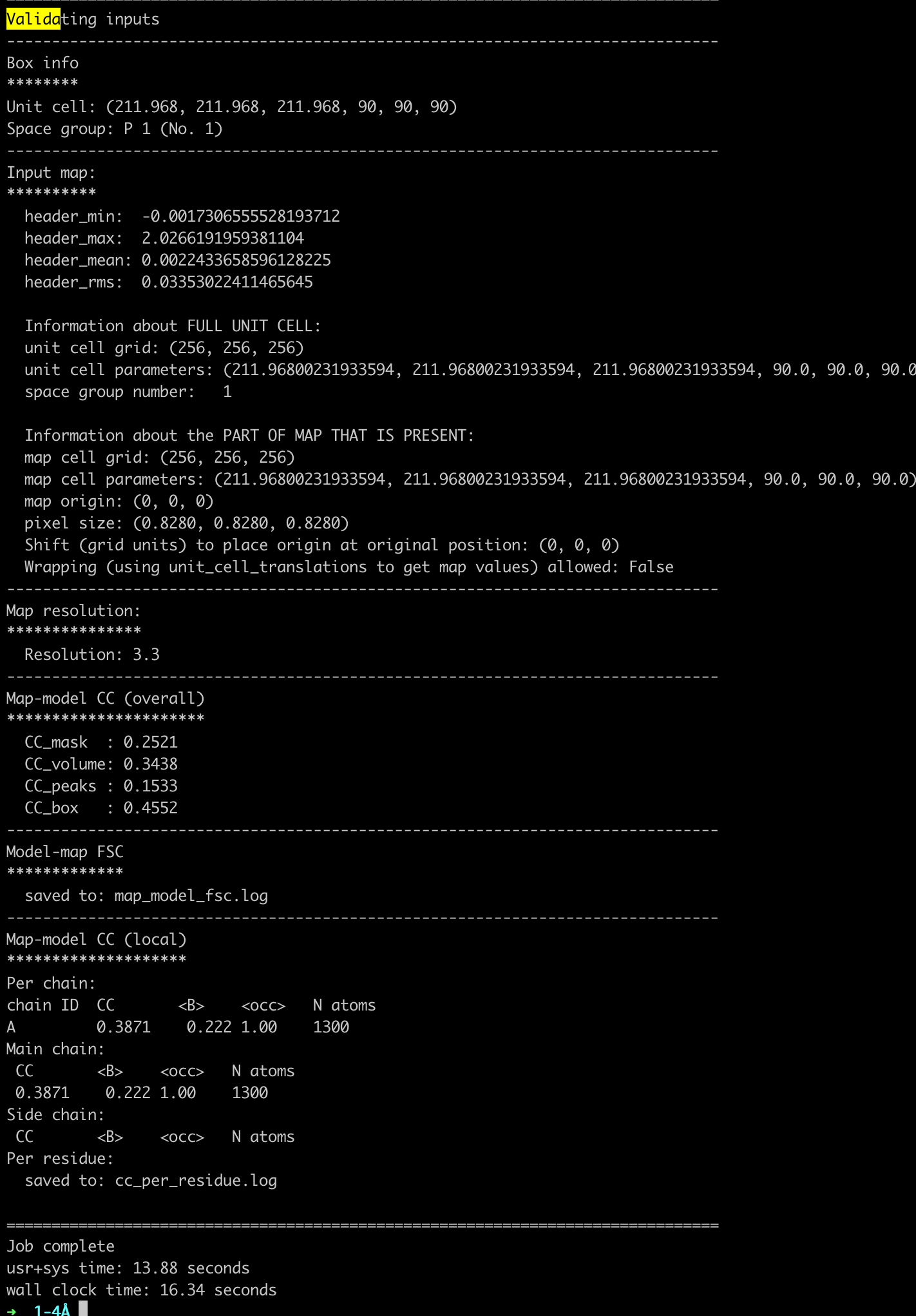
phenix.map_model_cc /home/huangfuyao/proj/CryFold/7uqx_win300_th0.8/see_alpha_output/ca_anchor_80_NMS_min0_0.1.cif \
/hdd1/msai/db/emdb/emd_26701.map \
resolution=3.3 \
per_residue=True \
output.filename=7uqx_ph_residue_cc.txt
Starting phenix.map_model_cc有一个bug是这个filename没什么用,最终会在同目录生成一个
cc_per_residue.log,里面记录了每个氨基酸的cc
3. 高级用法
powershell
> phenix.map_model_cc --show-defaults
map_model_cc {
resolution = None
scattering_table = wk1995 it1992 n_gaussian neutron *electron
atom_radius = None
compute {
cc_per_chain = True
cc_per_residue = True
cc_per_residue_group = False
fsc = True
cc_mask = True
cc_volume = True
cc_peaks = True
cc_box = True
cc_image = False
}
}
output {
file_name_prefix = None
filename = None
file_name = None
prefix = None
suffix = None
serial = 0
serial_format = "%03d"
overwrite = False
}(1) 计算局部区域 CC
若只需分析特定残基范围(如结合位点):
Shell
phenix.map_model_cc model.pdb map.mrc selection="chain A and resid 10:20"(2) 与 Python API 集成
通过脚本编程获取 CC 数据:
Python
from phenix.command_line import map_model_cc
result = map_model_cc.run(
args=["model.pdb", "map.mrc"],
output_per_residue=True
)
# 提取全局 CC
print("Global CC:", result.global_cc)
# 提取残基 CC
for residue in result.per_residue_cc:
print(f"{residue.chain_id} {residue.resseq} {residue.resname}: CC={residue.cc:.3f}")4. 结果解读
-
全局 CC:
-
> 0.8:模型与密度图匹配极好(高分辨率结构常见)。
-
0.6--0.8:中等质量,需检查局部问题。
-
< 0.5:可能存在严重错误(如模型偏差或低分辨率数据)。
-
-
残基 CC:
- 低值残基(如 CC < 0.3)可能对应:
5. 与其他工具对比
| 工具 | 适用场景 | 残基 CC 输出 | 额外功能 |
|---|---|---|---|
phenix.map_model_cc |
快速 CC 评估 | ✅ | 支持选区分析 |
phenix.real_space_refine |
结构优化中 CC 监控 | ✅ | 实时优化模型 |
phenix.validation_cryoem |
冷冻电镜专用验证 | ✅ | B因子、局部分辨率 |
6. 注意事项
-
分辨率影响:低分辨率下 CC 值普遍偏低,需谨慎解读。
-
原子掩模(masking) :计算 CC 时会自动排除溶剂区域,可通过
mask_params调整。 -
缺失原子处理:若模型缺失部分原子(如柔性区域),可能导致局部 CC 降低。
总结
phenix.map_model_cc 是 Phenix 中最直接、高效的模型-密度图相关性分析工具,特别适合:
-
快速验证新构建的模型质量。
-
定位需手动修正的低 CC 值残基。
-
与优化工具(如
real_space_refine)配合使用,指导迭代修正。