这篇论文提出了ThermoSeek,一个综合性的网络资源,用于分析来自嗜热和嗜冷物种的蛋白质序列和结构。具体来说,
- 数据收集:从美国国家生物技术信息中心(NCBI)的基因组数据库中收集了物种的分类ID,并根据"温度范围"标记为嗜热、超嗜热、嗜冷或冷适应。使用MMseqs2对蛋白质序列进行聚类和冗余消除,生成一个包含130,825个超嗜热蛋白、566,619个嗜热蛋白、486,139个嗜冷蛋白和19,793个冷适应蛋白的综合数据库。
- 序列比对:使用NCBI BLAST 2.13.0+和MMseqs2创建序列数据库,并通过"mmseqs easy-search"和"blastp"进行序列搜索。
- 结构搜索:利用Foldseek算法将蛋白质结构编码为20个离散值,表示二级结构特征和氨基酸之间的空间关系。使用MMseqs2进行结构搜索。
- 模体搜索:使用Fpocket v2.0识别超嗜热和嗜热蛋白质中的口袋,并将提取的蛋白质口袋编码为自定义的二进制格式。使用Kruskal算法构建最小生成树(MST),以优化搜索过程。
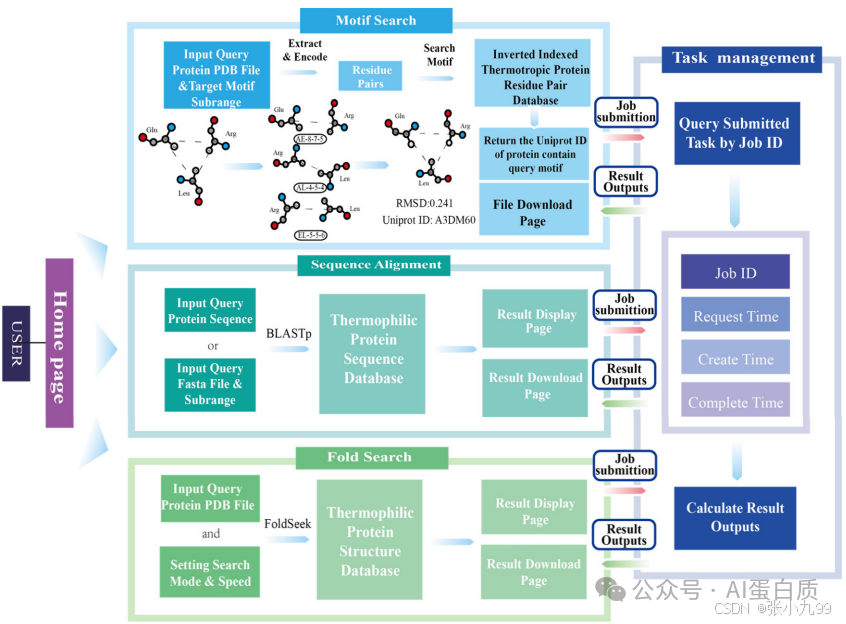
这篇论文提出了ThermoSeek,一个综合性的网络资源,用于分析来自嗜热和嗜冷物种的蛋白质序列和结构。具体来说,
- 数据收集:从美国国家生物技术信息中心(NCBI)的基因组数据库中收集了物种的分类ID,并根据"温度范围"标记为嗜热、超嗜热、嗜冷或冷适应。使用MMseqs2对蛋白质序列进行聚类和冗余消除,生成一个包含130,825个超嗜热蛋白、566,619个嗜热蛋白、486,139个嗜冷蛋白和19,793个冷适应蛋白的综合数据库。
- 序列比对:使用NCBI BLAST 2.13.0+和MMseqs2创建序列数据库,并通过"mmseqs easy-search"和"blastp"进行序列搜索。
- 结构搜索:利用Foldseek算法将蛋白质结构编码为20个离散值,表示二级结构特征和氨基酸之间的空间关系。使用MMseqs2进行结构搜索。
- 模体搜索:使用Fpocket v2.0识别超嗜热和嗜热蛋白质中的口袋,并将提取的蛋白质口袋编码为自定义的二进制格式。使用Kruskal算法构建最小生成树(MST),以优化搜索过程。