代码管理及Linux模拟工具Git for Windows安装使用教程
作者:曾美尹 中国农科院深圳基因组研究所
审核:刘永鑫 中国农科院深圳基因组研究所
更新时间:2025/11/4
Git for Windows提供了Git Bash等工具,可管理代码、同步GitHub项目并模拟Linux环境。核心功能含GitHub项目管理(克隆项目 git clone、同步 git pull、暂存 git add、提交 git commit、推送 git push)及协作(Fork & Pull requests)、模拟Linux终端外观,运行Linux命令(ls、grep),并且Git Bash还可在RStudio中配置使用。关于Git for Windows的下载安装以及使用教程如下:
视频教程
B站链接:https://www.bilibili.com/video/BV14Y1CBUEt7/
目录
🔷 Git for Windows下载安装
🔷 Git项目的创建、下载、同步、更新
🔸 配置Git本地账号
🔸 创建新项目及下载
🔸 同步和更新
🔸 参与他人项目
🔷 Git bash实现类Linux运行环境
Git for Windows下载安装
1.官网:打开官网 https://gitforwindows.org/,点击 Download 进行下载:
- 安装程序:下载完成后,点击.exe程序进行安装:
直接点击Next
默认安装目录为C:\Program Files\Git
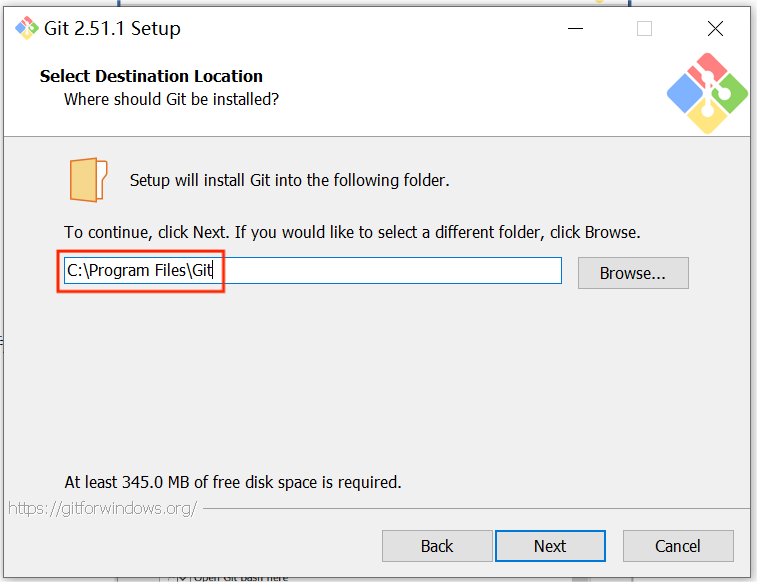
如果C盘空间不足,也可以改为其他安装目录
建议保持默认设定,点击Next直至Install
......
安装完成后便可使用Git了。
- 安装测试:安装完成后,我们可以在菜单栏找到Git系列,或者也可以在任何文件夹下单击鼠标右键快捷打开;
注意,在哪里启动GitBash,哪里就是当前目录,例如在菜单栏打开Git Bash,目录就是家目录(~);在D盘打开,目录就是/d
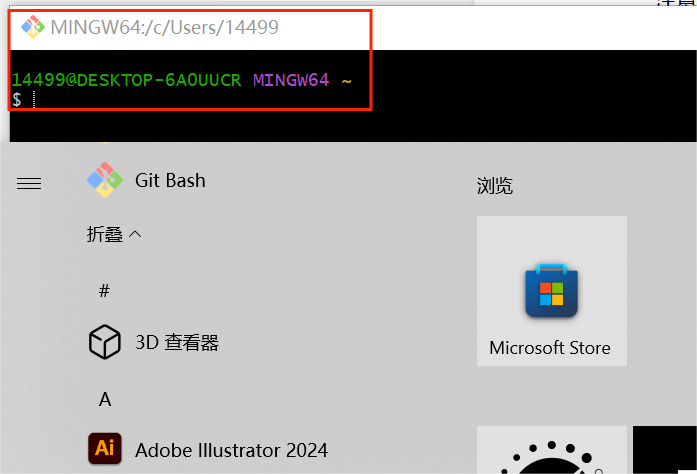
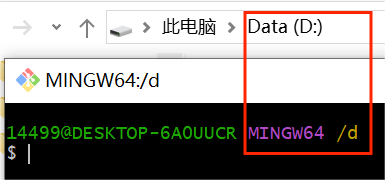
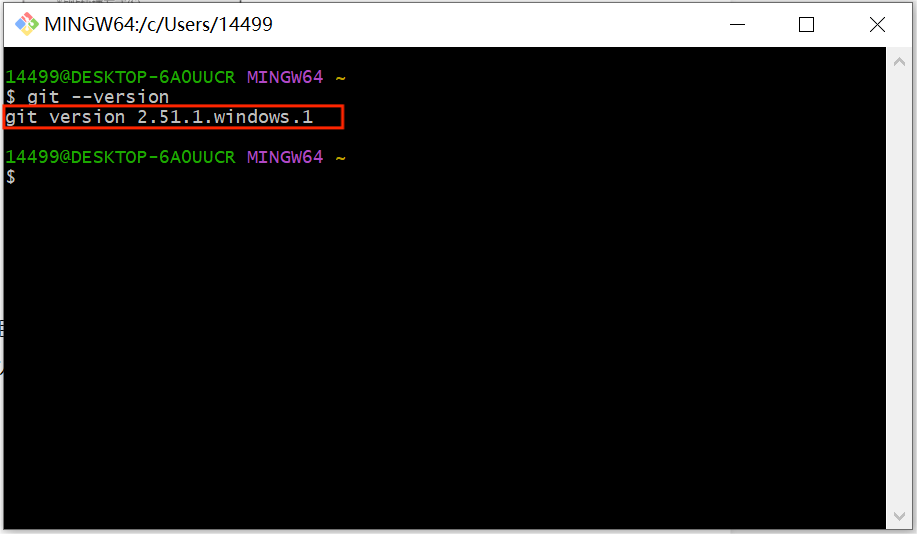
我们打开Git Bash,输入"git --version",如果显示Git版本信息则代表安装成功;
Git项目的创建、下载、同步、更新
安装完成后我们可以看到,Git主要由三个工具组成,分别是Git Bash 、Git CMD 、Git GUI,三者共用同一个Git引擎,只是交互方式不同。
Git Bash 是一个类Linux命令行环境,它可以运行所有Git命令(如git add)同步GitHub项目,也能运行大量Linux命令(如ls, cat, grep, cd),模拟Linux目录结构、路径写法(/home/user/project);Git CMD 是在Windows的CMD命令提示符中加入了Git的执行路径。它可运行所有Git命令,使用Windows命令语法,但不支持Linux命令;Git GUI是一个图形化工具,可以可视化地执行常见操作。
Git Bash作为其中最常用的工具,这里我们将对其进行介绍:
一. 配置Git本地账号
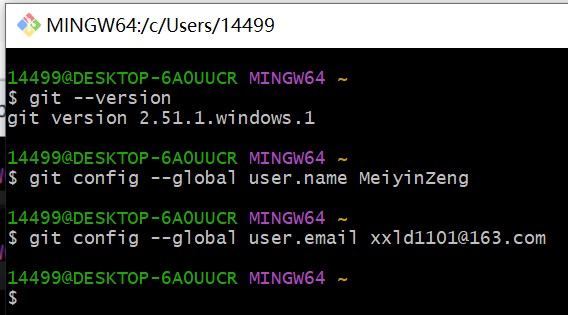
使用GitBash进行代码管理时Git需要提交使用者的用户名和邮箱,所以需要先配置好个人信息。(这里的个人信息指的是GitHub账号信息,还没有GitHub账号的用户需要先在GitHub官网https://github.com/注册个人账号)。打开GitBash,输入以下命令:
git config --global user.name "GitHub用户名"
git config --global user.email "邮箱"
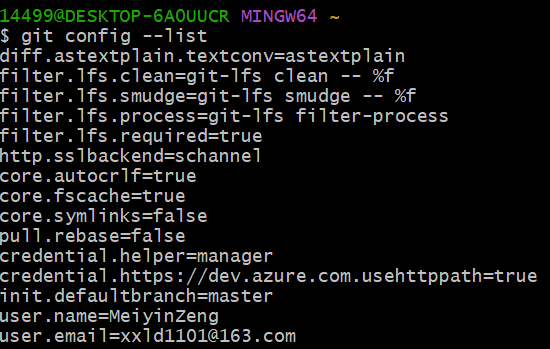
配置完成后可以使用 git config --list查看配置信息
二. 创建新项目及下载
- GitHub新建项目资源库:首先需要在GitHub官网(https://github.com )登陆个人账号,登陆后点击右上角+号,选择New repository;
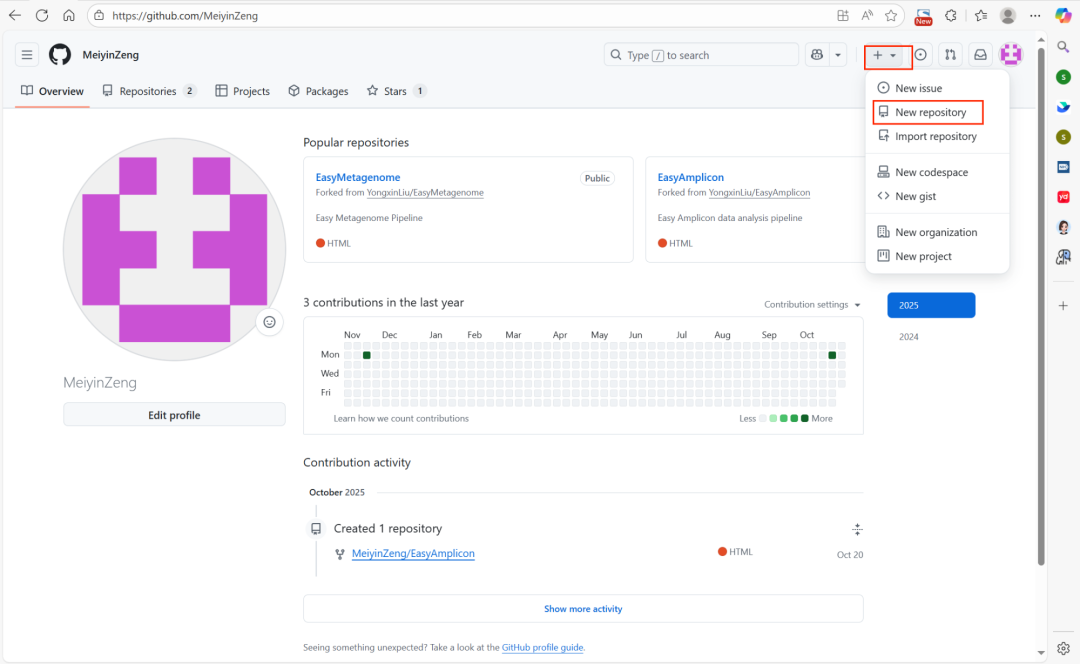
在随后的界面中可以填写资源库名、创建 README/.gitignore 等,这里我们建了一个名为test的项目,并添加了一个README文件
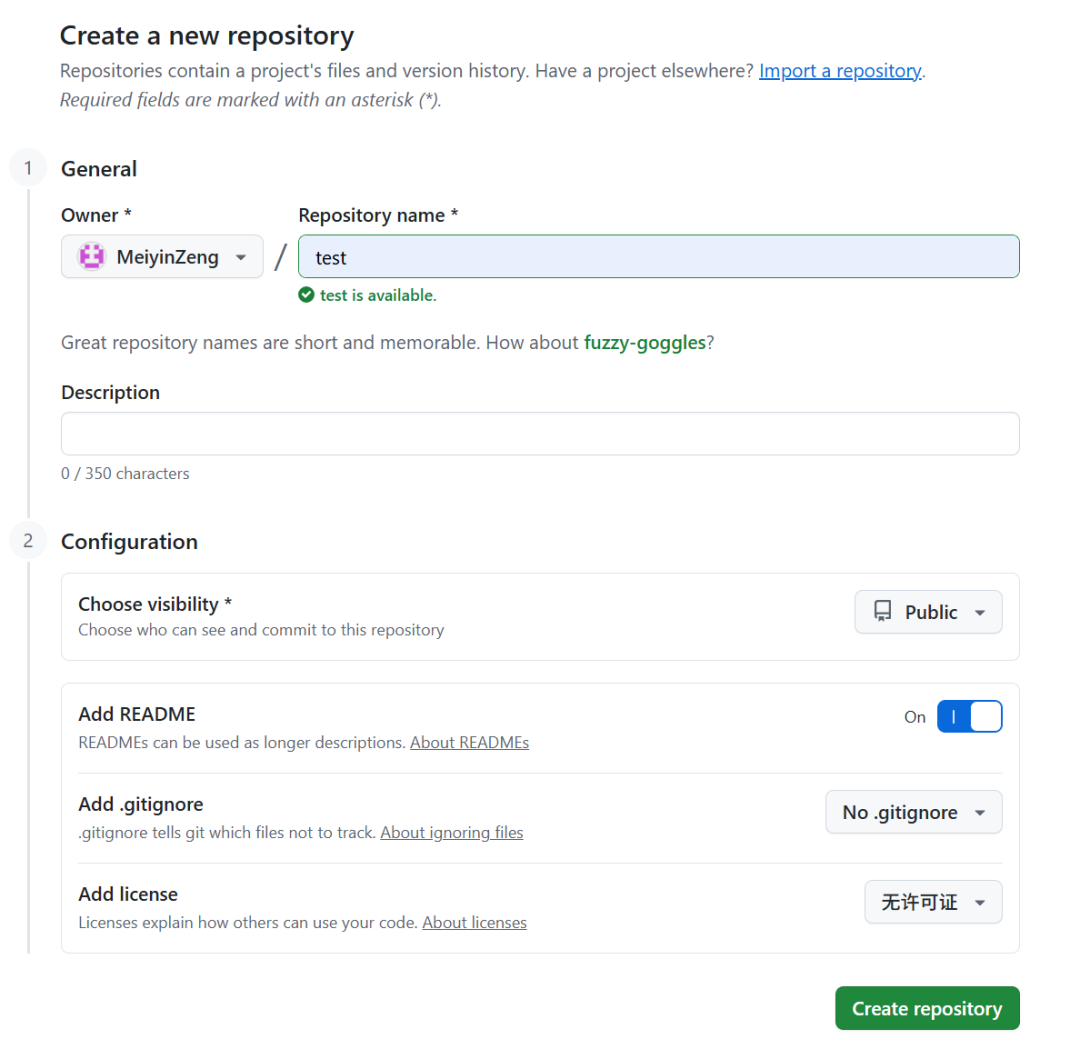
创建成功后会自动进入这个项目的主页面,这里可以看到该项目目前只有一个我们创建的README文件,如果想要添加其他文件,可以点击右上角 Add files 选项,选择新建文件在线编辑或者上传本地文件;或者,也可以通过Git Bash同步本地与远端资源库(详见下文)
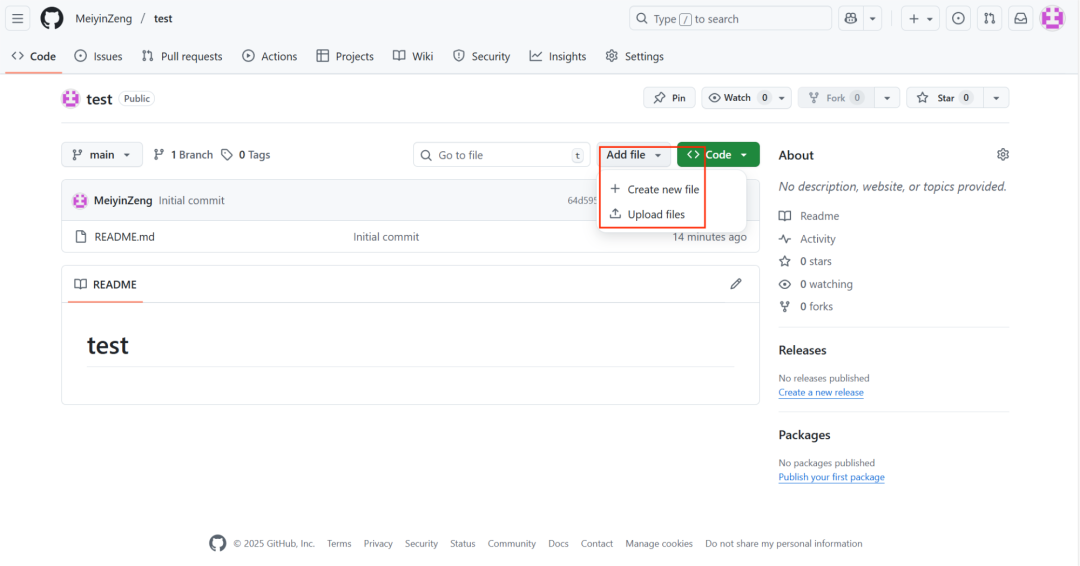
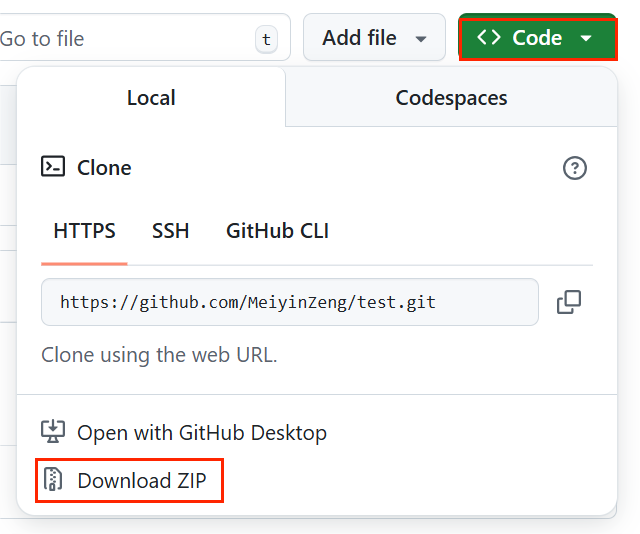
- GitHub项目下载:如果想要从GitHub下载项目到本地,可以点击项目主页的 Code 选项,选择 Download ZIP 下载整个项目;
如果想在Git Bash上下载并关联GitHub项目,可以通过 git clone命令与该项目的HTTPS URL实现。(传输速度受网络稳定性影响)
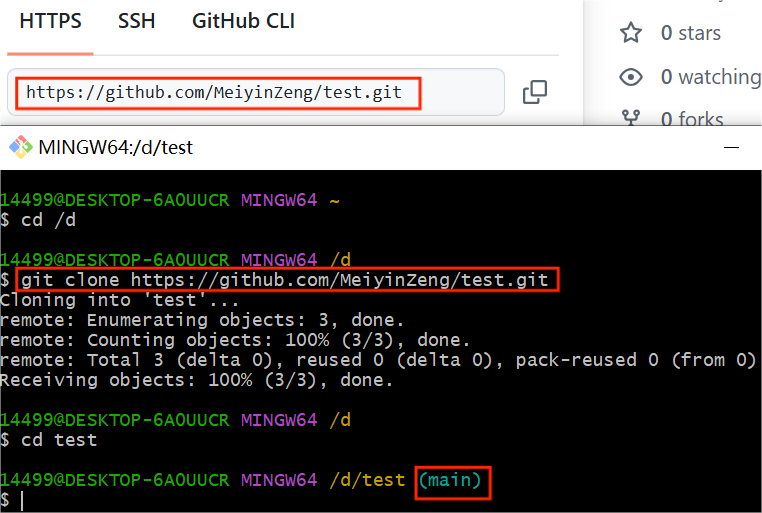
跳转到项目文件夹后会接一个*(main)* 字样,这是我们与GitHub连接的分支,以前一般为master,现在默认为main,也可以运行 git branch -M master将main修改为master。随后便可以在Git Bash上进行项目管理,详见下文。
三. 同步和更新
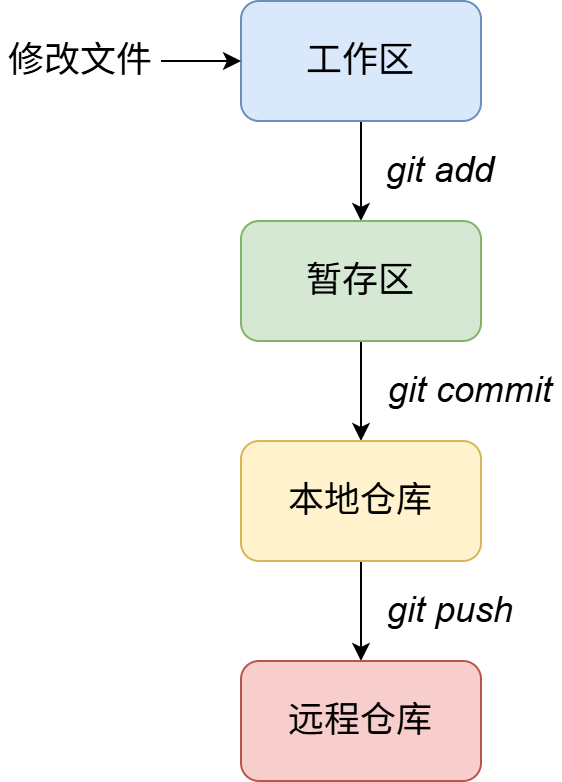
- Git分区解释 :在利用Git Bash进行项目管理之前,必须要先理解Git的各个分区。Git有四个区域------工作区Workspace 、暂存区Index/Stage 、本地仓库Local Repository 、远程仓库Remote ,工作区就是你的项目文件夹,平时修改代码、创建文件、删除文件,都发生在工作区;暂存区用于存放准备提交的文件,你可能修改了许多文件,但你只想提交其中一部分,那就可以用 git add 选择性放到暂存区,随后再 git commit 提交到本地仓库。提交到本地仓库的文件会形成永久记录,从而可以进行历史比较与版本回退;远程仓库就是GitHub上的仓库,当我们在本地仓库完成更新后,可以选择把其同步到云端,这个时候我们就可以利用 git push把本地仓库的修改历史同步到GitHub上,从而既能向他人展示代码,又能进行备份与多人协作。
- 项目同步及更新操作 :当我们在Git Bash上下载并关联了GitHub项目以后,就可以对其进行修改,有时一个项目有多人协作修改,我们就需要在修改之前先进行项目同步,确保当前是最新版后再修改并提交自己的更新内容;以上文新建的test项目为例,假设当前有其他人在协作这个项目,那么我们在进行修改前就需要运行 git pull把远程仓库里别人更新的代码拉下来,并与本地合并,确保当前为最新版后便可以对项目进行修改更新了;
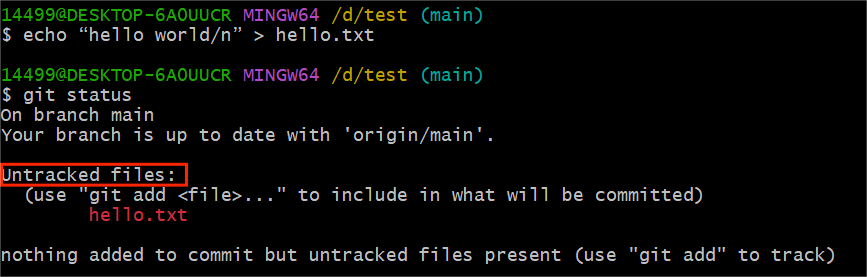
# 假设我们要新增一个文件hello.txt
git pull # 确保在多人协作情况下拿到最新版本
echo "hello world/n" > hello.txt # 新建一个内容为hello world的hello.txt文件,也可以直接在文件夹内手动新建
git status # 查看文件状态,显示Untracked就代表文件还在工作区,未被传递到暂存区
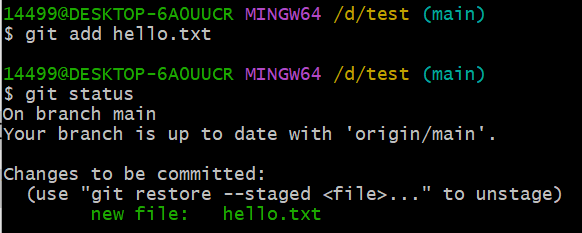
git add hello.txt # 放入"暂存区"
# git add . # 也可以直接将工作区全部改动都加入暂存区
git status # 这时候可以再查看一下文件状态,已经改变了
# 接下来就可以把暂存区的文件提交到本地仓库了
git commit -m "Add hello.txt"# -m添加提交说明
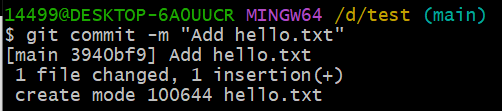
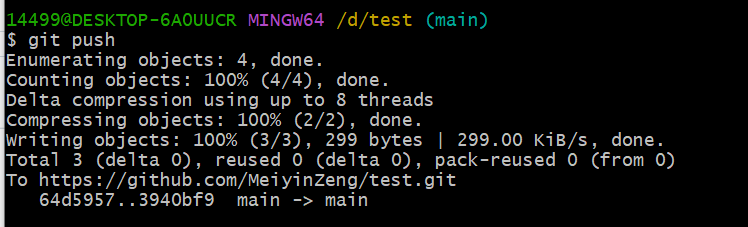
git push# 推送至GitHub
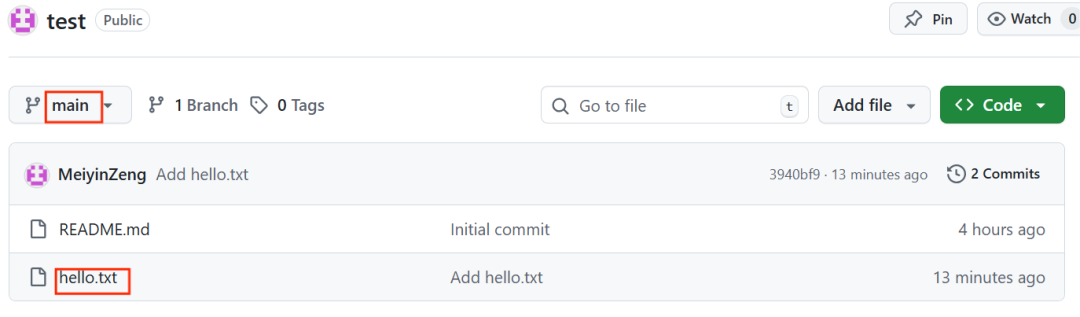
这时候我们再进入GitHub查看test项目,可以看到main分支下新增了一个hello.txt文件,代表我们更新成功了
四. 参与他人项目
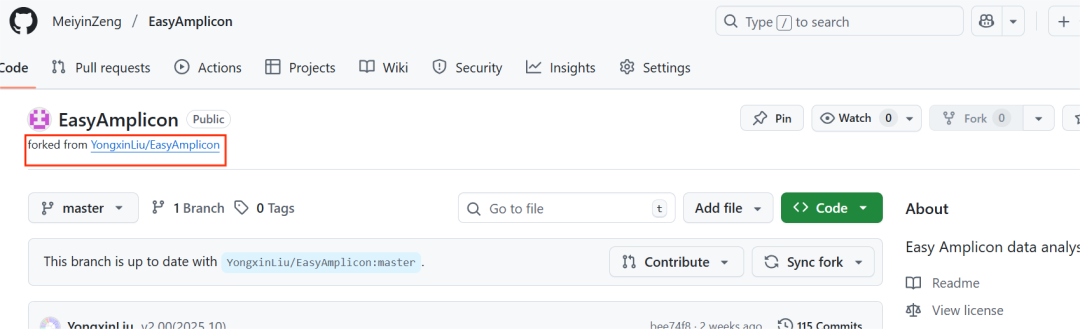
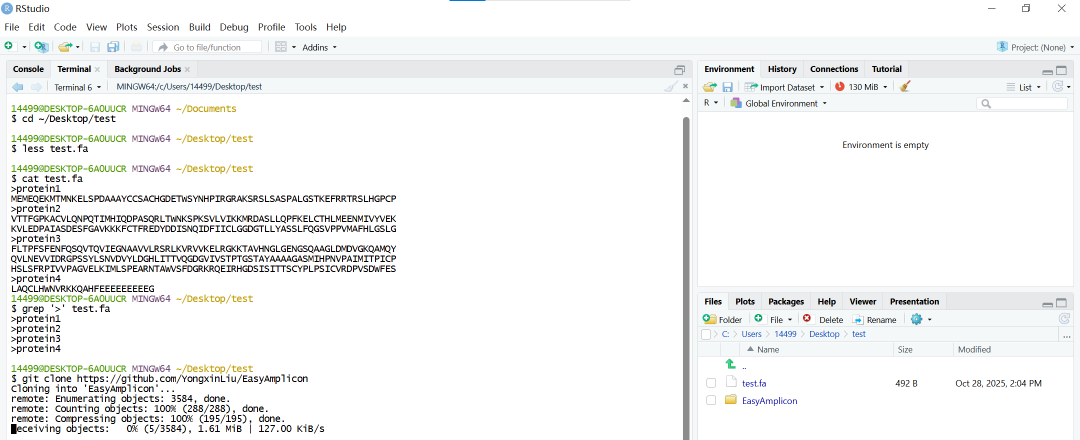
- Fork备份项目:我们可以在GitHub上备份他人项目以便本地使用或者参与合作更新,以EasyAmplicon包为例,我们可以在作者主页https://github.com/YongxinLiu/EasyAmplicon点击 Fork 将该项目备份至自己账号下;
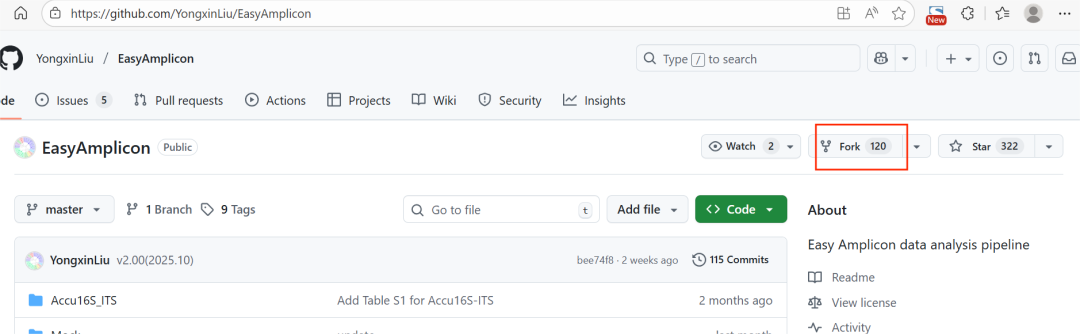
再回到我们主页就可以看到该项目了
-
下载、同步、更新:操作与个人项目相同,只不过把test更换为EasyAmplicon罢了;
-
请求同步Pull requests:如果我们对他人的项目进行了更新,并且想向原作者同步我们所做的修改,可以在更新后的fork项目主页点击Pull requests;
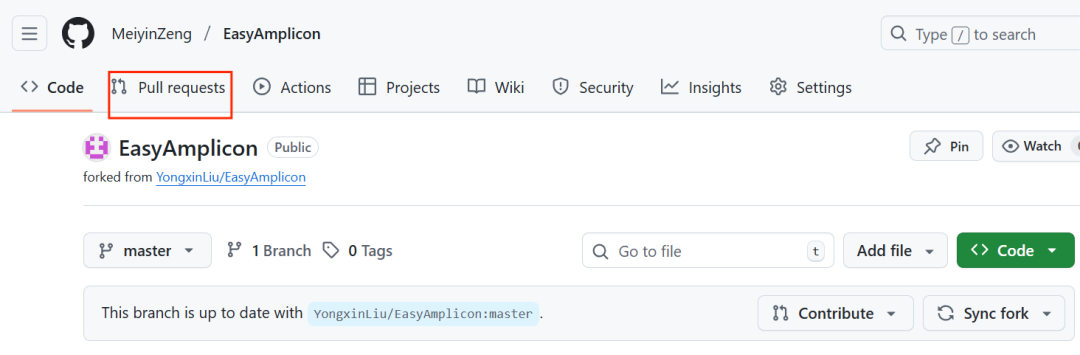
随后点击New pull request
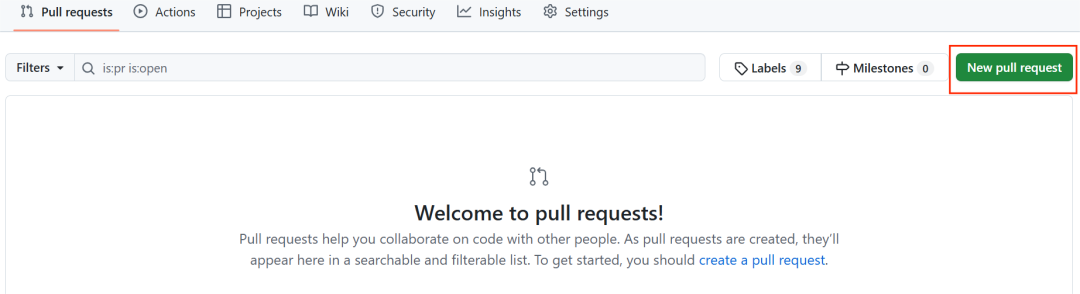
base repository 选择原作者的仓库,base 选它的主分支(main/master),head repository 选你的 fork,compare 选你刚推送的分支(main/master),写清标题与说明(做了什么、为什么、如何测试、可能影响、相关Issue/讨论链接等)后提交即可。(此处图片只做参考,因为我并没有对Fork项目做任何修改)
Git bash实现类Linux运行环境
Git for windows提供了大量linux命令的windows版本,可帮助用户在Windows系统Git bash命令行中实现绝大多数Linux命令和Shell脚本的运行。
- 查看可用的linux命令:本质上Git将Linux命令重新编写了适合windows使用的exe可执行文件版本,可运行以下代码查看一下系统中有哪些可用的linux命令;
# 进入系统命令目录
cd /bin
# 查看所有exe可执行程序
ls *.exe
列出了240余个Shell命令程序,不在目录的shell命令也可上网搜索相应的windows版本exe文件,人工添加至windows环境变量
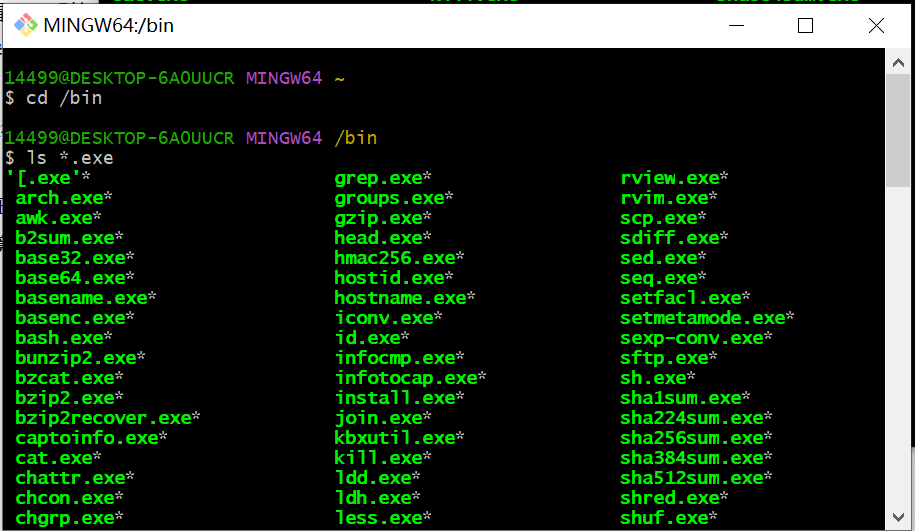
- fasta文件操作示例:我们在桌面上进建一个test文件夹,进入后右键执行git bash,这时我们的起始目录即为当前test目录~/Desktop/test;
首先我们创建一个fasta序列文件,在终端中输入cat >test.txt创建名为test的txt文件,回车后即开始编写文件状态,随后复制以下内容写入文件------
>protein1
MEMEQEKMTMNKELSPDAAAYCCSACHGDETWSYNHPIRGRAKSRSLSASPALGSTKEFRRTRSLHGPCP
>protein2
VTTFGPKACVLQNPQTIMHIQDPASQRLTWNKSPKSVLVIKKMRDASLLQPFKELCTHLMEENMIVYVEK
KVLEDPAIASDESFGAVKKKFCTFREDYDDISNQIDFIICLGGDGTLLYASSLFQGSVPPVMAFHLGSLG
>protein3
FLTPFSFENFQSQVTQVIEGNAAVVLRSRLKVRVVKELRGKKTAVHNGLGENGSQAAGLDMDVGKQAMQY
QVLNEVVIDRGPSSYLSNVDVYLDGHLITTVQGDGVIVSTPTGSTAYAAAAGASMIHPNVPAIMITPICP
HSLSFRPIVVPAGVELKIMLSPEARNTAWVSFDGRKRQEIRHGDSISITTSCYPLPSICVRDPVSDWFES
>protein4
LAQCLHWNVRKKQAHFEEEEEEEEEG
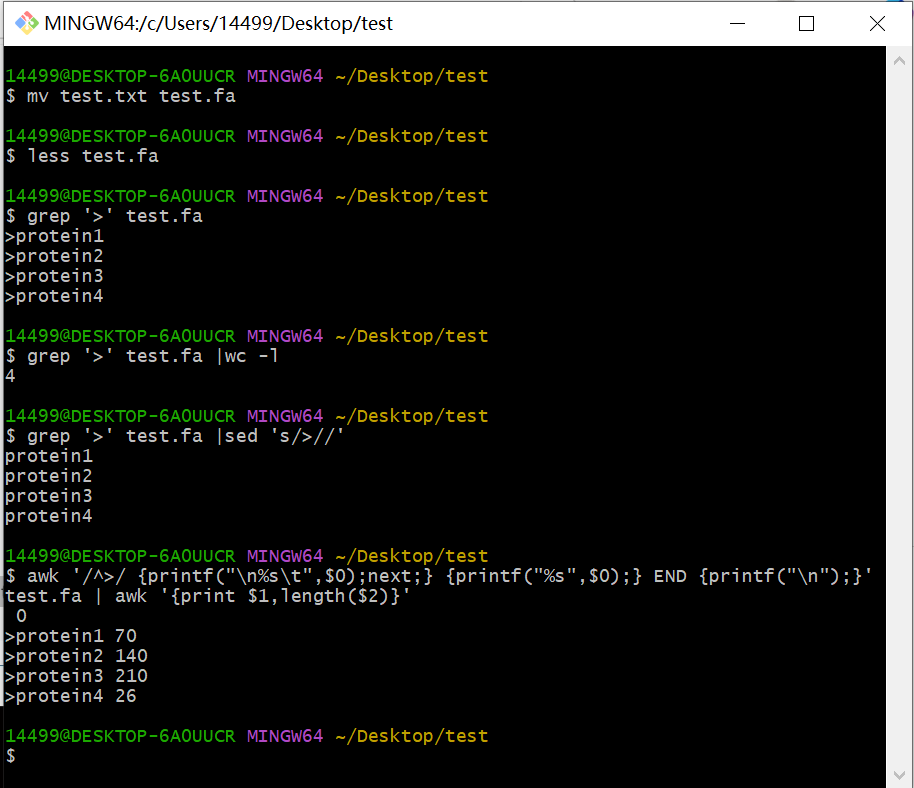
在Git Bash中,我们只需按鼠标滚轮即可快速粘贴剪切板中内容,再按Ctrl+D组合实现结束编辑并保存。成功保存文件后,我们可以对这个序列文件进行简单的操作
# 使用cat命令显示文件内容
cat test.txt
# fasta文件通常为.fa的扩展名,mv命令改名
mv test.txt test.fa
# 文件很大可使用less按页查看,空格翻页,q退出查看模式
less test.fa
# 只查看序列名称行
grep '>' test.fa
# 统计序列条数:即上条命令接wc统计字数,-l只统计行数
grep '>' test.fa |wc -l
# sed去除序列名中的大于号
grep '>' test.fa | sed 's/>//'
# 显示每条序列长度
# 基本思路将fasta文件多行并单行两列,为序列名和序列
# 统计第二列长度并输出
awk '/^>/ {printf("\n%s\t",0);next;} {printf("%s",0);} END {printf("\n");}' test.fa | awk '{print 1,length(2)}'
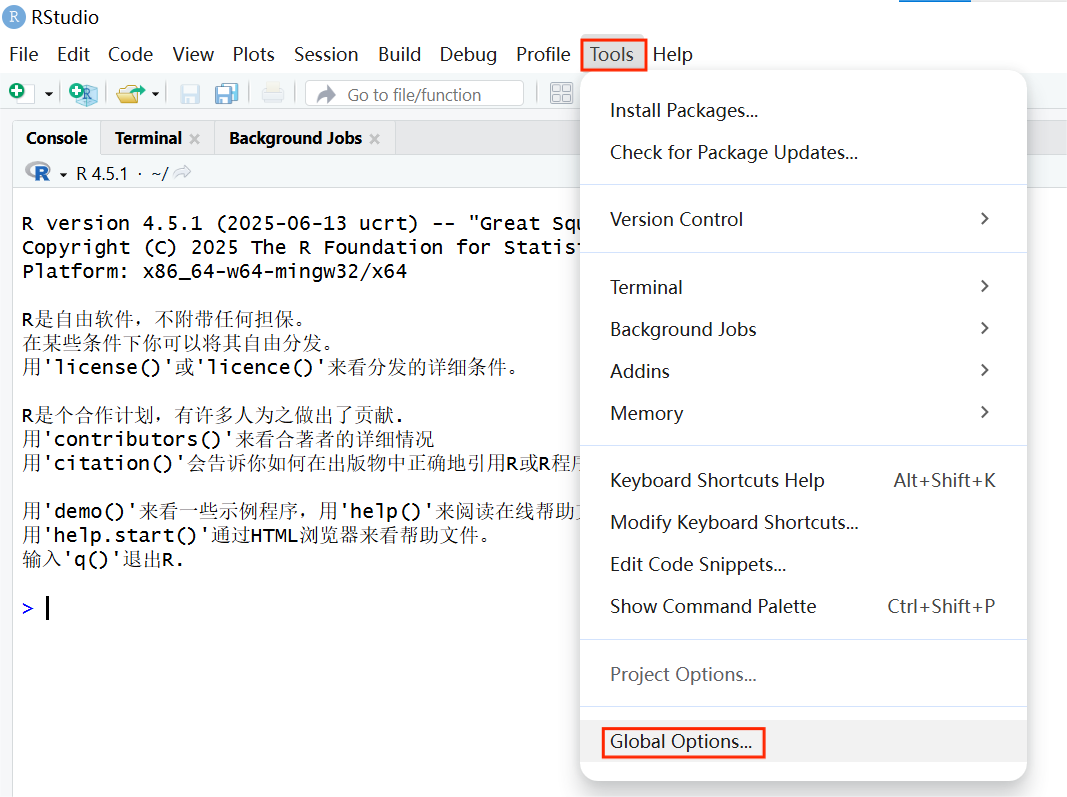
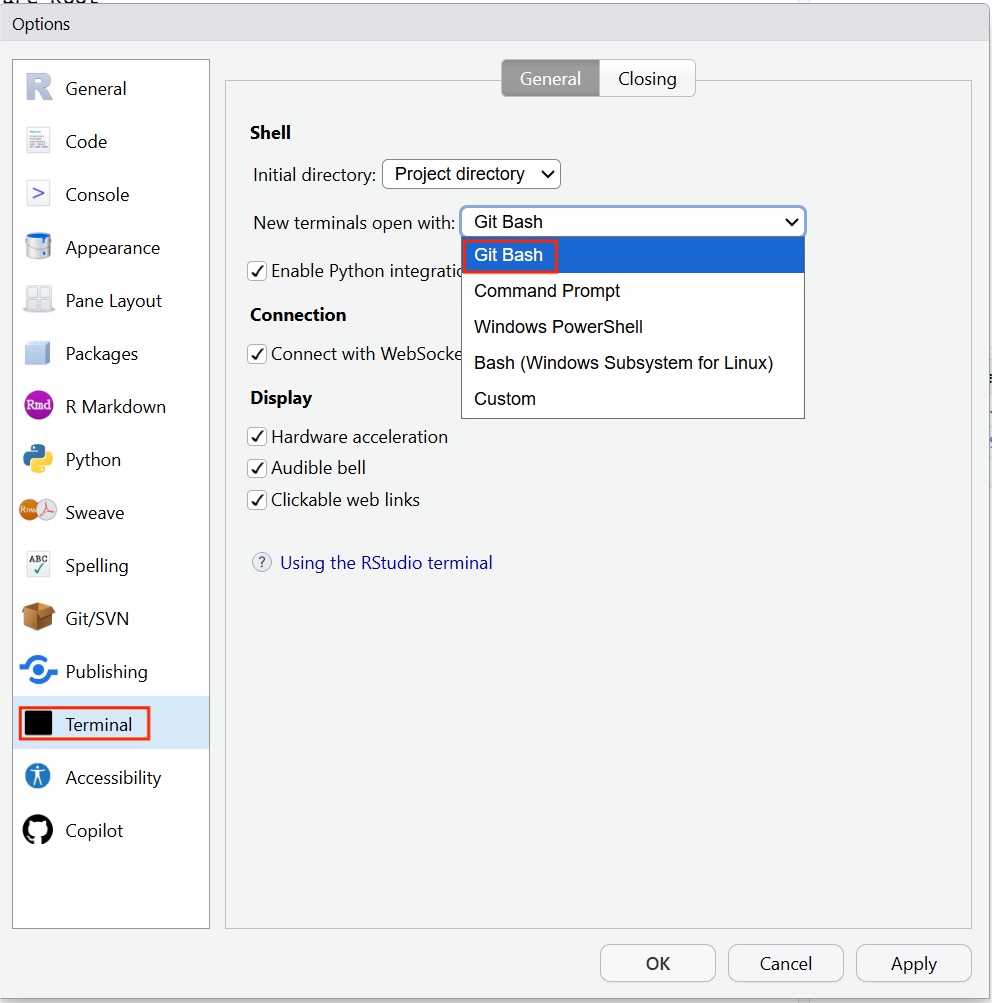
- 在RStudio中操作Git Bash:如果你的电脑中刚好安装了RStudio,那么可以直接在RStudio中运行Git Bash。只需要将Tools ------ Global Options ------
Terminal处的 New terminals open with 选项切换为Git Bash,随后在操作台打开一个New terminal就好了;
切换New Terminal后可以直接在该terminal下输入Linux及Git命令,与直接使用Git Bash没有区别,只是RStudio提供了更加可视化的界面,可在Files栏直接观察文件状态
宏基因组推荐
本公众号现全面开放投稿,希望文章作者讲出自己的科研故事,分享论文的精华与亮点。投稿请联系小编(微信号:yongxinliu 或 meta-genomics)
iMeta高引 fastp PhyloSuite ImageGP2iNAP2 ggClusterNet2
iMeta工具 SangerBox2 美吉2024 OmicStudioWekemo OmicShare
iMeta综述 高脂饮食菌群 发酵中药 口腔菌群 微塑料 癌症 宿主代谢
10000+:扩增子EasyAmplicon 比较基因组JCVI 序列分析SeqKit2 维恩图EVenn
iMetaOmics高引 猪微生物组 16S扩增子综述 易扩增子(EasyAmplicon)
点击阅读原文