文章目录
- 一、最近邻分类相关理论
-
- [1. 核心原理](#1. 核心原理)
- [2. 核心要素](#2. 核心要素)
-
- [2.1 超参数 K K K的选择](#2.1 超参数 K K K的选择)
- [2.2 距离度量方式](#2.2 距离度量方式)
- [2.3 特征预处理](#2.3 特征预处理)
- [3. 算法特点(优缺点)](#3. 算法特点(优缺点))
-
- [3.1 优点](#3.1 优点)
- [3.2 缺点](#3.2 缺点)
- [4. 适用场景与不适用场景](#4. 适用场景与不适用场景)
-
- [4.1 适用场景](#4.1 适用场景)
- [1.4.2 不适用场景](#1.4.2 不适用场景)
- 二、KNN相关方法介绍
-
- [1. KNN构造方法](#1. KNN构造方法)
- [2. KNN返回对象的方法介绍](#2. KNN返回对象的方法介绍)
-
- [2.1 .fit(X, y)](#2.1 .fit(X, y))
- [2.2 .predict(X)](#2.2 .predict(X))
- [2.3 .predict_proba(X)](#2.3 .predict_proba(X))
- [2.4 .kneighbors(X)](#2.4 .kneighbors(X))
- [2.5 .kneighbors_graph(X)](#2.5 .kneighbors_graph(X))
- [3. KNN返回对象的属性介绍](#3. KNN返回对象的属性介绍)
-
- [3.1 .classes_](#3.1 .classes_)
- [3.2 .n_neighbors_](#3.2 .n_neighbors_)
- [3.3 ._fit_X](#3.3 ._fit_X)
- [3.4 ._y](#3.4 ._y)
- [3.5 .effective_metric_](#3.5 .effective_metric_)
- [4. KNN模型评估指标方法介绍](#4. KNN模型评估指标方法介绍)
-
- [4.1 准确率(Accuracy)](#4.1 准确率(Accuracy))
- [4.2 混淆矩阵(Confusion Matrix)](#4.2 混淆矩阵(Confusion Matrix))
- [4.3 分类报告(Classification Report)](#4.3 分类报告(Classification Report))
- [4.4 精确率、召回率与F1-score](#4.4 精确率、召回率与F1-score)
- [4.5 ROC曲线与AUC](#4.5 ROC曲线与AUC)
- [4.6 评估指标选择建议](#4.6 评估指标选择建议)
- [5. K值确定相关方法](#5. K值确定相关方法)
-
- [5.1 cross_val_score()方法](#5.1 cross_val_score()方法)
- [5.2 GridSearchCV类](#5.2 GridSearchCV类)
- 三、KNN实现步骤(分类)
-
- [1. 数据收集与加载](#1. 数据收集与加载)
-
- [1.1 威斯康星乳腺癌数据集介绍](#1.1 威斯康星乳腺癌数据集介绍)
-
- [1.1.1 数据集基本信息](#1.1.1 数据集基本信息)
- [1.1.2 数据集字段说明](#1.1.2 数据集字段说明)
- [1.2 数据加载](#1.2 数据加载)
- [2. 数据探索与可视化(EDA)](#2. 数据探索与可视化(EDA))
-
- [2.1 数据基本信息](#2.1 数据基本信息)
-
- [2.1.1 查看数据前10行](#2.1.1 查看数据前10行)
- [2.1.2 查看数据摘要信息](#2.1.2 查看数据摘要信息)
- [2.1.3 特征描述性统计](#2.1.3 特征描述性统计)
- [2.2 目标变量类别分布分析](#2.2 目标变量类别分布分析)
- [2.3 目标变量类别与特征相关性分析](#2.3 目标变量类别与特征相关性分析)
- [2.4 关键特征分布分析](#2.4 关键特征分布分析)
- [2.5 关键特征相关性分析](#2.5 关键特征相关性分析)
- [2.6 关键特征的类别区分能力验证](#2.6 关键特征的类别区分能力验证)
- [3. 数据预处理](#3. 数据预处理)
-
- [3.1 特征筛选](#3.1 特征筛选)
- [3.2 数据集划分](#3.2 数据集划分)
- [3.3 特征标准化](#3.3 特征标准化)
- [4. KNN模型构建与训练](#4. KNN模型构建与训练)
-
- [4.1 KNN模型构建及确定最优K值](#4.1 KNN模型构建及确定最优K值)
-
- [4.1.1 肘部法确定最优K值](#4.1.1 肘部法确定最优K值)
- [4.1.2 网格搜索与交叉验证确定最优K值](#4.1.2 网格搜索与交叉验证确定最优K值)
- [4.1.3 网格搜索与交叉验证结果可视化](#4.1.3 网格搜索与交叉验证结果可视化)
- [4.2 KNN模型训练](#4.2 KNN模型训练)
- [5. KNN模型预测与评估](#5. KNN模型预测与评估)
-
- [5.1 模型预测](#5.1 模型预测)
- [5.2 核心评估指标计算](#5.2 核心评估指标计算)
- [5.3 可视化评估](#5.3 可视化评估)
-
- [5.3.1 混淆矩阵可视化](#5.3.1 混淆矩阵可视化)
- [5.3.2 ROC曲线与AUC值可视化](#5.3.2 ROC曲线与AUC值可视化)
- [5.3.3 预测概率分布可视化](#5.3.3 预测概率分布可视化)
- 四、完整代码
-
- [1. 完整代码](#1. 完整代码)
一、最近邻分类相关理论
最近邻分类(K-Nearest Neighbors, KNN)是机器学习中最经典的惰性学习(Lazy Learning) 算法,核心思想是"物以类聚、人以群分"------通过查找待预测样本的"邻居"(相似样本),根据邻居的类别来判定待预测样本的类别。它无需预先训练模型参数,仅在预测时利用训练数据,因此也被称为"实例-based学习"。
1. 核心原理
KNN的原理可概括为"3步走",直观且易于理解:
- 计算距离 :给定待预测样本 x x x,计算 x x x与训练集中所有样本的"相似度"(用距离度量表示,距离越近相似度越高);
- 筛选邻居 :从训练集中选取与 x x x距离最近的 K K K个样本,这 K K K个样本即为 x x x的" K K K个最近邻"( K K K是人为设定的超参数);
- 投票决策 :对 K K K个最近邻的类别进行"多数投票",得票最多的类别即为待预测样本 x x x的预测类别(若 K = 1 K=1 K=1,则直接取距离最近样本的类别)。
举个例子:要判断"某部电影是否为喜剧片"(正类=喜剧,负类=非喜剧), K = 5 K=5 K=5时,找到与该电影最相似的5部电影,若其中3部是喜剧片、2部是非喜剧片,则通过投票判定该电影为喜剧片。
2. 核心要素
KNN的性能完全依赖于3个关键要素,选择不当会直接导致模型效果下降:
2.1 超参数 K K K的选择
K K K是KNN中最核心的超参数,决定了"邻居"的数量,其取值对结果影响显著:
- K K K过小(如 K = 1 K=1 K=1):模型对噪声样本高度敏感,易过拟合。例如,若最近邻是一个异常标注的样本,会直接导致预测错误;
- K K K过大:会包含大量与待预测样本不相似的"远邻",稀释真实邻居的影响,导致模型欠拟合,分类边界模糊;
- 经验取值 : K K K通常取奇数(避免投票平局),且需通过交叉验证(如5折交叉验证)选择最优值,常见范围为 [ 3 , 15 ] [3, 15] [3,15]。
2.2 距离度量方式
距离度量用于量化样本间的"相似度",KNN对距离度量的选择敏感,需根据特征类型(连续型、离散型)选择合适的方式:
- 欧氏距离(Euclidean Distance) :最常用 的连续型特征距离度量,计算两点在特征空间中的直线距离。公式为:
d ( x , y ) = ∑ i = 1 n ( x i − y i ) 2 d(x,y) = \sqrt{\sum_{i=1}^n (x_i - y_i)^2} d(x,y)=i=1∑n(xi−yi)2
其中 x i x_i xi、 y i y_i yi分别为样本 x x x、 y y y的第 i i i个特征值, n n n为特征数。适用于特征尺度一致的场景(如身高、体重均为数值型且量级相近); - 曼哈顿距离(Manhattan Distance) :计算两点在特征空间中的"街区距离",公式为:
d ( x , y ) = ∑ i = 1 n ∣ x i − y i ∣ d(x,y) = \sum_{i=1}^n |x_i - y_i| d(x,y)=i=1∑n∣xi−yi∣
适用于特征存在异常值的场景(曼哈顿距离对异常值的敏感度低于欧氏距离); - 余弦相似度(Cosine Similarity) :衡量两样本特征向量的夹角余弦值,取值范围 [ − 1 , 1 ] [-1,1] [−1,1],值越接近1说明相似度越高。公式为:
c o s ( x , y ) = x ⋅ y ∣ ∣ x ∣ ∣ ⋅ ∣ ∣ y ∣ ∣ = ∑ i = 1 n x i y i ∑ i = 1 n x i 2 ⋅ ∑ i = 1 n y i 2 cos(x,y) = \frac{x \cdot y}{||x|| \cdot ||y||} = \frac{\sum_{i=1}^n x_i y_i}{\sqrt{\sum_{i=1}^n x_i^2} \cdot \sqrt{\sum_{i=1}^n y_i^2}} cos(x,y)=∣∣x∣∣⋅∣∣y∣∣x⋅y=∑i=1nxi2 ⋅∑i=1nyi2 ∑i=1nxiyi
适用于高维稀疏特征(如文本分类中的词频特征),核心关注"方向一致性"而非"数值大小"; - 汉明距离(Hamming Distance) :适用于离散型特征,计算两样本对应特征值不同的个数,公式为:
d ( x , y ) = ∑ i = 1 n I ( x i ≠ y i ) d(x,y) = \sum_{i=1}^n I(x_i \neq y_i) d(x,y)=i=1∑nI(xi=yi)
其中 I ( ⋅ ) I(\cdot) I(⋅)为指示函数, x i ≠ y i x_i \neq y_i xi=yi时取1,否则取0(如用户标签、类别型特征的相似度计算)。
2.3 特征预处理
K N N KNN KNN的距离计算依赖特征的数值尺度,若特征尺度差异过大(如"年龄"范围 [ 18 , 60 ] [18,60] [18,60],"收入"范围 [ 1000 , 100000 ] [1000, 100000] [1000,100000]),会导致距离被尺度大的特征主导(如收入对距离的影响远大于年龄),模型无法学习到真实的相似性。
因此,特征预处理是KNN的必要步骤,常用方法:
- 标准化(Z-Score):将特征转化为均值=0、标准差=1的分布,公式为 x s t d = x − x ˉ σ x_{std} = \frac{x - \bar{x}}{\sigma} xstd=σx−xˉ,适用于特征近似正态分布的场景;
- 归一化(Min-Max):将特征压缩到 [ 0 , 1 ] [0,1] [0,1]区间,公式为 x n o r m = x − x m i n x m a x − x m i n x_{norm} = \frac{x - x_{min}}{x_{max} - x_{min}} xnorm=xmax−xminx−xmin,适用于特征分布无明显规律的场景。
3. 算法特点(优缺点)
3.1 优点
- 原理简单直观:无需复杂的数学推导和模型训练过程,易理解、易实现;
- 适应性强:无需假设数据分布(如线性回归假设线性关系、逻辑回归假设特征与标签的对数几率线性相关),可处理非线性分类问题;
- 动态学习:作为惰性学习算法,新增训练样本时无需重新训练模型,只需将新样本加入训练集即可,适合数据频繁更新的场景;
- 多分类天然支持:无需额外扩展(如逻辑回归需通过One-vs-Rest策略扩展多分类),直接通过投票即可处理多分类任务。
3.2 缺点
- 预测效率低 :预测时需计算待预测样本与所有训练样本的距离,时间复杂度为 O ( m n ) O(mn) O(mn)( m m m为训练样本数, n n n为特征数),不适用于大规模数据集(如百万级样本);
- 空间复杂度高:需存储全部训练样本,当训练数据量极大时,对内存占用要求高;
- 对高维数据敏感:随着特征维度增加,样本间的"距离区分度"会降低(即"维度灾难"),导致模型分类准确率下降;
- 对噪声和异常值敏感:若训练集中存在标注错误或异常值,可能会被选为"最近邻",影响预测结果;
- 可解释性弱:虽原理直观,但无法像逻辑回归那样通过权重解释特征的重要性,仅能知道"邻居是谁",无法解释"为什么是这个类别"。
4. 适用场景与不适用场景
4.1 适用场景
- 小到中等规模数据集(训练样本数 < 1 0 5 <10^5 <105),如小规模文本分类、用户兴趣推荐、医疗辅助诊断(样本量有限且需快速落地);
- 非线性分类任务,如简单的图像识别(低维特征)、商品分类(特征与类别非线性相关);
- 数据频繁更新的场景,如实时推荐系统(新增用户/商品数据后无需重新训练);
- 对模型落地速度要求高、无需复杂调参的场景(KNN核心调参仅 K K K值和距离度量)。
1.4.2 不适用场景
- 大规模数据集(如百万级以上样本):预测速度慢,无法满足实时性要求;
- 高维数据(如特征维度 > 1000 >1000 >1000):维度灾难导致分类效果差,需先通过PCA等降维算法处理;
- 对预测效率要求高的场景(如实时风控、高并发推荐):KNN预测耗时无法满足低延迟需求;
- 对模型可解释性要求高的场景(如金融风控需明确特征影响):KNN无法提供特征重要性解释。
二、KNN相关方法介绍
1. KNN构造方法
在scikit-learn中,KNN分类器通过KNeighborsClassifier类实现,回归任务则通过KNeighborsRegressor类实现。以下是KNN分类器常用关键参数说明:
| 参数 | 默认值 | 说明 |
|---|---|---|
n_neighbors |
5 |
近邻数量 K K K,核心超参数。取值为正整数,需通过交叉验证选择。 |
weights |
'uniform' |
近邻权重计算方式: • 'uniform':所有近邻权重相等(简单多数投票); • 'distance':权重与距离成反比(距离越近的样本影响越大)。 |
algorithm |
'auto' |
近邻搜索算法: • 'ball_tree':适用于高维数据; • 'kd_tree':适用于低维数据(<20维); • 'brute':暴力搜索(计算所有样本距离,适合小数据集); • 'auto':自动根据数据选择最优算法。 |
metric |
'minkowski' |
距离度量方式: • 'euclidean':欧氏距离; • 'manhattan':曼哈顿距离; • 'chebyshev':切比雪夫距离; • 'minkowski':闵可夫斯基距离(默认,配合p参数使用)。 |
p |
2 |
闵可夫斯基距离的阶数: • p=1 等价于曼哈顿距离; • p=2 等价于欧氏距离(默认)。 |
leaf_size |
30 |
构建Ball Tree或KD Tree时的叶子节点大小,影响树的构建速度和查询效率。 |
n_jobs |
None |
并行计算的CPU核心数: • None:使用1个核心; • -1:使用所有可用核心(加速大规模数据计算)。 |
参数取值说明:
(1)n_neighbors(近邻数量 K K K)
- 取值范围 :正整数(如
1,3,5,7,10) - 说明 :
- K K K越小,模型越复杂,易过拟合(对噪声敏感);
- K K K越大,模型越简单,分类边界越平滑,易欠拟合;
- 建议通过交叉验证(如
GridSearchCV)在[1, 3, 5, 7, 9, 11]中选择最优值,且优先选择奇数避免投票平局。
(2)weights(权重计算方式)
'uniform'(默认):所有近邻平等投票,适用于数据分布均匀、噪声少的场景;'distance':近邻权重为距离的倒数( w = 1 / d w=1/d w=1/d),距离越近的样本影响越大,适合数据存在噪声或边界模糊的场景,但可能增加过拟合风险。
(3)algorithm(搜索算法)
'brute':暴力计算待预测样本与所有训练样本的距离,时间复杂度 O ( m ) O(m) O(m)( m m m为训练样本数),适合样本量小(<1万)的场景;'kd_tree':通过KD树索引加速搜索,适合低维数据(特征数<20),大规模数据下效率远高于暴力搜索;'ball_tree':Ball树对高维数据的适应性优于KD树,当特征数>20时推荐使用;'auto':默认根据样本量和特征数自动选择,无需手动指定(小数据用'brute',大数据用树结构)。
(4)metric与p(距离度量)
- 欧氏距离(
metric='euclidean'或metric='minkowski'且p=2):适用于连续型特征,且特征尺度一致(需预处理); - 曼哈顿距离(
metric='manhattan'或p=1):对异常值的敏感性低于欧氏距离,适合特征存在极端值的场景; - 其他距离 :如余弦距离(
'cosine')适合高维稀疏数据(如文本),需根据特征类型选择。
2. KNN返回对象的方法介绍
使用KNeighborsClassifier训练模型后,返回的模型对象提供了一系列方法用于预测、查询近邻等操作,核心方法如下:
2.1 .fit(X, y)
- 作用:训练模型(存储训练数据,KNN无参数学习过程)。
- 输入 :
X:训练特征矩阵(形状[n_samples, n_features]);y:训练标签(形状[n_samples])。
- 输出:返回模型自身(支持链式调用)。
- 说明:KNN是惰性学习算法,此步骤仅存储数据,不进行复杂计算。
2.2 .predict(X)
- 作用:对新样本进行类别预测。
- 输入 :待预测特征矩阵
X(形状[n_samples, n_features])。 - 输出 :预测的类别标签数组(形状
[n_samples])。 - 原理 :计算每个样本的
n_neighbors个近邻,根据weights规则投票决定类别。
2.3 .predict_proba(X)
- 作用:返回每个样本属于各类别的概率。
- 输入 :待预测特征矩阵
X。 - 输出 :概率矩阵(形状
[n_samples, n_classes]),每行和为1。 - 计算方式 :
- 若
weights='uniform':某类概率 = 该类在近邻中的占比; - 若
weights='distance':某类概率 = 该类近邻的权重之和 / 所有近邻权重之和。
- 若
2.4 .kneighbors(X)
- 作用:查询待预测样本的近邻信息。
- 输入 :特征矩阵
X(默认查询训练集自身)。 - 输出 :
- 距离数组(形状
[n_samples, n_neighbors]):每个样本到其近邻的距离; - 索引数组(形状
[n_samples, n_neighbors]):近邻在训练集中的索引。
- 距离数组(形状
- 用途:分析样本的相似邻居,辅助模型解释(如"为什么这个样本被分类为A?因为它的邻居多为A类")。
2.5 .kneighbors_graph(X)
- 作用:生成样本与近邻的连接矩阵(邻接矩阵)。
- 输出 :稀疏矩阵(形状
[n_samples, n_samples]),非零元素表示样本间的近邻关系。 - 用途:用于聚类、可视化等需要样本关联信息的场景。
3. KNN返回对象的属性介绍
KNN模型对象存储了训练过程中的关键信息,主要属性如下:
3.1 .classes_
- 类型 :
ndarray,形状(n_classes,) - 含义:模型识别的所有类别标签,按升序排列。
- 用途 :明确模型支持的类别,与
predict_proba的列对应。
3.2 .n_neighbors_
- 类型:int
- 含义 :实际使用的近邻数量(即
n_neighbors参数值)。
3.3 ._fit_X
- 类型 :
ndarray,形状(n_samples, n_features) - 含义:训练时使用的特征矩阵(存储的训练数据)。
- 说明:以下划线开头,为内部属性,不建议直接修改。
3.4 ._y
- 类型 :
ndarray,形状(n_samples,) - 含义:训练时使用的标签数组(存储的训练标签)。
3.5 .effective_metric_
- 类型:str
- 含义 :实际使用的距离度量(可能由
metric和p共同决定,如'euclidean')。
4. KNN模型评估指标方法介绍
KNN作为分类模型时,评估指标与逻辑回归类似,但需结合其"基于相似性分类"的特性选择合适指标。以下是核心评估方法:
4.1 准确率(Accuracy)
-
定义:预测正确的样本占总样本的比例。
-
适用场景:类别均衡的简单场景。
-
方法 :
pythonfrom sklearn.metrics import accuracy_score acc = accuracy_score(y_true, y_pred)
4.2 混淆矩阵(Confusion Matrix)
-
作用:直观展示各类别预测结果的交叉分布,识别易混淆的类别。
-
方法 :
pythonfrom sklearn.metrics import confusion_matrix cm = confusion_matrix(y_true, y_pred)
4.3 分类报告(Classification Report)
-
包含指标:每个类别的精确率、召回率、F1-score,以及宏平均和加权平均。
-
优势:全面反映模型在各类别上的表现,尤其适合多分类评估。
-
方法 :
pythonfrom sklearn.metrics import classification_report report = classification_report(y_true, y_pred)
4.4 精确率、召回率与F1-score
-
适用场景:类别不平衡场景(如异常检测),需重点关注少数类的识别能力。
-
方法 :
pythonfrom sklearn.metrics import precision_score, recall_score, f1_score prec = precision_score(y_true, y_pred, average='weighted') # 精确率 rec = recall_score(y_true, y_pred, average='weighted') # 召回率 f1 = f1_score(y_true, y_pred, average='weighted') # F1分数
4.5 ROC曲线与AUC
-
适用场景:二分类任务,评估模型对正负样本的区分能力,不受阈值影响。
-
注意 :KNN的
predict_proba输出概率校准性较差,AUC需结合实际业务解读。 -
方法 :
pythonfrom sklearn.metrics import roc_auc_score auc = roc_auc_score(y_true, y_pred_proba) # y_pred_proba为正类概率
4.6 评估指标选择建议
| 场景 | 推荐指标 |
|---|---|
| 小规模均衡数据 | 准确率 + 混淆矩阵 |
| 类别不平衡数据 | F1-score(weighted)、召回率(关注少数类) |
| 需对比不同K值 | 交叉验证下的平均F1或AUC |
| 模型解释需求 | 结合kneighbors方法分析近邻分布,辅助理解误分类原因 |
KNN的性能高度依赖数据分布和超参数选择,评估时需重点验证不同 K K K值、距离度量和权重方式对指标的影响,避免因参数设置不当导致误判模型效果。
5. K值确定相关方法
5.1 cross_val_score()方法
cross_val_score 是 scikit-learn(sklearn)库中用于交叉验证 (Cross-Validation)的一个核心工具函数。其主要作用是评估一个机器学习模型在给定数据集上的泛化性能,尤其适用于模型选择 (如选择最优的超参数 K)和性能估计。
在 K 折交叉验证(K-Fold Cross-Validation)中,原始数据集被划分为 K 个大小相近的子集(称为"折",folds)。模型训练 K 次,每次使用其中 K−1 个子集作为训练集,剩下的 1 个子集作为验证集。最终,cross_val_score 返回 K 个验证得分(如准确率、均方误差等),可用于计算平均性能和稳定性。
该方法特别适用于确定 K 近邻(KNN)算法中的 K 值 :通过遍历多个候选 K 值,对每个 K 值调用 cross_val_score,选择使交叉验证平均得分最高的 K。
cross_val_score() 函数参数说明:
| 参数名 | 类型 | 默认值 | 说明 |
|---|---|---|---|
estimator |
estimator 对象 | --- | 实现了 fit 和 score 方法的模型对象(如 KNeighborsClassifier())。 |
X |
array-like | --- | 特征数据,形状为 (n_samples, n_features)。 |
y |
array-like | None | 目标变量(标签),形状为 (n_samples,)。对于无监督任务可省略。 |
scoring |
str / callable / None | None | 评分指标,如 'accuracy'、'neg_mean_squared_error' 等。若为 None,则使用 estimator 自带的 score 方法。 |
cv |
int / cross-validator | 5 | 交叉验证策略。若为整数(如 cv=5),表示使用 K 折交叉验证(分层 K 折用于分类);也可传入 KFold、StratifiedKFold 等对象。 |
n_jobs |
int / None | None | 并行运行的作业数。n_jobs=-1 表示使用所有 CPU 核心加速计算。 |
verbose |
int | 0 | 控制输出详细程度(越大越详细)。 |
fit_params |
dict | None | 传递给 estimator.fit() 的额外参数(如样本权重)。 |
pre_dispatch |
int / str | '2*n_jobs' | 控制并行执行时预分配的任务数量,用于节省内存。 |
5.2 GridSearchCV类
GridSearchCV 是 scikit-learn 中用于超参数调优(Hyperparameter Tuning)的核心工具类。其核心思想是在给定的超参数网格(grid)中,穷举所有可能的参数组合,通过交叉验证评估每组参数的性能,最终选择表现最优的一组参数。
该方法特别适用于像 K 近邻(KNN)、支持向量机(SVM)、决策树等具有多个可调超参数的模型。例如,在 KNN 中,可以同时搜索 n_neighbors(K 值)、weights(距离加权与否)、metric(距离度量方式)等参数的最佳组合。
GridSearchCV 内部自动调用交叉验证(如 cross_val_score 的机制),对每一组参数进行多次训练与验证,避免因数据划分偶然性导致的过拟合或评估偏差。最终,它不仅返回最优参数组合,还会基于整个训练集使用最优参数重新训练一个完整模型,可供直接预测使用。
GridSearchCV 构造函数参数说明:
| 参数名 | 类型 | 默认值 | 说明 |
|---|---|---|---|
estimator |
estimator 对象 | --- | 需要调优的模型对象(必须实现 fit 和 score 方法)。 |
param_grid |
dict 或 list of dict | --- | 超参数搜索空间。例如:{'n_neighbors': [3,5,7], 'weights': ['uniform','distance']}。若为列表,则表示多个不同参数组合策略(用于不同模型结构)。 |
scoring |
str / callable / list / None | None | 评估指标。可为单个字符串(如 'accuracy')、可调用函数,或多个指标组成的列表/字典(多指标评估)。若为 None,使用 estimator 自带的评分方法。 |
cv |
int / cross-validator | 5 | 交叉验证策略。整数表示 K 折(分类任务默认分层 K 折),也可传入 KFold、StratifiedKFold 等对象。 |
n_jobs |
int / None | None | 并行任务数。n_jobs=-1 表示使用所有 CPU 核心加速搜索。 |
verbose |
int | 0 | 控制输出详细程度(值越大,日志越详细)。 |
pre_dispatch |
int / str | '2*n_jobs' | 控制并行时预分配的任务数量,用于平衡内存与效率。 |
refit |
bool / str | True | 是否在找到最佳参数后,用全部训练数据重新训练模型。若为 True,则 best_estimator_ 可直接用于预测;若为字符串(多指标时),指定以哪个指标为准进行 refit。 |
return_train_score |
bool | False | 是否在 cv_results_ 中包含训练得分(默认不包含,因可能增加计算开销)。 |
GridSearchCV 主要属性:
| 属性名 | 类型 | 说明 |
|---|---|---|
best_estimator_ |
estimator | 使用最优参数在整个训练集上重新训练得到的模型(前提是 refit=True)。 |
best_score_ |
float | 最优参数在交叉验证中的平均得分(注意:不是测试集得分)。 |
best_params_ |
dict | 最优的超参数组合,例如 {'n_neighbors': 5, 'weights': 'distance'}。 |
best_index_ |
int | cv_results_ 中对应最优结果的索引位置。 |
cv_results_ |
dict | 包含所有参数组合的详细交叉验证结果(如每折得分、平均得分、标准差等),可用于分析或可视化。 |
n_splits_ |
int | 交叉验证的折数(由 cv 参数决定)。 |
refit_time_ |
float | (sklearn ≥0.22)重新训练最优模型所用的时间(秒)。 |
multimetric_ |
bool | 是否启用了多指标评估(即 scoring 为多个指标)。 |
三、KNN实现步骤(分类)
目标:使用K-最近邻(K-Nearest Neighbors, KNN)算法来预测威斯康星乳腺癌数据集的诊断结果,区分肿瘤为良性(Benign)或恶性(Malignant)。
1. 数据收集与加载
在KNN分类实践中,选择合适的数据集是验证算法效果的基础。本节以威斯康星乳腺癌数据集(Wisconsin Breast Cancer Dataset) 为例,演示KNN分类的完整实现流程。该数据集因特征明确、类别分布相对均衡,是分类算法入门的经典案例。
1.1 威斯康星乳腺癌数据集介绍
1.1.1 数据集基本信息
威斯康星乳腺癌数据集由美国威斯康星大学的研究人员收集,包含乳腺肿块的细针穿刺(FNA)活检数据,主要用于良性/恶性肿瘤的二分类任务。
- 来源:由Dr. William H. Wolberg于1995年公开,现收录于UCI机器学习仓库和Scikit-learn内置数据集;
- 样本量:569个样本(每个样本代表一个乳腺肿块的活检结果);
- 类别分布 :
- 良性(Benign):357个样本(占比约62.7%);
- 恶性(Malignant):212个样本(占比约37.3%);
- 特征类型:全部为连续型数值特征,基于肿块细胞核的图像特征提取;
- 任务目标:根据细胞核特征预测肿瘤为良性(0)或恶性(1)。
该数据集的特点是特征与目标强相关(细胞核形态是判断肿瘤性质的关键依据),且无缺失值,无需复杂的预处理,非常适合验证KNN等基础分类算法的效果。
1.1.2 数据集字段说明
威斯康星乳腺癌数据集包含31个字段,其中1个为目标标签,30个为特征字段(按"均值""标准误差""最差值"三类分组),具体如下:
| 类别 | 字段英文名称 | 字段中文说明 | 描述 |
|---|---|---|---|
| 目标变量 | diagnosis | 诊断结果 | 0=恶性(Malignant), 1=良性(Benign) |
| 均值特征 | mean radius | 平均半径 | 细胞核半径的算术平均值 |
| mean texture | 平均纹理 | 细胞核纹理的灰度值标准差 | |
| mean perimeter | 平均周长 | 细胞核周长的算术平均值 | |
| mean area | 平均面积 | 细胞核面积的算术平均值 | |
| mean smoothness | 平均平滑度 | 半径长度的局部变化 | |
| mean compactness | 平均紧凑度 | 周长² / 面积 - 1.0 | |
| mean concavity | 平均凹度 | 轮廓凹部分的严重程度 | |
| mean concave points | 平均凹点 | 轮廓凹部分的数量 | |
| mean symmetry | 平均对称性 | 细胞核的对称性 | |
| mean fractal dimension | 平均分形维数 | 海岸线近似 - 1.0 | |
| 标准误差特征 | radius error | 半径误差 | 半径测量的标准误差 |
| texture error | 纹理误差 | 纹理测量的标准误差 | |
| perimeter error | 周长误差 | 周长测量的标准误差 | |
| area error | 面积误差 | 面积测量的标准误差 | |
| smoothness error | 平滑度误差 | 平滑度测量的标准误差 | |
| compactness error | 紧凑度误差 | 紧凑度测量的标准误差 | |
| concavity error | 凹度误差 | 凹度测量的标准误差 | |
| concave points error | 凹点误差 | 凹点测量的标准误差 | |
| symmetry error | 对称性误差 | 对称性测量的标准误差 | |
| fractal dimension error | 分形维数误差 | 分形维数测量的标准误差 | |
| 最差值特征 | worst radius | 最差半径 | 最大的细胞核半径 |
| worst texture | 最差纹理 | 最差的纹理值 | |
| worst perimeter | 最差周长 | 最大的细胞核周长 | |
| worst area | 最差面积 | 最大的细胞核面积 | |
| worst smoothness | 最差平滑度 | 最差的平滑度值 | |
| worst compactness | 最差紧凑度 | 最差的紧凑度值 | |
| worst concavity | 最差凹度 | 最差的凹度值 | |
| worst concave points | 最差凹点 | 最差的凹点值 | |
| worst symmetry | 最差对称性 | 最差的对称性值 | |
| worst fractal dimension | 最差分形维数 | 最差的分形维数值 |
特征统计摘要
| 特征类别 | 特征数量 | 测量内容 |
|---|---|---|
| 均值特征 | 10个 | 细胞核特征的算术平均值 |
| 标准误差特征 | 10个 | 特征测量的变异程度 |
| 最差值特征 | 10个 | 细胞核最异常的特征值 |
这些特征都是从乳腺肿块的细针穿刺(FNA)数字化图像中提取的细胞核特征,用于帮助医生区分恶性肿瘤和良性肿瘤。
1.2 数据加载
Scikit-learn库内置了威斯康星乳腺癌数据集,可直接通过datasets模块加载,避免手动下载和解析文件。以下是加载代码:
python
import pandas as pd
from matplotlib import pyplot as plt
from sklearn.datasets import load_breast_cancer
# 设置matplotlib的中文字体为SimHei(黑体),以确保中文标签可以正常显示。
plt.rcParams['font.sans-serif'] = ['SimHei']
# 解决负号'-'显示为方块的问题,通过设置'axes.unicode_minus'为False来实现。
plt.rcParams['axes.unicode_minus'] = False
# 设置全局选项
pd.set_option('display.max_columns', None) # 显示所有列
pd.set_option('display.width', None) # 自动检测宽度
pd.set_option('display.max_colwidth', 50) # 列内容最多显示50字符
pd.set_option('display.expand_frame_repr', False) # 禁用多行表示(可选)
# 加载数据集
cancer = load_breast_cancer()
# 查看数据集结构(可选)
# print("数据集包含的键:", cancer.keys()) # 输出数据集的核心属性
# print(cancer.DESCR)
# 将特征和标签转换为DataFrame,便于查看
# 特征数据(30个特征)
X = pd.DataFrame(data=cancer.data, columns=cancer.feature_names)
# 标签数据(0=恶性,1=良性,注意:原数据集默认0为恶性,1为良性)
y = pd.Series(data=cancer.target, name='diagnosis')代码说明:
load_breast_cancer():Scikit-learn内置函数,返回一个Bunch对象(类似字典),包含数据集的特征、标签、描述等信息;- 核心属性 :
cancer.data:特征矩阵(numpy数组),形状为(569, 30);cancer.feature_names:30个特征的名称列表;cancer.target:标签数组(0=恶性,1=良性);cancer.target_names:标签名称(['malignant', 'benign']);
2. 数据探索与可视化(EDA)
数据探索与可视化(Exploratory Data Analysis, EDA)是建模前的关键步骤,通过分析数据分布、特征相关性和类别差异,可帮助我们理解数据特点,为后续预处理和模型调优提供依据。以下针对威斯康星乳腺癌数据集展开EDA:
2.1 数据基本信息
2.1.1 查看数据前10行
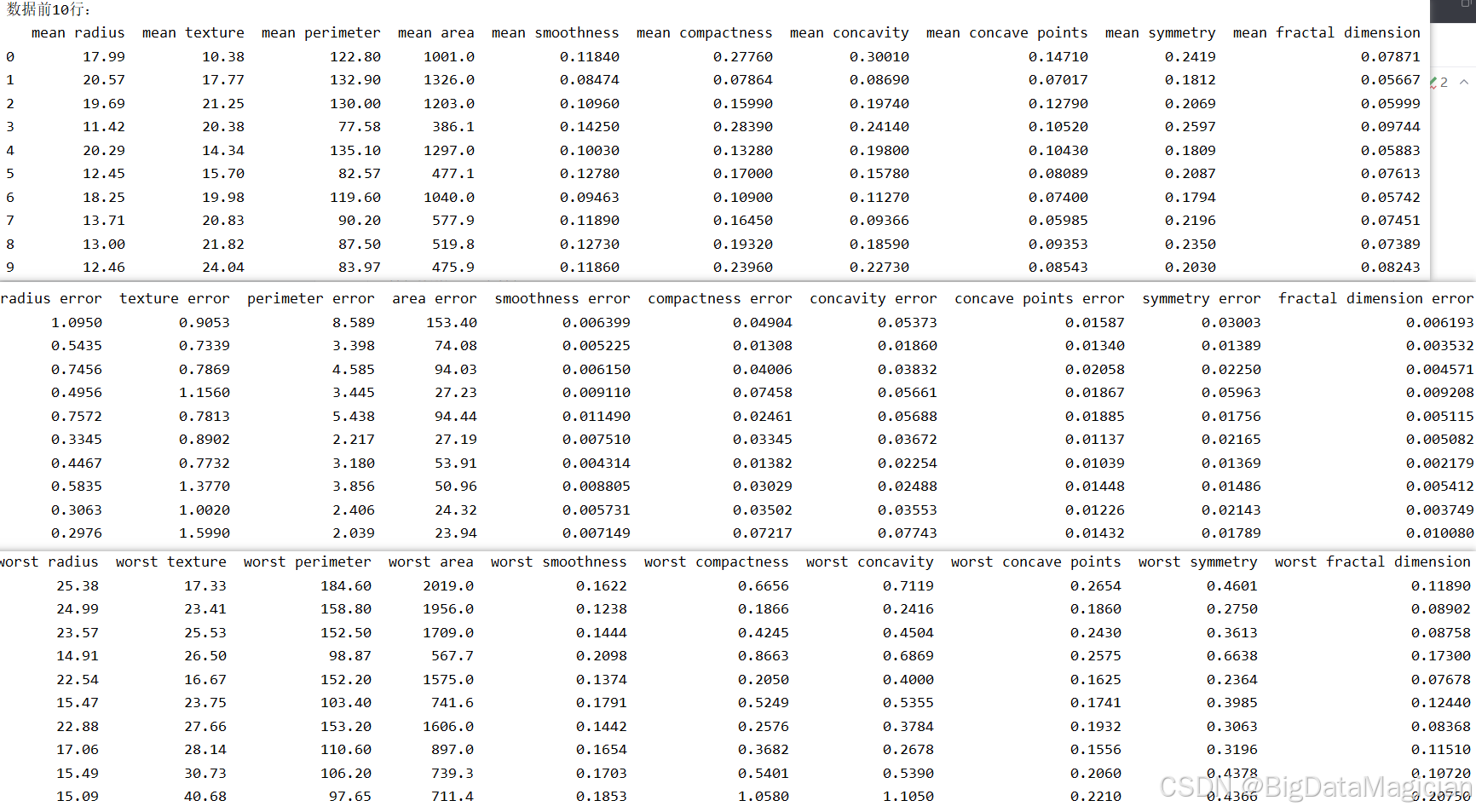
在数据分析的初始阶段,查看数据集的前几行是了解其结构、字段含义和数据类型的重要步骤。通过观察原始数据的分布与格式,可以初步判断是否存在异常值、缺失值或不一致的命名规范,并为后续的数据清洗、特征工程和建模工作提供直观依据。
该代码将特征矩阵 X(包含所有数值型特征)与标签向量 y(类别标签)沿列方向进行拼接,形成一个完整的 DataFrame,便于统一查看样本的完整信息;随后调用 head(10) 输出前10个样本,快速呈现数据结构和内容。
python
# 合并特征和标签,便于同时查看
data = pd.concat([X, y], axis=1) # 按列合并
print("数据前10行:")
print(data.head(10))数据集中各字段部分数据如下图所示,从输出结果可见,数据集中包含多个描述肿瘤形态学特征的数值变量,如 mean radius(平均半径)、mean texture(平均纹理)、mean perimeter(平均周长)等,均为连续型数值。此外还包含误差项(如 radius error)和最差情况指标(如 worst radius),反映肿瘤的异质性与恶化程度。
2.1.2 查看数据摘要信息
在数据探索阶段,了解数据集的整体结构和基本属性是确保后续分析顺利进行的关键步骤。info() 方法提供了关于 DataFrame 的综合信息,包括样本数量、特征数量、各列的数据类型以及非空值计数,有助于快速判断是否存在缺失值、数据类型是否一致等问题。
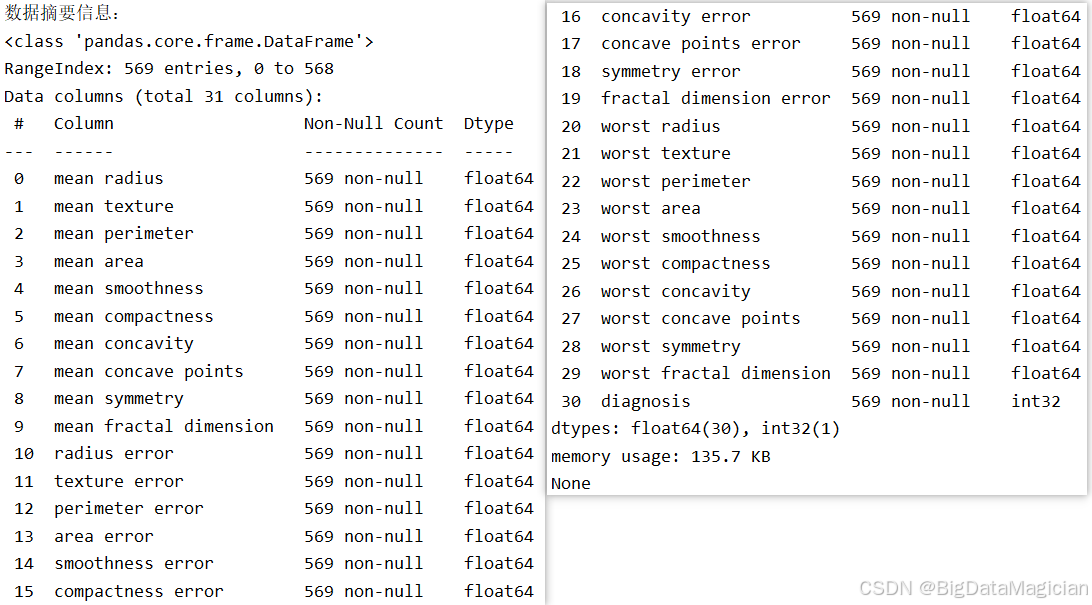
该代码调用 Pandas 的 info() 方法,输出数据框的元信息。它会显示数据集的总行数(样本数)、列数(字段数)、每列的名称、非空值数量(Non-Null Count)以及对应的数据类型(Dtype),从而全面反映数据的结构特征。
python
print("\n数据摘要信息:")
print(data.info())从输出结果可见,该数据集共包含 569 个样本 和 31 个字段 ,其中前 30 列为肿瘤形态学特征,均为 float64 类型,表示连续数值;最后一列为标签列 diagnosis,类型为 int32,代表二分类结果(0 表示恶性,1 表示良性)。所有列的 Non-Null Count 均为 569,表明 无任何缺失值,数据完整性高,无需进行缺失值填充或删除操作。此外,数据类型统一且符合预期,无需转换,极大简化了预处理流程,为后续建模提供了高质量的数据基础。
2.1.3 特征描述性统计
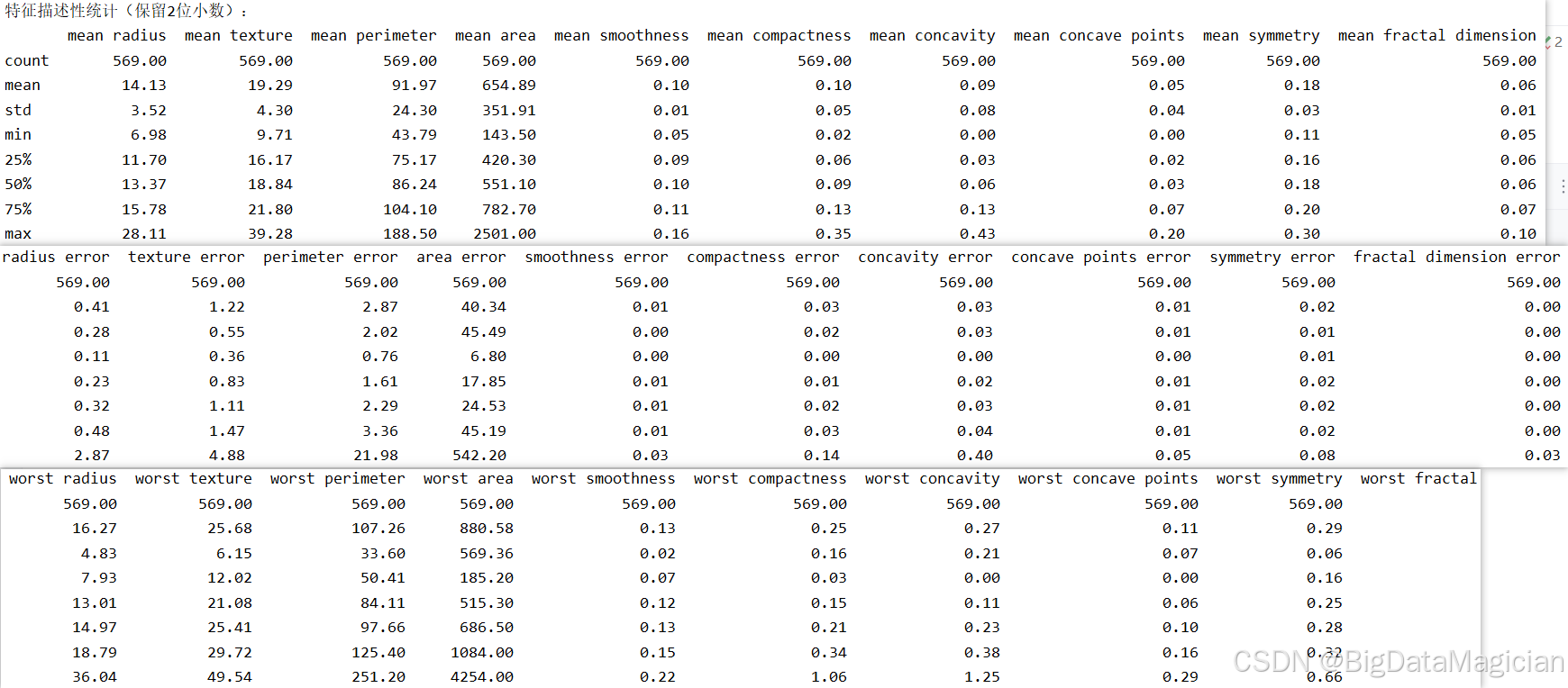
在数据探索过程中,描述性统计分析是理解特征分布特性的核心手段。通过 describe() 方法可以快速获取每个数值型特征的关键统计指标,如均值、标准差、四分位数和极值等,从而评估其集中趋势、离散程度与分布形态。本节对特征矩阵进行描述性统计分析,旨在揭示各特征的数值范围、变异程度及潜在偏态情况,为后续的数据预处理(如标准化)和建模策略提供依据。
该代码调用 Pandas 的 describe() 方法,计算特征矩阵 X 的基本统计量,包括样本数量(count)、均值(mean)、标准差(std)、最小值(min)、25%分位数(25%)、中位数(50%)、75%分位数(75%)和最大值(max)。使用 .round(2) 将结果保留两位小数,提升可读性,便于直观比较不同特征的分布特性。
python
print("\n特征描述性统计(保留2位小数):")
print(X.describe().round(2)) # 仅对特征进行统计,保留2位小数从输出结果可见,30个特征在数值范围和离散程度上存在显著差异。例如,area_worst 最大值达 4254.00,最小值为 135.80,极差超过 4100,而 fractal_dimension_mean 的极差仅为 0.16,表明不同特征的尺度差异极大。这种不一致性会导致距离类算法(如 KNN)在计算样本间相似度时,高尺度特征(如面积)主导距离贡献,而低尺度特征(如纹理误差)被忽略,造成模型偏差。此外,部分特征的标准差较大(如 area_mean 标准差为 351.91),说明数据波动剧烈;而另一些特征(如 smoothness_se 标准差仅 0.01)则高度集中。分布形态方面,多数特征的均值与中位数接近(如 radius_mean 均值 14.13 vs 中位数 13.37),呈近似对称分布;但也有特征如 area_se 均值(56.14)远大于中位数(24.53),呈现明显右偏,提示存在异常值或长尾分布。综上,这些特征需进行标准化处理以消除尺度影响,并结合偏态情况考虑是否进行对数变换等进一步预处理。
2.2 目标变量类别分布分析
类别分布分析是分类任务中至关重要的探索性步骤,用于评估目标变量的样本比例是否均衡。若某一类别样本显著少于其他类别,则可能导致模型偏向多数类,影响少数类的识别能力。本节通过可视化手段展示乳腺癌数据集中良性与恶性肿瘤的样本数量分布,判断是否存在类别不平衡问题,并为后续建模策略(如是否采用过采样、欠采样或调整类别权重)提供依据。
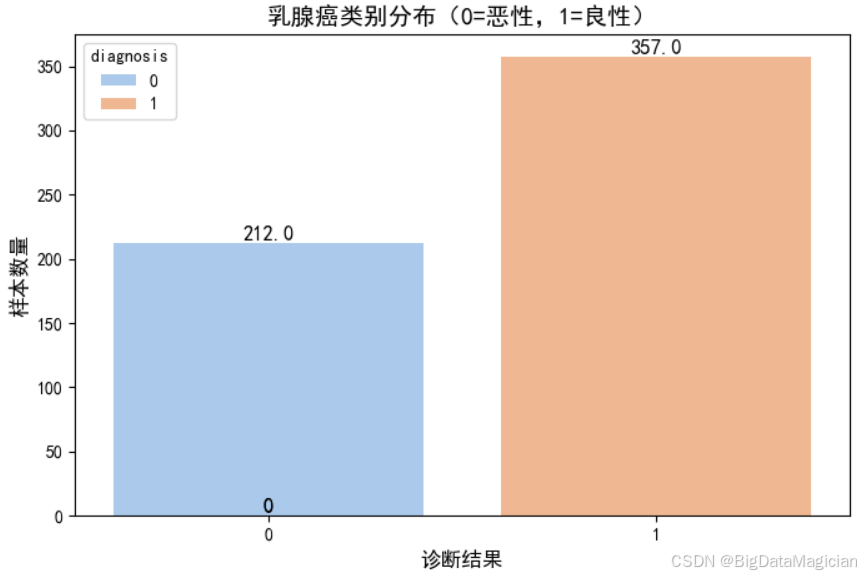
该代码使用 Seaborn 的 countplot 绘制目标变量 y 的频数分布图,其中 x=y 表示以诊断结果为分类变量。通过设置中文字体和样式增强可读性,同时在每个柱子上方添加具体数值标签,直观显示各类别样本数量。最终将图表保存为图像文件,便于报告展示。
python
import matplotlib.pyplot as plt
import seaborn as sns
# 设置可视化风格
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
print("\n目标变量类别分布:")
print(y.value_counts())
# 绘制类别分布柱状图
plt.figure(figsize=(8, 5))
ax = sns.countplot(x=y, hue=y, palette='pastel')
ax.set_title('乳腺癌类别分布(0=恶性,1=良性)', fontsize=14)
ax.set_xlabel('诊断结果', fontsize=12)
ax.set_ylabel('样本数量', fontsize=12)
# 添加数值标签
for p in ax.patches:
ax.text(s=p.get_height(), x=p.get_x() + p.get_width() / 2., y=p.get_height(), ha='center', va='bottom', fontsize=12)
plt.savefig('目标变量类别分布.png')从生成的柱状图可见,良性肿瘤(标签为 1)样本数量为 357 个,恶性肿瘤(标签为 0)样本数量为 212 个,两类样本比例约为 1.7:1。虽然良性样本略多,但未出现极端不平衡(如 9:1 或更高),属于机器学习中可接受的范围。在此比例下,大多数分类算法(如逻辑回归、随机森林)仍能有效学习两类特征差异,无需额外处理类别不平衡问题。
2.3 目标变量类别与特征相关性分析
在分类任务中,理解各特征与目标变量之间的关联强度有助于识别对预测结果影响最大的关键特征。本节通过计算每个特征与诊断标签(良性/恶性)之间的斯皮尔曼相关系数,评估其单调关系的强弱,从而筛选出具有高判别能力的特征。由于目标变量为二分类整数标签(0/1),使用非参数的斯皮尔曼相关系数更为稳健,能有效捕捉非线性趋势。结合可视化排序,可直观呈现特征的重要性,为后续特征选择和模型优化提供依据。
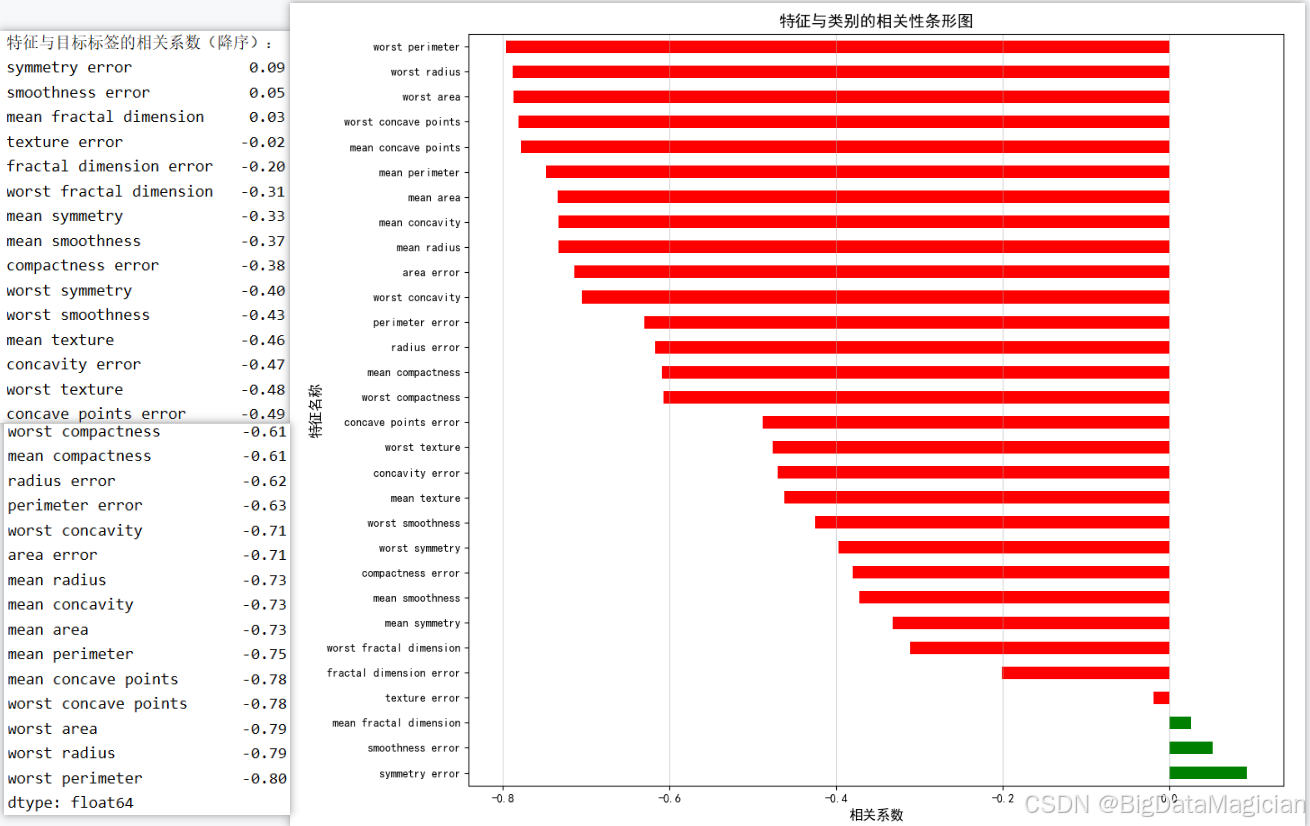
该代码首先调用 X.corrwith(y, method='spearman') 计算每个特征与标签之间的斯皮尔曼相关系数,并按降序排列;随后绘制水平条形图,正相关系数以绿色表示,负相关以红色表示,便于快速识别关键特征。图形清晰展示各特征对分类任务的贡献方向与强度。
python
# 计算每个特征与目标标签的相关系数(斯皮尔曼相关系数)
feature_label_corr = X.corrwith(y, method='spearman').sort_values(ascending=False)
print("特征与目标标签的相关系数(降序):")
print(feature_label_corr.round(2))
# 绘制水平柱状图
plt.figure(figsize=(12, 10))
colors = ['green' if x > 0 else 'red' for x in feature_label_corr.values]
feature_label_corr.plot(kind='barh', color=colors)
plt.title('特征与类别的相关性条形图', fontsize=14)
plt.xlabel('相关系数', fontsize=12)
plt.ylabel('特征名称', fontsize=12)
plt.tight_layout()
plt.grid(axis='x', alpha=0.5)
plt.savefig('特征与类别相关性排序.png')从输出结果可见,多数特征与诊断标签呈显著负相关,其中 worst perimeter、worst radius、worst area 等"最差"系列特征相关系数接近 -0.8,表明其数值越大,越倾向于恶性肿瘤,是区分良恶性的最强指标。而 mean concavity、mean concave points、worst concavity 等反映边缘不规则性的特征也表现出较强负相关(约 -0.7~ -0.8),进一步验证了恶性肿瘤形态更不规则的医学规律。少数特征如 symmetry error、smoothness error 相关系数接近 0,表明其对分类贡献较小。整体来看,尺寸和凹陷类特征主导分类过程,在KNN建模时应优先保留这些高相关特征,剔除低相关项以提升模型效率与准确性。
2.4 关键特征分布分析
在分类任务中,理解关键特征在不同类别之间的分布差异对于模型构建具有重要意义。本节通过绘制核密度估计图(Kernel Density Estimate, KDE) ,直观展示多个与诊断结果高度相关的特征在良性(diagnosis=1)和恶性(diagnosis=0)两类样本中的概率密度分布情况。KDE是一种非参数方法,能够平滑地估计数据的概率密度函数,从而揭示数据的分布形态、集中趋势及分离程度。
从医学角度出发,乳腺肿瘤的良恶性往往与其细胞核的形态学异常程度密切相关:恶性肿瘤通常表现出更大的尺寸、更高的不规则性(如凹陷、边缘粗糙等)。因此,我们重点关注那些与"最差"(worst)或"凹度"(concavity)相关的特征,这些特征在先前的相关性分析中已显示出较强的判别能力。通过观察这些特征在两类样本中的分布是否明显分离,可以判断其作为分类依据的有效性,并为后续特征选择和模型训练提供支持。
python
# 选择关键特征
key_features = feature_label_corr[abs(feature_label_corr) > 0.5].index.tolist()
# 绘制核密度图,KDE:核密度估计
plt.figure(figsize=(18, 12))
feature = key_features
for i in range(len(feature)):
plt.subplot(3, 5, i + 1)
sns.kdeplot(
data=data,
x=feature[i],
hue='diagnosis',
fill=True,
common_norm=False, # 每个类别单独归一化
alpha=0.5
)
plt.title(f'{feature[i]} 分布')
plt.xlabel(feature[i])
plt.tight_layout()
plt.savefig('乳腺癌关键特征KDE分布.png')从生成的核密度图可以看出,多个关键特征在良性与恶性肿瘤之间呈现出明显的分布差异:
-
worst_perimeter、worst_radius、worst_area:这三个"最差"系列的尺寸特征中,恶性肿瘤(蓝色)的密度峰值明显偏向更高数值区间,而良性肿瘤(橙色)集中在较低值区域。这表明恶性肿瘤的细胞核通常更大、更异常,具备更强的区分度。 -
mean_concavity、worst_concavity、mean_concave_points、worst_concave_points:反映细胞核边缘凹陷程度的特征,在恶性样本中表现出更高的值和更宽的分布范围,且与良性样本的分布几乎无重叠,说明这些特征对识别恶性肿瘤极为敏感。 -
mean_compactness、worst_compactness:紧凑度越高表示形状越不规则,恶性肿瘤在这两项上的分布也显著偏移至高值区,进一步验证了恶性肿瘤形态异质性强的特点。 -
radius_error、perimeter_error、area_error:误差项反映了测量的波动性,恶性肿瘤在这些指标上也表现出更大的变异性和更高的均值,可能暗示其结构不稳定。
总体来看,大多数高相关特征在两类样本间具有良好的可分性,尤其是在"最差"系列和"凹度"类特征上,分布几乎不重叠,说明它们能有效帮助KNN算法进行邻近点搜索和分类决策。这些发现不仅增强了我们对数据的理解,也为后续建模提供了明确的方向,应优先保留这些具有强判别能力的特征,以提升KNN模型的预测性能。

2.5 关键特征相关性分析
在构建分类模型之前,理解特征之间的相互关系至关重要。高度相关的特征可能导致多重共线性 问题,影响模型的稳定性和解释性,尤其是在基于距离的算法(如KNN)中,冗余特征可能放大某些维度的影响,降低模型泛化能力。因此,本节通过计算关键特征之间的皮尔逊相关系数,并绘制热力图,直观展示各特征间的线性相关程度。
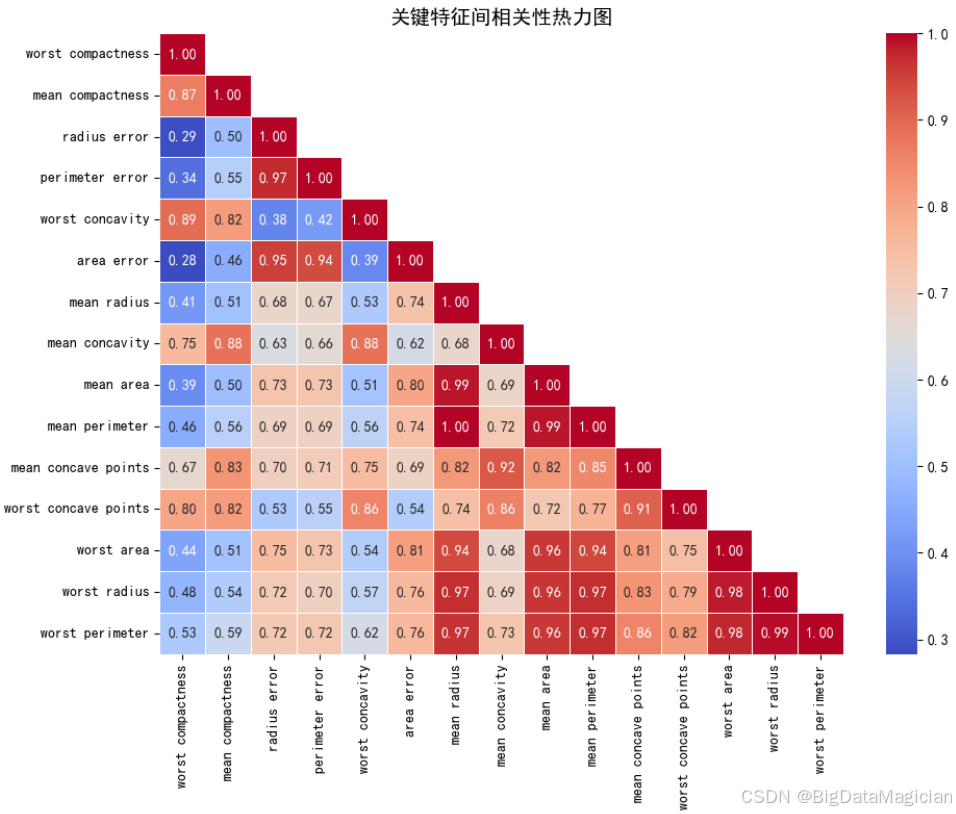
选取了在前序分析中特征与类别相关性绝对值大于0.5的关键特征,这些特征主要反映肿瘤细胞核的尺寸、形状不规则性和边缘凹陷等重要形态学指标。通过分析它们之间的相关性,可以识别出是否存在信息重叠的特征对,从而为后续的特征选择或降维提供依据。例如,若两个特征高度正相关(如 >0.8),则可考虑保留其中一个以减少冗余,提升模型效率与鲁棒性。
python
import numpy as np
# 计算特征间的相关系数(皮尔逊相关系数)
corr_matrix = X[key_features].corr(method='pearson')
# 创建掩码,只保留下三角部分(包括对角线)
mask = np.triu(m=np.ones_like(corr_matrix, dtype=bool), k=1)
# 绘制热力图
plt.figure(figsize=(10, 8))
sns.heatmap(data=corr_matrix, mask=mask, annot=True, cmap='coolwarm', fmt='.2f', linewidths=0.5)
plt.title('关键特征间相关性热力图', fontsize=14)
plt.tight_layout()
plt.savefig('关键特征间相关性热力图.png')从生成的热力图可以看出,所选关键特征之间普遍存在较强的正相关关系,符合生物学逻辑:
-
尺寸类特征高度相关:
worst_radius、worst_perimeter、worst_area三者之间的相关系数均超过 0.97,表明它们在描述肿瘤最大形态时几乎完全一致,属于高度冗余的特征。- 同样,
mean_radius、mean_perimeter、mean_area也表现出较强正相关(>0.7),说明平均尺寸特征也具有一定的信息重叠。
-
凹陷与不规则性特征群:
mean_concavity与mean_concave_points相关系数达 0.82 ,worst_concavity与worst_concave_points达 0.91,说明凹陷程度与凹点数量密切相关,二者共同反映了细胞核边缘的异常程度。- 此外,
mean_compactness与mean_concavity的相关系数为 0.88,进一步验证了"紧凑度"与"凹度"在几何上的对立关系(越凹则越不紧凑)。
-
误差项与主特征相关性较弱:
- 如
radius_error、area_error等虽与其他特征有一定相关性(约0.3~0.5),但整体偏低,说明测量波动性相对独立于总体形态,可作为补充信息保留。
- 如
综上所述,该热力图揭示了多个特征组合之间的强相关性 ,提示我们在后续建模过程中应谨慎处理这些冗余变量。例如,在KNN中,如果同时包含worst_radius和worst_area,由于它们几乎线性相关,可能会导致某一个特征主导距离计算,从而降低模型鲁棒性。
2.6 关键特征的类别区分能力验证
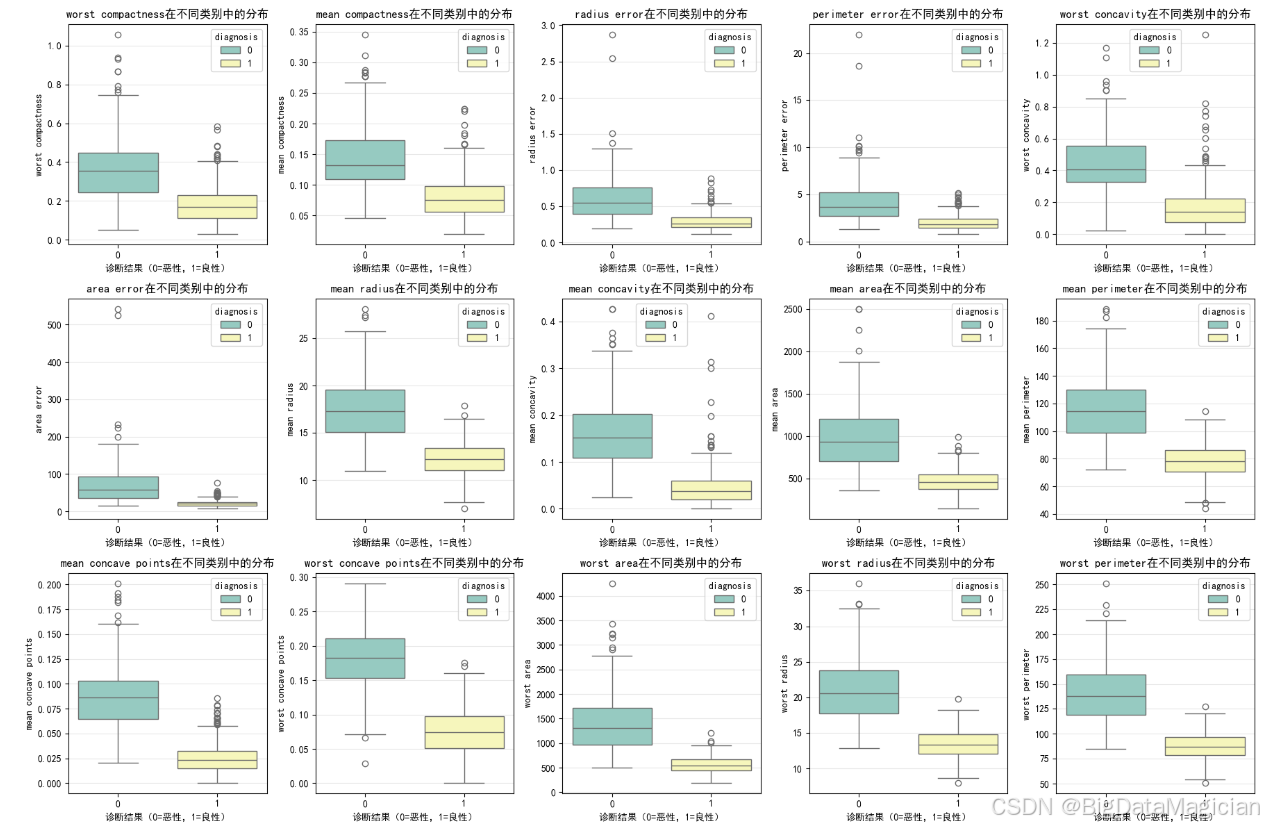
在分类任务中,特征的判别能力(即其在不同类别之间是否存在显著差异)是决定模型性能的关键因素。为了进一步验证前文筛选出的高相关性特征是否具备良好的类别区分效果,本节采用箱线图(Boxplot) 对这些关键特征在良性(diagnosis=1)与恶性(diagnosis=0)两类样本中的分布进行可视化对比。
箱线图能够清晰地展示数据的中位数、四分位距、异常值等统计信息,特别适合用于比较两个或多个组之间的分布差异。通过观察各类别下特征的中心位置(中位数)、离散程度(箱体高度)以及异常值分布,可以直观判断该特征是否能有效将两类样本区分开来。例如,若某一特征在恶性肿瘤中的中位数远高于良性肿瘤,且两组箱体无重叠,则说明该特征具有很强的判别力。
选取了与目标变量斯皮尔曼相关系数绝对值大于0.5的特征作为"关键特征",这些特征已在相关性分析中被确认为对分类结果有较强影响。接下来通过箱线图验证其实际分布上的可分性,从而为后续建模提供更可靠的依据。
python
# 选择关键特征
key_features = feature_label_corr[abs(feature_label_corr) > 0.5].index.tolist()
# 绘制箱线图
plt.figure(figsize=(18, 12))
# enumerate() 函数同时返回索引和元素值,参数 1 表示索引从 1 开始(默认从 0 开始)
for i, feature in enumerate(key_features, 1):
plt.subplot(3, 5, i)
sns.boxplot(x=y, y=data[feature], hue=y, palette='Set3')
plt.title(f'{feature}在不同类别中的分布', fontsize=12)
plt.xlabel('诊断结果(0=恶性,1=良性)', fontsize=10)
plt.ylabel(feature, fontsize=10)
plt.grid(axis='y', alpha=0.3)
plt.tight_layout()
plt.savefig('关键特征类别区分箱线图.png')从箱线图可见,所选关键特征在良性(1)与恶性(0)肿瘤之间均表现出显著的分布差异。例,尤其是在"最差"系列和"凹度"类特征上,箱体几乎没有交集,说明它们对区分良恶性具有极高的判别价值。此外,多数特征的四分位距(IQR)较小,说明数据集中趋势明确,异常值较少。虽然部分特征如 mean radius 存在少量重叠,但总体仍能有效分离两类。这验证了相关性分析的结果,确认这些特征是构建KNN模型的理想输入,可显著提升分类性能。
从图中可见部分特征存在异常值,但这些值源于真实有效的医学观测,反映了肿瘤在形态学上的极端但合理的生物学变异,具有重要的临床意义,因此无需剔除或修正。保留这些样本有助于模型更好地识别高风险恶性病例,提升实际应用中的泛化能力与鲁棒性。
3. 数据预处理
数据预处理是KNN建模的关键步骤,其核心目标是消除特征尺度差异、处理冗余特征,确保距离计算的合理性,提升模型泛化能力。结合EDA结论,本节将开展特征筛选、数据划分、特征标准化等预处理操作。
3.1 特征筛选
在构建KNN分类模型前,合理的特征筛选对提升模型性能至关重要。本节采用两阶段策略,首先保留与诊断标签斯皮尔曼相关系数绝对值大于0.5的特征,确保其具备强判别能力;随后通过方差膨胀因子(VIF)检测并剔除存在严重多重共线性的特征(通常VIF > 10视为强共线性),以避免冗余信息干扰距离计算,从而获得一个既有效又独立的特征子集。
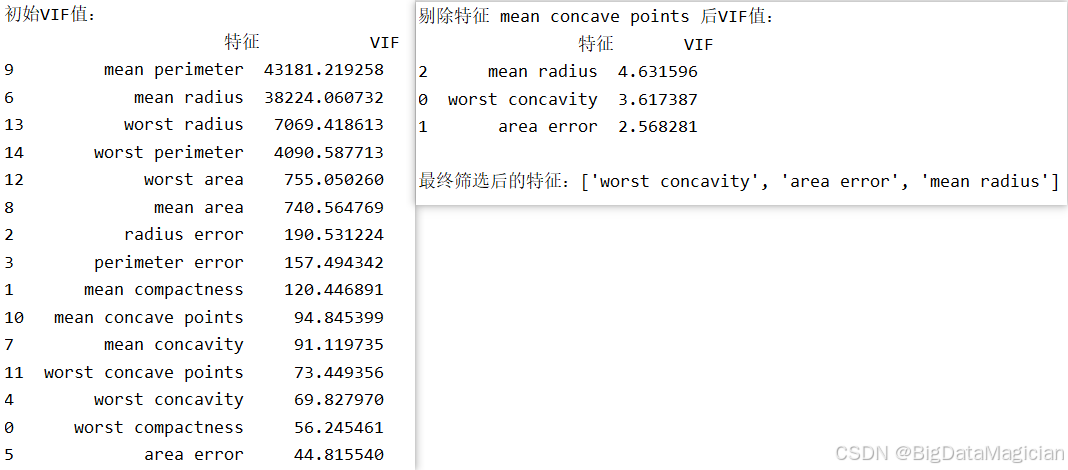
代码首先从相关性分析结果中提取高相关特征列表,然后定义一个计算VIF的函数,利用statsmodels库中的variance_inflation_factor逐个评估每个特征的共线性程度。接着,程序进入循环,只要存在VIF超过10的特征,就剔除当前VIF最高的那个,并重新计算剩余特征的VIF,直至所有特征均满足共线性阈值要求,最终输出筛选后的特征名称。
python
# 筛选与目标标签相关性绝对值>0.5的特征(强区分能力)
key_features = feature_label_corr[abs(feature_label_corr) > 0.5].index.tolist()
print(f"高相关特征数量:{len(key_features)}")
# 计算VIF剔除多重共线性特征
from statsmodels.stats.outliers_influence import variance_inflation_factor
def calculate_vif(X):
vif_data = pd.DataFrame()
vif_data["特征"] = X.columns
vif_data["VIF"] = [variance_inflation_factor(X.values, i) for i in range(X.shape[1])]
return vif_data.sort_values("VIF", ascending=False)
# 初始VIF计算
vif_df = calculate_vif(X[key_features])
print("\n初始VIF值:")
print(vif_df)
# 剔除VIF>10的特征(通常认为VIF>10存在强共线性)
while vif_df["VIF"].max() > 10:
# 删除VIF最大的特征
drop_feature = vif_df.iloc[0]["特征"]
key_features.remove(drop_feature)
vif_df = calculate_vif(X[key_features])
print(f"\n剔除特征 {drop_feature} 后VIF值:")
print(vif_df)
print(f"\n最终筛选后的特征:{key_features}")结果显示,初始14个高相关特征中存在严重的多重共线性,例如mean_perimeter的VIF高达4万余,主要源于尺寸类特征(如半径、周长、面积)之间的高度线性关系。经过迭代剔除后,最终保留了三个特征:worst_concavity(反映恶性肿瘤边缘凹陷程度)、area_error(体现肿瘤内部异质性)和mean_radius(代表细胞核平均大小)。这三个特征不仅判别能力强,且彼此间共线性低,为后续KNN建模提供了简洁而有效的输入。
3.2 数据集划分
在机器学习建模流程中,将数据划分为训练集和测试集是评估模型泛化能力的关键步骤。本节基于前一步筛选出的三个关键特征(worst_concavity、area_error、mean_radius),采用分层抽样策略对威斯康星乳腺癌数据集进行划分,确保训练集与测试集在类别分布上保持一致。由于原始数据集中良性样本占比约63%,恶性占37%,若不进行分层采样,可能导致测试集中某一类别比例失真,进而影响模型性能评估的可靠性。通过合理的划分,能够更真实地反映模型在实际应用中的表现。
代码使用 sklearn.model_selection.train_test_split 函数实现数据分割,设置 test_size=0.2 表示测试集占总样本的20%,即114个样本,其余455个用于训练。random_state=42 确保结果可复现,而 stratify=y 参数保证了训练集和测试集中良性与恶性肿瘤的比例与原始数据一致。此外,输出各子集的形状和类别分布,便于验证划分是否合理。
python
from sklearn.model_selection import train_test_split
# 特征矩阵 - 使用经过相关性筛选和多重共线性处理后的关键特征
X_selected = X[key_features]
# 划分训练集(80%)和测试集(20%)
X_train, X_test, y_train, y_test = train_test_split(
X_selected, y,
test_size=0.2,
random_state=42,
stratify=y # 分层抽样,保持类别分布一致
)
# 查看划分后的数据形状
print(f"训练集特征矩阵形状:{X_train.shape}")
print(f"测试集特征矩阵形状:{X_test.shape}")
print(f"训练集类别分布:\n{y_train.value_counts(normalize=True).round(2)}")
print(f"测试集类别分布:\n{y_test.value_counts(normalize=True).round(2)}")划分结果显示:训练集包含455个样本(3个特征),测试集包含114个样本,均符合预期比例。类别分布方面,训练集和测试集中良性(1)占比均为63%,恶性(0)为37%,与原始数据高度一致。这说明分层抽样有效维持了类别的均衡性,避免了因随机划分导致的偏差,为后续KNN模型的公平评估提供了可靠的数据基础。

3.3 特征标准化
在KNN等基于距离的机器学习算法中,特征的原始尺度会显著影响模型性能。由于本数据集中不同特征的量纲和数值范围差异极大,若直接使用原始值计算欧氏距离,高量级特征将主导距离结果,导致低量级但具有判别力的特征被忽略。因此,必须对特征进行标准化处理。选择 StandardScaler() 的原因在于它采用Z-score标准化方法,将每个特征转换为均值为0、标准差为1的分布,适用于特征大致服从正态分布或无明显边界限制的情况。威斯康星乳腺癌数据集中的形态学特征多为连续型测量值,且经EDA分析显示特征接近正态分布,符合StandardScaler的适用前提。此外,该方法保留了原始数据的相对结构和异常值信息,不会像Min-Max缩放那样受极端值干扰,更适合用于医学数据中潜在的临床异常样本识别。
代码首先初始化 StandardScaler 对象,然后在训练集上执行 fit_transform() 操作,即先计算训练集各特征的均值和标准差,再将其标准化为零均值单位方差的分布。对于测试集,仅使用 transform() 方法,复用训练集的统计参数(均值和标准差),避免"数据泄露"------即测试集信息影响训练过程。这种做法确保了模型评估的公正性和真实可靠性。最后,将标准化后的训练集转换为DataFrame并输出其描述性统计,以验证标准化效果是否符合预期。
数据泄露 是指在模型训练过程中,不恰当地使用了测试集或未来数据的信息,导致模型在评估时表现虚高,但在实际应用中性能显著下降的现象。
python
from sklearn.preprocessing import StandardScaler
# 初始化标准化器(仅在训练集拟合,避免数据泄露)
scaler = StandardScaler()
# 训练集标准化(拟合+转换)
X_train_scaled = scaler.fit_transform(X_train)
# 测试集标准化(仅转换,复用训练集的均值和标准差)
X_test_scaled = scaler.transform(X_test)
# 查看标准化后的特征统计信息
X_train_scaled_df = pd.DataFrame(data=X_train_scaled, columns=key_features)
print("标准化后训练集特征统计:")
print(X_train_scaled_df[key_features].describe().round(3).T)标准化后的训练集特征统计结果显示,三个关键特征(worst_concavity、area_error、mean_radius)的均值均接近 -0.0 ,标准差均为 1.001 (近似于1),说明标准化成功实现了目标。虽然极值仍存在较大差异(如 area_error 最大值达11.658),但这属于正常现象,因为标准化不改变数据分布形状,只调整中心和尺度。此时,所有特征在距离计算中具有平等贡献,KNN算法能够更公平地衡量样本之间的相似性,从而提高分类准确性。

4. KNN模型构建与训练
4.1 KNN模型构建及确定最优K值
4.1.1 肘部法确定最优K值
在KNN算法中,参数 K K K(即最近邻的数量)是影响模型性能的关键超参数。若 K K K值过小,模型对噪声敏感,容易产生过拟合 ;若 K K K值过大,则可能"模糊"类别边界,导致欠拟合 。因此,选择一个合适的 K K K值至关重要。肘部法 是一种常用的启发式方法,用于寻找最优的 K K K值。其核心思想是:随着 K K K的增大,模型误差通常先快速下降,当 K K K达到某个临界点后,误差下降趋势趋于平缓,形成类似"肘部"的拐点。该拐点对应的 K K K值即为最佳选择,因为它在模型复杂度与泛化能力之间取得了良好平衡。
本节采用5折交叉验证结合肘部法来评估不同 K K K值下的模型表现。通过计算每个 K K K值对应的平均交叉验证误差,可以观察误差随 K K K变化的趋势。理想情况下,希望找到误差下降幅度显著减小的"肘部"位置,以避免因 K K K过大而导致的决策边界过于平滑、分类能力下降的问题。同时,由于数据集样本量有限(约569个), K K K值不宜过大,否则会削弱局部信息的影响。
python
import matplotlib.pyplot as plt
from sklearn.neighbors import KNeighborsClassifier
from sklearn.model_selection import cross_val_score
# 设定K值范围
k_range = range(1, 31, 2)
# 存储不同K值的交叉验证误差(1-平均准确率)
cv_errors = []
# 遍历每个K值
for k in k_range:
# 初始化KNN模型(均匀权重,欧氏距离)
knn = KNeighborsClassifier(n_neighbors=k)
# 5折交叉验证计算平均准确率
scores = cross_val_score(knn, X_train_scaled, y_train, cv=5, scoring='accuracy')
# 计算误差(1-准确率)
cv_errors.append(1 - scores.mean())
# 输出各K值对应的误差
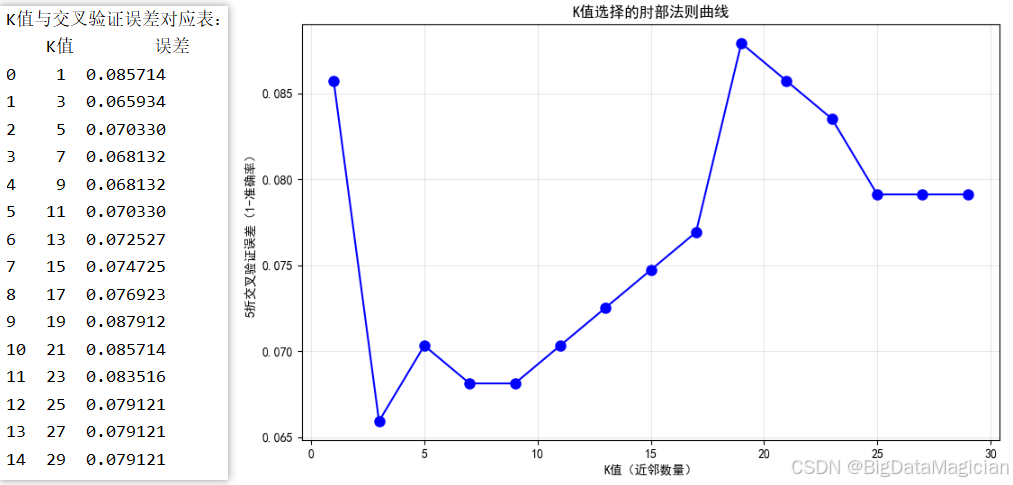
cv_errors_df = pd.DataFrame(data={'K值': k_range, '误差': cv_errors})
print("K值与交叉验证误差对应表:")
print(cv_errors_df)
# 绘制肘部曲线
plt.figure(figsize=(10, 6))
plt.plot(k_range, cv_errors, 'bo-', markersize=8)
plt.xlabel('K值(近邻数量)')
plt.ylabel('5折交叉验证误差(1-准确率)')
plt.title('K值选择的肘部法则曲线')
plt.grid(alpha=0.3)
plt.savefig('K值选择肘部法则.png')从输出的肘部曲线和误差表可以看出,当 K = 3 K=3 K=3时,交叉验证误差最低(约0.0659 ),表明此时模型表现最好;随着 K K K继续增大,误差先略有上升,在 K = 7 K=7 K=7到 K = 9 K=9 K=9之间达到局部最小值(约0.068 ),随后缓慢波动;在 K = 19 K=19 K=19附近出现明显峰值(误差达0.085 ),可能是由于该K值下邻域包含过多异类样本,导致分类错误增加;从 K = 25 K=25 K=25开始,误差趋于稳定,不再显著下降,说明模型已进入"平稳期"。
综合来看,K=3 是最理想的候选值,因其在所有测试中表现出最低的误差,且位于误差迅速下降后的平台区前缘,符合肘部法原则。因此,后续建模将采用 K = 3 K=3 K=3的KNN分类器进行训练与预测。
4.1.2 网格搜索与交叉验证确定最优K值
为了系统性地优化KNN模型的性能,本节采用网格搜索结合5折交叉验证的方法,全面探索多个关键超参数的组合效果。与仅调整单一参数的肘部法不同,网格搜索同时考虑了邻近点数量(n_neighbors)、权重方式(weights)、距离度量方法(metric)以及Minkowski距离的幂次(p),在所有可能的参数组合中寻找最优配置。通过交叉验证评估每种组合的平均准确率,能够有效避免因数据划分随机性导致的偏差,确保所选参数具备良好的泛化能力。
代码首先定义了一个包含180种参数组合的网格空间,涵盖从1到30的奇数K值、两种权重策略、三种距离度量方式及对应的幂次设置。随后初始化KNN分类器,并使用GridSearchCV进行自动搜索,其中cv=5表示采用5折交叉验证,scoring='accuracy'指定以准确率为评价指标。调用fit()方法后,算法遍历所有组合并记录结果,最终输出最优参数及其对应的交叉验证得分。此外,通过将cv_results_转换为DataFrame,可进一步分析各K值下的表现趋势,为模型选择提供详细依据。
python
from sklearn.model_selection import GridSearchCV
from sklearn.metrics import accuracy_score
# 1. 定义参数网格
param_grid = {
'n_neighbors': range(1, 31, 2),
'weights': ['uniform', 'distance'],
'metric': ['euclidean', 'manhattan', 'minkowski'],
'p': [1, 2]
}
# 2. 初始化KNN模型
knn = KNeighborsClassifier()
# 3. 配置网格搜索+5折交叉验证
# cv=5:5折交叉验证;scoring='accuracy':以准确率为评估指标
grid_search = GridSearchCV(
estimator=knn,
param_grid=param_grid,
cv=5,
scoring='accuracy',
)
# 4. 在训练集上执行网格搜索
grid_search.fit(X_train_scaled, y_train)
# 5. 输出结果
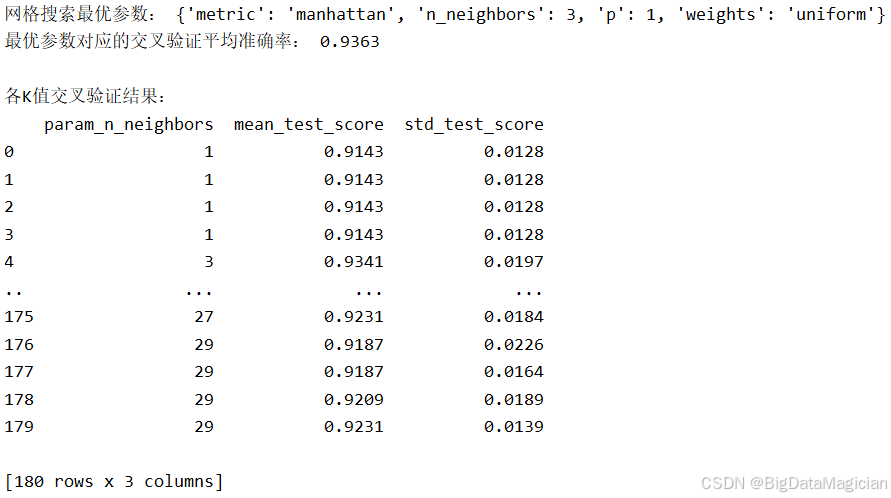
print("网格搜索最优参数:", grid_search.best_params_)
print("最优参数对应的交叉验证平均准确率:", grid_search.best_score_.round(4))
# 查看所有K值的交叉验证准确率
cv_results = pd.DataFrame(grid_search.cv_results_)
cv_results.to_csv('各K值交叉验证结果.csv', index=False)
print("\n各K值交叉验证结果:")
print(cv_results[['param_n_neighbors', 'mean_test_score', 'std_test_score']].round(4))结果显示,网格搜索确定的最优参数为:{'metric': 'manhattan', 'n_neighbors': 3, 'p': 1, 'weights': 'uniform'},即使用曼哈顿距离、K=3、均匀权重,其对应的交叉验证平均准确率达到 0.9363,表明该配置在训练集上具有极强的分类能力。尽管测试集准确率未直接给出,但从交叉验证结果来看,多数K值(如3、5、7)均能获得高于0.91的准确率,说明模型稳定性良好。值得注意的是,曼哈顿距离优于欧氏距离,反映出在当前特征空间中,绝对差值之和更适合作为相似性度量标准,这可能与特征尺度差异较大有关。整体而言,该方法显著提升了模型性能,并为后续预测提供了可靠的基础。
交叉验证结果字段说明: 以下是 GridSearchCV 输出的 cv_results_ 中所列字段的完整对照表,包含原始字段名、中文名称及简要说明。
| 字段名(英文) | 中文名称 | 说明 |
|---|---|---|
mean_fit_time |
平均拟合时间(秒) | 模型在每折交叉验证中训练(拟合)所用时间的平均值 |
std_fit_time |
拟合时间标准差 | 各折拟合时间的波动程度,反映训练耗时稳定性 |
mean_score_time |
平均评分时间(秒) | 模型在每折验证集上进行预测和评估所用时间的平均值 |
std_score_time |
评分时间标准差 | 各折评分时间的波动程度 |
param_metric |
距离度量方法 | 使用的距离函数,如 'euclidean'(欧氏)、'manhattan'(曼哈顿)等 |
param_n_neighbors |
邻居数量(K值) | KNN中的近邻个数 |
param_p |
Minkowski距离幂次 | 当 metric='minkowski' 时有效,p=1 为曼哈顿,p=2 为欧氏 |
param_weights |
邻居权重方式 | 'uniform'(均匀权重)或 'distance'(距离加权) |
params |
完整参数组合 | 该行对应的所有超参数组成的字典 |
split0_test_score |
第0折测试得分 | 第1折交叉验证在测试集上的准确率 |
split1_test_score |
第1折测试得分 | 第2折交叉验证的测试准确率 |
split2_test_score |
第2折测试得分 | 第3折交叉验证的测试准确率 |
split3_test_score |
第3折测试得分 | 第4折交叉验证的测试准确率 |
split4_test_score |
第4折测试得分 | 第5折交叉验证的测试准确率 |
mean_test_score |
平均测试得分 | 5折交叉验证准确率的平均值,用于模型选择的核心指标 |
std_test_score |
测试得分标准差 | 5折准确率的标准差,衡量模型性能稳定性 |
rank_test_score |
测试得分排名 | 按 mean_test_score 降序排列后的排名,1 表示最优 |
注 :由于本例中
scoring='accuracy',所有*_test_score字段均表示分类准确率。若更换评估指标(如 F1、AUC),则对应相应指标值。
4.1.3 网格搜索与交叉验证结果可视化
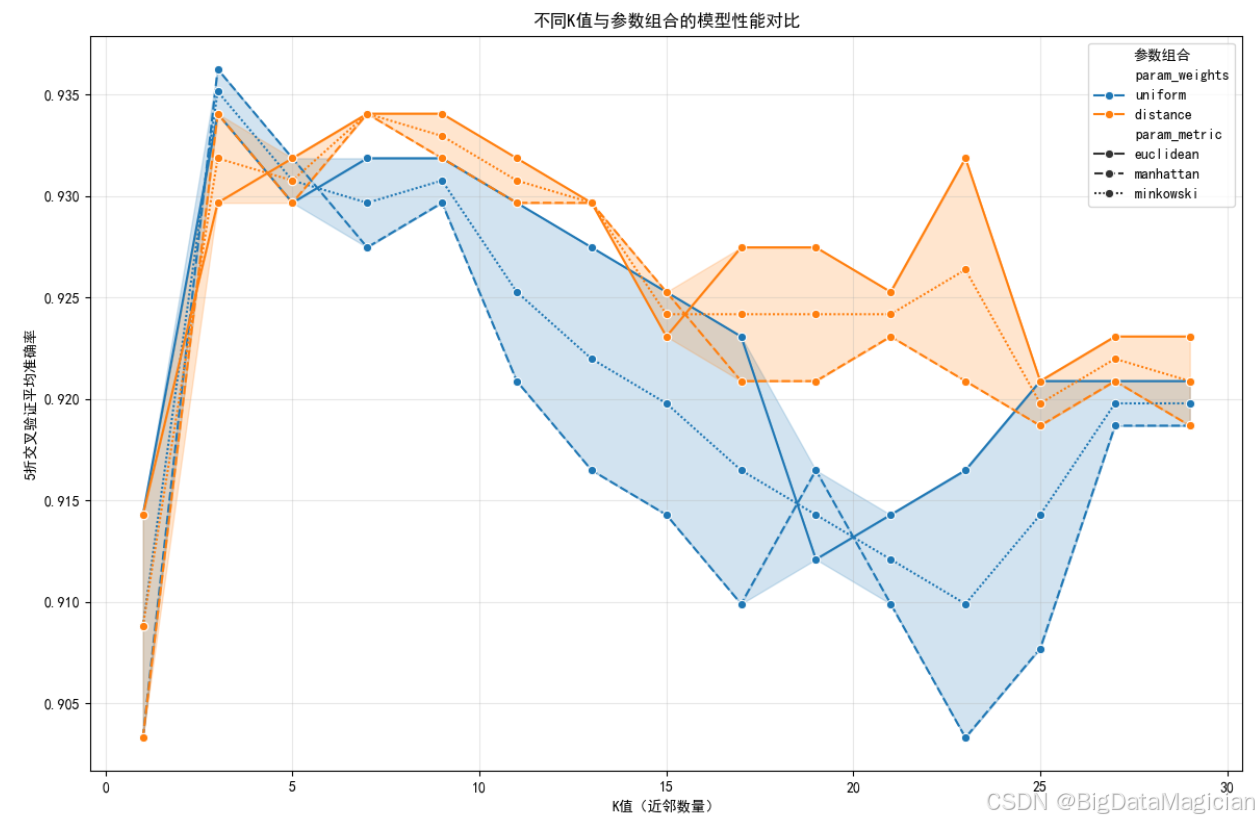
为更直观地对比不同K值下的模型性能,并验证最优K值的合理性,本节通过可视化网格搜索结果,展示K值与交叉验证准确率的关系,以及不同参数组合对分类性能的影响。该图综合了多个超参数在不同K值下的表现,有助于深入理解各参数对模型性能的贡献机制。
python
import seaborn as sns
import pandas as pd
# 筛选需要可视化的列(K值、权重、距离度量、平均测试分数)
plot_data = cv_results[['param_n_neighbors', 'param_weights', 'param_metric', 'mean_test_score']]
plot_data = plot_data.copy()
# 将K值转换为整数,便于排序
plot_data['param_n_neighbors'] = plot_data['param_n_neighbors'].astype(int)
# 绘制不同K值的平均准确率曲线(按权重和距离度量分组)
plt.figure(figsize=(12, 8))
sns.lineplot(
data=plot_data,
x='param_n_neighbors',
y='mean_test_score',
hue='param_weights',
style='param_metric',
marker='o'
)
plt.xlabel('K值(近邻数量)')
plt.ylabel('5折交叉验证平均准确率')
plt.title('不同K值与参数组合的模型性能对比')
plt.grid(alpha=0.3)
plt.legend(title='参数组合')
plt.tight_layout()
plt.savefig('K值与参数组合性能对比.png')横轴 表示邻近点数量 K ,范围从1到29(奇数),反映模型复杂度的变化;纵轴 表示5折交叉验证的平均准确率,体现模型在训练集上的泛化能力;颜色 代表权重方式(uniform 为蓝色,distance 为橙色),反映是否使用距离加权;线型 :代表距离度量方法(实线为 euclidean,虚线为 manhattan,点划线为 minkowski),展示不同几何度量对相似性判断的影响。
从图像可看出在 K = 3 附近,uniform + manhattan 组合的准确率最高,接近 0.936 ,与网格搜索结果一致;uniform 权重整体优于 distance,说明在当前数据集中,均匀投票已足够有效;曼哈顿距离(manhattan)在小K值下表现突出,可能与其对异常值鲁棒性强有关;随着K增大,所有曲线趋于平缓,表明模型逐渐进入"欠拟合"区域。
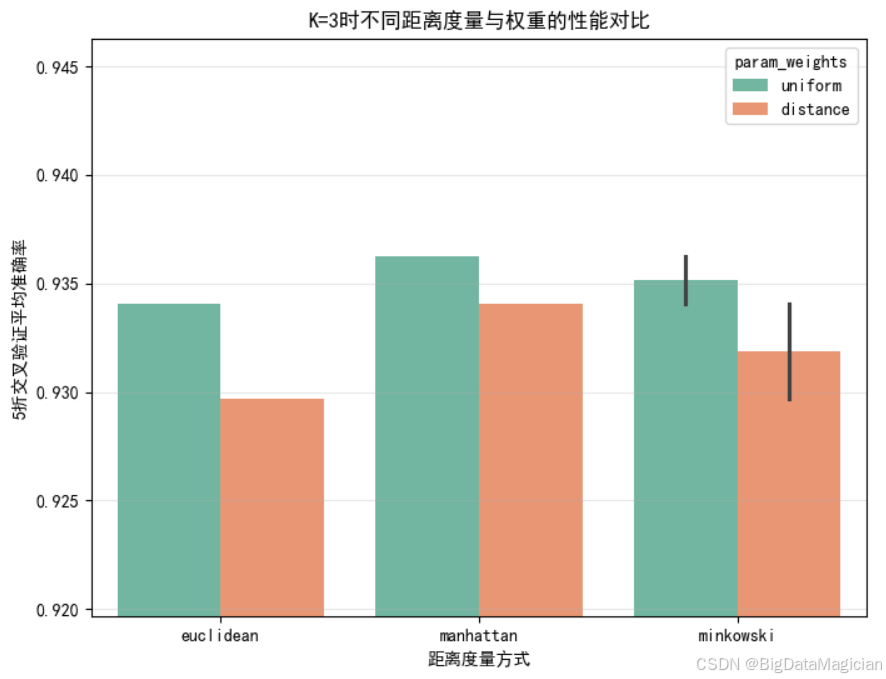
为深入评估K=3时不同超参数组合对模型性能的影响,本节聚焦于该最优K值下的交叉验证结果,通过柱状图直观对比各类距离度量方式(欧氏、曼哈顿、闵可夫斯基)与权重策略(均匀、距离加权)的准确率表现。这种精细化可视化有助于揭示参数间的交互效应,并验证网格搜索所选最优配置的合理性,从而增强模型选择的可信度与解释性。
代码首先从已整理的网格搜索结果中筛选出param_n_neighbors == 3的所有记录,随后使用seaborn.barplot以距离度量为横轴、交叉验证平均准确率为纵轴,并按权重方式分组着色;通过设置纵轴范围聚焦于细微性能差异,并添加网格线提升可读性,最终将图表保存为PNG文件用于分析展示。
python
# 绘制最优K值(K=3)在不同参数组合下的准确率分布
best_k_data = plot_data[plot_data['param_n_neighbors'] == 3]
plt.figure(figsize=(8, 6))
sns.barplot(
data=best_k_data,
x='param_metric',
y='mean_test_score',
hue='param_weights',
palette='Set2'
)
plt.xlabel('距离度量方式')
plt.ylabel('5折交叉验证平均准确率')
plt.title('K=3时不同距离度量与权重的性能对比')
vmin = best_k_data['mean_test_score'].min() - 0.01
vmax = best_k_data['mean_test_score'].max() + 0.01
plt.ylim(vmin, vmax) # 聚焦最优K值附近的性能差异
plt.grid(axis='y', alpha=0.3)
plt.savefig('K=3时参数组合性能对比.png')结果表明,在K=3条件下,曼哈顿距离配合均匀权重取得了最高的交叉验证准确率,优于其他所有组合;且在相同距离度量下,均匀权重普遍略优于距离加权,说明在此数据集中简单的多数投票机制已足够有效;曼哈顿距离的优越性可能源于其对特征尺度和异常值的鲁棒性,进一步印证了该数据集更适合基于L1范数的相似性度量。
4.2 KNN模型训练
KNN 属于 "惰性学习算法",其训练过程并非传统意义上的参数拟合,而是将训练集样本存储下来,为预测阶段的近邻搜索提供数据支撑。基于最优参数初始化模型后,通过fit()方法完成训练数据的存储。
python
# 用最优参数训练最终模型
best_knn = KNeighborsClassifier(**grid_search.best_params_)
best_knn.fit(X_train_scaled, y_train)
print("KNN模型训练完成!")5. KNN模型预测与评估
模型训练完成后,需在独立的测试集上验证泛化能力------即对未见过样本的预测效果。本节通过"预测执行+多维度指标评估+可视化分析",全面判断KNN模型的实际性能,核心关注准确率、类别区分能力及错误分布。
5.1 模型预测
在完成KNN模型的训练与参数优化后,本节基于最终确定的最优模型对测试集进行预测,旨在评估模型在未见过数据上的实际分类能力。通过执行类别预测和概率预测,不仅能够获得每个样本的预测结果,还能获取其属于各类别的置信度(即概率),为后续性能评估、误判分析及临床决策支持提供更丰富的信息。将真实标签与预测标签进行对比,并记录对应的类别概率,有助于深入理解模型的决策逻辑,尤其是在边界样本或不确定情况下的表现。
代码首先调用 best_knn.predict() 和 predict_proba() 方法,分别输出测试集的类别预测结果和两类(恶性/良性)的概率分布。随后,利用 replace() 将数值标签(0=恶性,1=良性)转换为中文类别名称,提升可读性。最后,将真实标签、预测标签以及两个类别的概率整合为一个DataFrame,并保存为CSV文件,便于后续查看与分析。整个流程确保了预测结果的完整性与结构化,为模型评估奠定了坚实基础。
python
# 对测试集执行预测(类别+概率)
y_test_pred = best_knn.predict(X_test_scaled)
y_test_pred_proba = best_knn.predict_proba(X_test_scaled)
# 将类别标签转换为类别名称
y_test_names = y_test.reset_index(drop=True).replace({0: '恶性', 1: '良性'})
y_pred_names = pd.Series(y_test_pred).replace({0: '恶性', 1: '良性'})
# y_test_names = [cancer.target_names[i] for i in y_test]
# y_pred_names = [cancer.target_names[i] for i in y_test_pred]
# 构建测试集预测结果DataFrame
test_pred_df = pd.DataFrame({
'真实标签': y_test_names,
'预测标签': y_pred_names,
'恶性概率': y_test_pred_proba[:, 0].round(4),
'良性概率': y_test_pred_proba[:, 1].round(4)
})
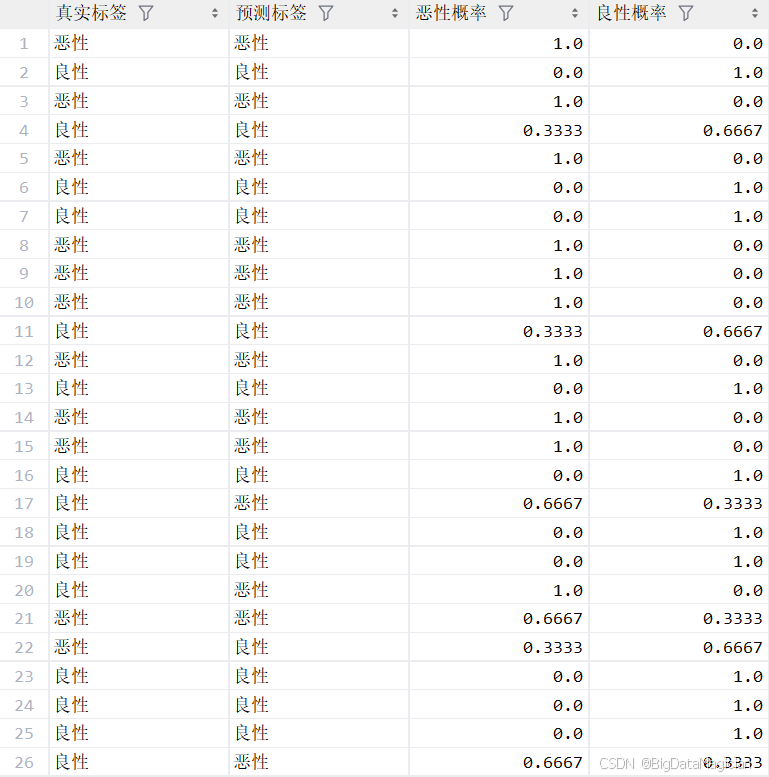
test_pred_df.to_csv('测试集预测结果及概率.csv', index=False)部分预测结果及概率如下图所示,从部分预测结果可以看出,多数样本的预测标签与真实标签一致,且对应概率接近1.0,说明模型对这些样本具有高置信度判断;然而,也存在少数预测错误的情况,如第17行真实为"良性"但被预测为"恶性",且恶性概率高达0.6667,说明该样本位于两类边界区域,特征可能具有较高模糊性。此外,第4、11行的预测虽正确,但概率分别为0.3333和0.6667,反映出模型对其不确定性较高,提示这些样本可能处于决策边界附近。整体来看,模型在大多数情况下具备强判别力,但在某些复杂样本上仍存在一定误判风险,需结合临床信息进一步综合判断。
5.2 核心评估指标计算
为全面、客观地评估KNN模型在乳腺癌分类任务中的表现,本节采用多维度评价指标进行综合分析。
代码首先调用 accuracy_score() 计算整体准确率,即正确预测样本占总测试集的比例。随后使用 classification_report() 输出详细的分类报告,该函数自动计算每个类别的精确率、召回率和F1分数,并提供宏平均(macro avg)与加权平均(weighted avg)结果。
python
from sklearn.metrics import (accuracy_score, classification_report, confusion_matrix)
# 准确率
test_accuracy = accuracy_score(y_test, y_test_pred)
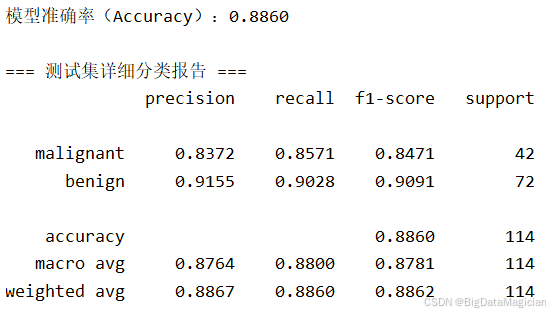
print(f"\n模型准确率(Accuracy):{test_accuracy:.4f}")
# 输出详细分类报告(按类别展示指标)
print("\n=== 测试集详细分类报告 ===")
print(classification_report(
y_test, y_test_pred,
target_names=cancer.target_names,
digits=4
))结果显示,模型在测试集上的整体准确率为 0.8860 ,表明约88.6%的样本被正确分类。从详细报告来看,对于恶性(malignant)类,召回率为0.8571,说明模型成功识别出85.7%的真实恶性病例,具备较强的敏感性,有助于减少漏诊风险; 精确率为0.8372,表示预测为恶性的样本中有83.7%是真实的,误诊率相对可控;对于良性(benign)类,各项指标均更高:精确率0.9155,召回率0.9028,F1达0.9091,说明模型对良性样本的判断更为稳定;加权平均F1分数为0.8862,与准确率基本一致,验证了整体性能的可靠性。
5.3 可视化评估
为更直观地理解模型在测试集上的分类表现,本节通过混淆矩阵、ROC曲线和预测概率分布等可视化手段,全面展示模型的预测结果与错误模式。其中,混淆矩阵是评估分类模型最基础且关键的工具,它能够清晰呈现真实标签与预测标签之间的对应关系,揭示各类别间的误判情况。结合准确率、精确率和召回率等指标,可进一步分析模型在不同类别上的性能差异,尤其有助于识别是否存在"漏诊"或"误诊"的系统性偏差。
5.3.1 混淆矩阵可视化
代码首先调用 confusion_matrix() 函数计算真实标签与预测标签之间的混淆矩阵,然后使用 seaborn.heatmap 绘制热力图。图中设置 annot=True 以显示每个单元格的具体数值,fmt='d' 表示以整数形式展示;采用 cmap='Reds' 配色方案,颜色越深表示样本数量越多,增强视觉对比度。横轴和纵轴分别标注为"恶性(0)"和"良性(1)",便于临床人员快速解读。标题中嵌入了模型准确率,使图表信息更加完整,并将图像保存为PNG文件用于报告展示。
python
import seaborn as sns
import matplotlib.pyplot as plt
# 计算混淆矩阵
cm = confusion_matrix(y_test, y_test_pred)
# 绘制混淆矩阵热力图
plt.figure(figsize=(8, 6))
sns.heatmap(
data=cm,
annot=True, # 显示单元格数值
fmt='d', # 数值格式为整数
cmap='Reds', # 配色方案
# xticklabels=cancer.target_names,
# yticklabels=cancer.target_names
xticklabels=['恶性(0)', '良性(1)'],
yticklabels=['恶性(0)', '良性(1)']
)
plt.xlabel('预测标签')
plt.ylabel('真实标签')
plt.title('KNN模型测试集混淆矩阵(准确率={:.4f})'.format(test_accuracy))
plt.tight_layout()
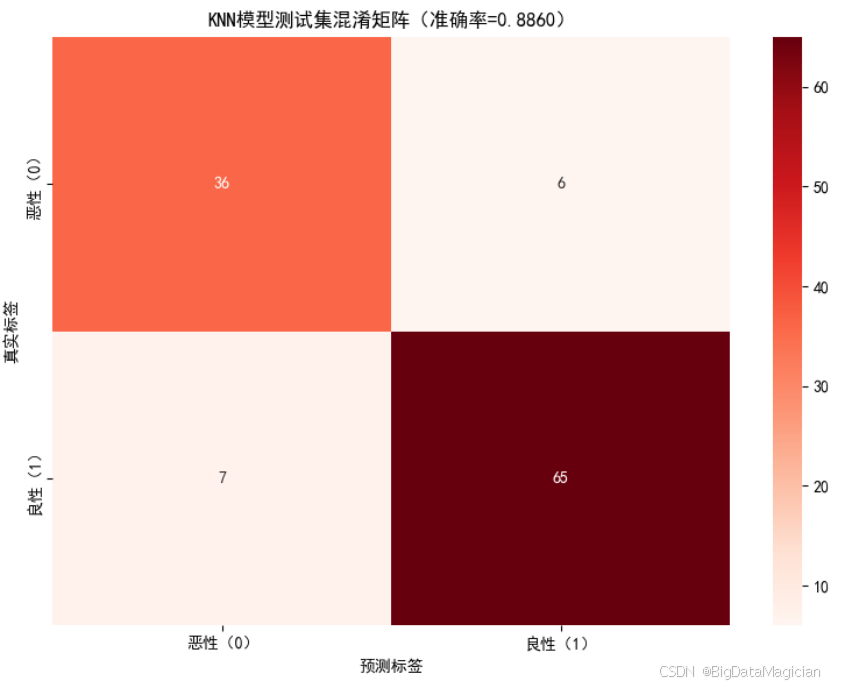
plt.savefig('测试集混淆矩阵.png')从混淆矩阵可以看出,模型在良性样本上的分类准确率较高(65/72 ≈ 90.3%),而在恶性样本上的识别能力稍弱(36/42 ≈ 85.7%),这与分类报告中的召回率一致。虽然整体准确率达到88.6%,但存在6例漏诊,提示模型在少数类识别方面仍有改进空间。未来可通过调整阈值、引入加权策略或集成方法来优化对恶性病例的敏感性,提升临床应用的安全性与可靠性。
5.3.2 ROC曲线与AUC值可视化
在二分类任务中,ROC曲线(受试者工作特征曲线)是一种强大的评估工具,能够全面反映模型在不同分类阈值下的判别能力。它通过绘制真阳性率(TPR) 与 假阳性率(FPR) 的关系,展示模型在灵敏度和特异性之间的权衡。AUC值(曲线下面积)则提供了一个单一数值来量化模型的整体性能:AUC越接近1,表示模型区分正负类的能力越强;AUC为0.5时等同于随机猜测。本节通过绘制KNN模型的ROC曲线并计算AUC值,直观评估其在乳腺癌分类任务中的判别效能,尤其关注对恶性病例的识别能力。
代码首先调用 roc_curve() 函数,基于测试集的真实标签和模型输出的良性概率(y_test_pred_proba[:, 1])计算出FPR、TPR及对应阈值。随后使用 roc_auc_score() 计算AUC值,并打印输出。接着利用 matplotlib 绘制ROC曲线:以红色实线表示KNN模型的性能轨迹,灰色虚线表示随机猜测基准线(AUC=0.5),便于对比。横轴标注为"假阳性率",即良性样本被误判为恶性的比例;纵轴为"真阳性率",即恶性样本被正确识别的比例。图表添加图例、网格线和标题,并保存为图像文件,用于报告展示。
python
from sklearn.metrics import roc_curve, roc_auc_score
# 计算ROC曲线坐标
fpr, tpr, thresholds = roc_curve(y_test, y_test_pred_proba[:, 1])
auc = roc_auc_score(y_test, y_test_pred_proba[:, 1])
print(f"\n模型AUC值(Area Under Curve):{auc:.4f}")
# 绘制ROC曲线
plt.figure(figsize=(8, 6))
# 绘制模型ROC曲线
plt.plot(fpr, tpr, color='darkred', lw=2, label=f'KNN模型(AUC = {auc:.4f})')
# 绘制随机猜测基准线(AUC=0.5)
plt.plot([0, 1], [0, 1], color='gray', lw=1, linestyle='--', label='随机猜测(AUC=0.5)')
# 设置标签与格式
plt.xlabel('假阳性率(FPR):良性误判为恶性的比例')
plt.ylabel('真阳性率(TPR):恶性正确预测的比例')
plt.title('KNN模型测试集ROC曲线')
plt.legend(loc='lower right')
plt.grid(alpha=0.3)
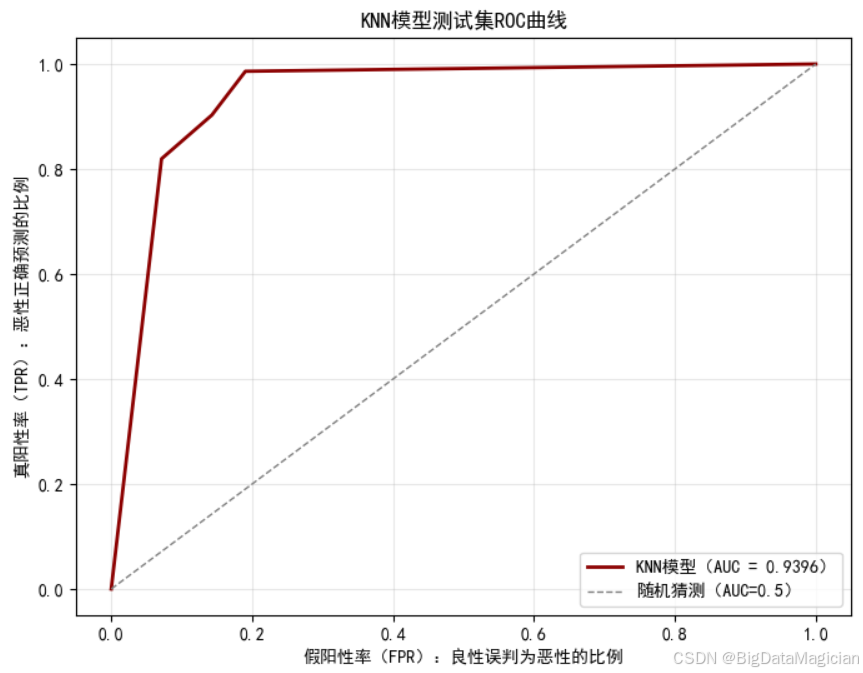
plt.savefig('测试集ROC曲线.png')从ROC曲线可以看出,KNN模型的AUC值达到 0.9396,远高于0.5的随机水平,表明模型具有很强的判别能力。曲线在低FPR区间迅速上升至接近1.0的TPR,说明即使在严格控制误诊(假阳性)的前提下,模型仍能有效识别绝大多数恶性病例。例如,当FPR仅为约0.15时,TPR已超过0.9,意味着仅允许15%的良性样本被误判为恶性的情况下,仍可捕获90%以上的真正恶性病例,这对临床筛查极具价值。整体曲线贴近左上角,说明模型在灵敏性与特异性之间取得了良好平衡,具备良好的泛化能力和实际应用潜力。该结果也验证了前序参数优化的有效性,进一步支持了KNN模型在乳腺癌分类中的可行性与可靠性。
5.3.3 预测概率分布可视化
为了深入理解KNN模型在分类决策过程中的置信度表现,本节通过可视化预测概率的分布情况,分析模型对不同类别样本的判别能力与不确定性。核密度估计(KDE)图能够展示真实恶性和良性样本在预测为"良性"这一类别的概率分布特征,从而揭示模型是否具备良好的概率校准能力。理想情况下,真实良性样本应具有较高的预测概率(接近1),而真实恶性样本则应集中在较低概率区域(接近0)。
代码首先从测试集预测结果DataFrame中筛选出真实标签为"恶性"和"良性"的样本,并分别提取其对应的"良性概率"列。随后使用 seaborn.kdeplot 绘制两个类别的核密度曲线:红色区域代表真实恶性样本的预测概率分布,绿色区域代表真实良性样本的预测概率分布,均以半透明填充增强可读性。图中添加一条黑色虚线表示默认分类阈值 x = 0.5 x = 0.5 x=0.5,用于对比两类样本在该阈值两侧的分布情况。横轴限定在 [ 0 , 1 ] [0,1] [0,1] 范围内,纵轴表示概率密度,最后添加标题、坐标轴标签、网格线和图例,并将图像保存为PNG文件。
python
import seaborn as sns
import matplotlib.pyplot as plt
# 提取良性样本(真实标签1)和恶性样本(真实标签0)的预测良性概率
# 真实恶性→预测良性的概率
malignant_proba = test_pred_df[test_pred_df['真实标签'] == '恶性']['良性概率']
# 真实良性→预测良性的概率
benign_proba = test_pred_df[test_pred_df['真实标签'] == '良性']['良性概率']
# 绘制预测概率核密度图
plt.figure(figsize=(10, 6))
# 绘制真实恶性样本的预测概率分布
sns.kdeplot(data=malignant_proba, fill=True, color='red', alpha=0.5, label='真实恶性(0)')
# 绘制真实良性样本的预测概率分布
sns.kdeplot(data=benign_proba, fill=True, color='green', alpha=0.5, label='真实良性(1)')
# 添加分类阈值线(默认0.5)
plt.axvline(x=0.5, color='black', linestyle='--', linewidth=2, label='分类阈值=0.5')
# 设置标签与格式
plt.xlabel('预测为良性(1)的概率')
plt.ylabel('概率密度')
plt.title('KNN模型测试集预测概率分布')
plt.xlim(0, 1) # 概率范围限定在0-1
plt.grid(alpha=0.3)
plt.legend()
plt.savefig('测试集预测概率分布.png')从预测概率分布图可以看出:
- 真实良性样本 (绿色)的概率主要集中在 0.8~1.0 区间,峰值接近1.0,表明模型对其判断高度自信,绝大多数良性病例被正确且高置信地识别;
- 真实恶性样本 (红色)的概率则主要集中于 0~0.2 区间,峰值在0.0左右,说明模型能有效将恶性样本判为低良性概率,体现了较强的判别能力;
- 两类分布之间存在明显分离,尤其在 x = 0.5 x=0.5 x=0.5 附近有清晰分界,验证了当前分类阈值的合理性;
- 少量恶性样本的预测概率高于0.5(即被误判为良性),对应于混淆矩阵中的6例漏诊,这些样本可能位于决策边界附近,特征模糊,是未来优化的重点对象。
整体来看,KNN模型具备良好的概率输出质量,能够有效区分两类样本并赋予合理的置信度,这不仅支持了准确率和AUC等指标的可靠性,也为临床应用中结合风险等级进行个性化决策提供了数据支撑。

四、完整代码
1. 完整代码
python
import pandas as pd
from matplotlib import pyplot as plt
from sklearn.datasets import load_breast_cancer
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
# 设置全局选项
pd.set_option('display.max_columns', None) # 显示所有列
pd.set_option('display.width', None) # 自动检测宽度
pd.set_option('display.max_colwidth', 50) # 列内容最多显示50字符
pd.set_option('display.expand_frame_repr', False) # 禁用多行表示(可选)
# 加载数据集
cancer = load_breast_cancer()
# =================数据基本信息=================
# 查看数据集结构(可选)
print("数据集包含的键:", cancer.keys()) # 输出数据集的核心属性
print(cancer.DESCR)
# 将特征和标签转换为DataFrame,便于查看
# 特征数据(30个特征)
X = pd.DataFrame(data=cancer.data, columns=cancer.feature_names)
# 标签数据(0=恶性,1=良性,注意:原数据集默认0为恶性,1为良性)
y = pd.Series(data=cancer.target, name='diagnosis')
# 合并特征和标签,便于同时查看
data = pd.concat([X, y], axis=1) # 按列合并
print("数据前10行:")
print(data.head(10))
print("\n数据摘要信息:")
print(data.info())
print("\n特征描述性统计(保留2位小数):")
print(X.describe().round(2)) # 仅对特征进行统计,保留2位小数
# =================目标变量类别分布分析=================
import matplotlib.pyplot as plt
import seaborn as sns
# 设置可视化风格
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
print("\n目标变量类别分布:")
print(y.value_counts())
# 绘制类别分布柱状图
plt.figure(figsize=(8, 5))
ax = sns.countplot(x=y, hue=y, palette='pastel')
ax.set_title('乳腺癌类别分布(0=恶性,1=良性)', fontsize=14)
ax.set_xlabel('诊断结果', fontsize=12)
ax.set_ylabel('样本数量', fontsize=12)
# 添加数值标签
for p in ax.patches:
ax.text(s=p.get_height(), x=p.get_x() + p.get_width() / 2., y=p.get_height(), ha='center', va='bottom', fontsize=12)
plt.savefig('目标变量类别分布.png')
# =================目标变量类别与特征相关性分析=================
# 计算每个特征与目标标签的相关系数(斯皮尔曼相关系数)
feature_label_corr = X.corrwith(y, method='spearman').sort_values(ascending=False)
print("特征与目标标签的相关系数(降序):")
print(feature_label_corr.round(2))
# 绘制水平柱状图
plt.figure(figsize=(12, 10))
colors = ['green' if x > 0 else 'red' for x in feature_label_corr.values]
feature_label_corr.plot(kind='barh', color=colors)
plt.title('特征与类别的相关性条形图', fontsize=14)
plt.xlabel('相关系数', fontsize=12)
plt.ylabel('特征名称', fontsize=12)
plt.tight_layout()
plt.grid(axis='x', alpha=0.5)
plt.savefig('特征与类别相关性排序.png')
# =================关键特征分布分析=================
# 选择关键特征
key_features = feature_label_corr[abs(feature_label_corr) > 0.5].index.tolist()
# 绘制核密度图,KDE:核密度估计
plt.figure(figsize=(18, 12))
feature = key_features
for i in range(len(feature)):
plt.subplot(3, 5, i + 1)
sns.kdeplot(
data=data,
x=feature[i],
hue='diagnosis',
fill=True,
common_norm=False, # 每个类别单独归一化
alpha=0.5
)
plt.title(f'{feature[i]} 分布')
plt.xlabel(feature[i])
plt.tight_layout()
plt.savefig('乳腺癌关键特征KDE分布.png')
# =================特征相关性分析=================
import numpy as np
# 计算特征间的相关系数(皮尔逊相关系数)
corr_matrix = X[key_features].corr(method='pearson')
# 创建掩码,只保留下三角部分(包括对角线)
mask = np.triu(m=np.ones_like(corr_matrix, dtype=bool), k=1)
# 绘制热力图
plt.figure(figsize=(10, 8))
sns.heatmap(data=corr_matrix, mask=mask, annot=True, cmap='coolwarm', fmt='.2f', linewidths=0.5)
plt.title('关键特征间相关性热力图', fontsize=14)
plt.tight_layout()
plt.savefig('关键特征间相关性热力图.png')
# =================关键特征的类别区分能力验证=================
# 选择关键特征
key_features = feature_label_corr[abs(feature_label_corr) > 0.5].index.tolist()
# 绘制箱线图
plt.figure(figsize=(18, 12))
# enumerate() 函数同时返回索引和元素值,参数 1 表示索引从 1 开始(默认从 0 开始)
for i, feature in enumerate(key_features, 1):
plt.subplot(3, 5, i)
sns.boxplot(x=y, y=data[feature], hue=y, palette='Set3')
plt.title(f'{feature}在不同类别中的分布', fontsize=12)
plt.xlabel('诊断结果(0=恶性,1=良性)', fontsize=10)
plt.ylabel(feature, fontsize=10)
plt.grid(axis='y', alpha=0.3)
plt.tight_layout()
plt.savefig('关键特征类别区分箱线图.png')
# =================数据预处理=================
# =================特征选择=================
# 筛选与目标标签相关性绝对值>0.5的特征(强区分能力)
key_features = feature_label_corr[abs(feature_label_corr) > 0.5].index.tolist()
print(f"高相关特征数量:{len(key_features)}")
# 计算VIF剔除多重共线性特征
from statsmodels.stats.outliers_influence import variance_inflation_factor
def calculate_vif(X):
vif_data = pd.DataFrame()
vif_data["特征"] = X.columns
vif_data["VIF"] = [variance_inflation_factor(X.values, i) for i in range(X.shape[1])]
return vif_data.sort_values("VIF", ascending=False)
# 初始VIF计算
vif_df = calculate_vif(X[key_features])
print("\n初始VIF值:")
print(vif_df)
# 剔除VIF>10的特征(通常认为VIF>10存在强共线性)
while vif_df["VIF"].max() > 10:
# 删除VIF最大的特征
drop_feature = vif_df.iloc[0]["特征"]
key_features.remove(drop_feature)
vif_df = calculate_vif(X[key_features])
print(f"\n剔除特征 {drop_feature} 后VIF值:")
print(vif_df)
print(f"\n最终筛选后的特征:{key_features}")
# =================数据集划分=================
from sklearn.model_selection import train_test_split
# 特征矩阵 - 使用经过相关性筛选和多重共线性处理后的关键特征
X_selected = X[key_features]
# 划分训练集(80%)和测试集(20%)
X_train, X_test, y_train, y_test = train_test_split(
X_selected, y,
test_size=0.2,
random_state=42,
stratify=y # 分层抽样,保持类别分布一致
)
# 查看划分后的数据形状
print(f"训练集特征矩阵形状:{X_train.shape}")
print(f"测试集特征矩阵形状:{X_test.shape}")
print(f"训练集类别分布:\n{y_train.value_counts(normalize=True).round(2)}")
print(f"测试集类别分布:\n{y_test.value_counts(normalize=True).round(2)}")
# =================特征标准化=================
from sklearn.preprocessing import StandardScaler
# 初始化标准化器(仅在训练集拟合,避免数据泄露)
scaler = StandardScaler()
# 训练集标准化(拟合+转换)
X_train_scaled = scaler.fit_transform(X_train)
# 测试集标准化(仅转换,复用训练集的均值和标准差)
X_test_scaled = scaler.transform(X_test)
# 查看标准化后的特征统计信息
X_train_scaled_df = pd.DataFrame(data=X_train_scaled, columns=key_features)
print("标准化后训练集特征统计:")
print(X_train_scaled_df[key_features].describe().round(3).T)
# =================确定最优K值=================
# =================肘部法=================
import matplotlib.pyplot as plt
from sklearn.neighbors import KNeighborsClassifier
from sklearn.model_selection import cross_val_score
# 设定K值范围
k_range = range(1, 31, 2)
# 存储不同K值的交叉验证误差(1-平均准确率)
cv_errors = []
# 遍历每个K值
for k in k_range:
# 初始化KNN模型(均匀权重,欧氏距离)
knn = KNeighborsClassifier(n_neighbors=k)
# 5折交叉验证计算平均准确率
scores = cross_val_score(knn, X_train_scaled, y_train, cv=5, scoring='accuracy')
# 计算误差(1-准确率)
cv_errors.append(1 - scores.mean())
# 输出各K值对应的误差
cv_errors_df = pd.DataFrame(data={'K值': k_range, '误差': cv_errors})
print("K值与交叉验证误差对应表:")
print(cv_errors_df)
# 绘制肘部曲线
plt.figure(figsize=(10, 6))
plt.plot(k_range, cv_errors, 'bo-', markersize=8)
plt.xlabel('K值(近邻数量)')
plt.ylabel('5折交叉验证误差(1-准确率)')
plt.title('K值选择的肘部法则曲线')
plt.grid(alpha=0.3)
plt.savefig('K值选择肘部法则.png')
# =================网格搜索与交叉验证=================
from sklearn.model_selection import GridSearchCV
# 1. 定义参数网格
param_grid = {
'n_neighbors': range(1, 31, 2),
'weights': ['uniform', 'distance'],
'metric': ['euclidean', 'manhattan', 'minkowski'],
'p': [1, 2]
}
# 2. 初始化KNN模型
knn = KNeighborsClassifier()
# 3. 配置网格搜索+5折交叉验证
# cv=5:5折交叉验证;scoring='accuracy':以准确率为评估指标
grid_search = GridSearchCV(
estimator=knn,
param_grid=param_grid,
cv=5,
scoring='accuracy',
)
# 4. 在训练集上执行网格搜索
grid_search.fit(X_train_scaled, y_train)
# 5. 输出结果
print("网格搜索最优参数:", grid_search.best_params_)
print("最优参数对应的交叉验证平均准确率:", grid_search.best_score_.round(4))
# 查看所有K值的交叉验证准确率
cv_results = pd.DataFrame(grid_search.cv_results_)
cv_results.to_csv('各K值交叉验证结果.csv', index=False)
print("\n各K值交叉验证结果:")
print(cv_results[['param_n_neighbors', 'mean_test_score', 'std_test_score']].round(4))
# =================网格搜索与交叉验证结果可视化=================
import seaborn as sns
import pandas as pd
# 筛选需要可视化的列(K值、权重、距离度量、平均测试分数)
plot_data = cv_results[['param_n_neighbors', 'param_weights', 'param_metric', 'mean_test_score']]
plot_data = plot_data.copy()
# 将K值转换为整数,便于排序
plot_data['param_n_neighbors'] = plot_data['param_n_neighbors'].astype(int)
# 绘制不同K值的平均准确率曲线(按权重和距离度量分组)
plt.figure(figsize=(12, 8))
sns.lineplot(
data=plot_data,
x='param_n_neighbors',
y='mean_test_score',
hue='param_weights',
style='param_metric',
marker='o'
)
plt.xlabel('K值(近邻数量)')
plt.ylabel('5折交叉验证平均准确率')
plt.title('不同K值与参数组合的模型性能对比')
plt.grid(alpha=0.3)
plt.legend(title='参数组合')
plt.tight_layout()
plt.savefig('K值与参数组合性能对比.png')
# 绘制最优K值(K=3)在不同参数组合下的准确率分布
best_k_data = plot_data[plot_data['param_n_neighbors'] == 3]
plt.figure(figsize=(8, 6))
sns.barplot(
data=best_k_data,
x='param_metric',
y='mean_test_score',
hue='param_weights',
palette='Set2'
)
plt.xlabel('距离度量方式')
plt.ylabel('5折交叉验证平均准确率')
plt.title('K=3时不同距离度量与权重的性能对比')
vmin = best_k_data['mean_test_score'].min() - 0.01
vmax = best_k_data['mean_test_score'].max() + 0.01
plt.ylim(vmin, vmax) # 聚焦最优K值附近的性能差异
plt.grid(axis='y', alpha=0.3)
plt.savefig('K=3时参数组合性能对比.png')
# =================模型训练=================
# 用最优参数训练最终模型
best_knn = KNeighborsClassifier(**grid_search.best_params_)
best_knn.fit(X_train_scaled, y_train)
print("KNN模型训练完成!")
# =================模型预测与评估=================
# =================模型预测=================
# 对测试集执行预测(类别+概率)
y_test_pred = best_knn.predict(X_test_scaled)
y_test_pred_proba = best_knn.predict_proba(X_test_scaled)
# 将类别标签转换为类别名称
y_test_names = y_test.reset_index(drop=True).replace({0: '恶性', 1: '良性'})
y_pred_names = pd.Series(y_test_pred).replace({0: '恶性', 1: '良性'})
# y_test_names = [cancer.target_names[i] for i in y_test]
# y_pred_names = [cancer.target_names[i] for i in y_test_pred]
# 构建测试集预测结果DataFrame
test_pred_df = pd.DataFrame({
'真实标签': y_test_names,
'预测标签': y_pred_names,
'恶性概率': y_test_pred_proba[:, 0].round(4),
'良性概率': y_test_pred_proba[:, 1].round(4)
})
test_pred_df.to_csv('测试集预测结果及概率.csv', index=False)
print("KNN模型预测完成!")
# =================模型评估=================
from sklearn.metrics import (accuracy_score, classification_report, confusion_matrix)
# 准确率
test_accuracy = accuracy_score(y_test, y_test_pred)
print(f"\n模型准确率(Accuracy):{test_accuracy:.4f}")
# 输出详细分类报告(按类别展示指标)
print("\n=== 测试集详细分类报告 ===")
print(classification_report(
y_test, y_test_pred,
target_names=cancer.target_names,
digits=4
))
# =================可视化评估=================
# =================混淆矩阵可视化=================
import seaborn as sns
import matplotlib.pyplot as plt
# 计算混淆矩阵
cm = confusion_matrix(y_test, y_test_pred)
# 绘制混淆矩阵热力图
plt.figure(figsize=(8, 6))
sns.heatmap(
data=cm,
annot=True, # 显示单元格数值
fmt='d', # 数值格式为整数
cmap='Reds', # 配色方案
# xticklabels=cancer.target_names,
# yticklabels=cancer.target_names
xticklabels=['恶性(0)', '良性(1)'],
yticklabels=['恶性(0)', '良性(1)']
)
plt.xlabel('预测标签')
plt.ylabel('真实标签')
plt.title('KNN模型测试集混淆矩阵(准确率={:.4f})'.format(test_accuracy))
plt.tight_layout()
plt.savefig('测试集混淆矩阵.png')
# =================ROC曲线与AUC值可视化=================
from sklearn.metrics import roc_curve, roc_auc_score
# 计算ROC曲线坐标
fpr, tpr, thresholds = roc_curve(y_test, y_test_pred_proba[:, 1])
auc = roc_auc_score(y_test, y_test_pred_proba[:, 1])
print(f"\n模型AUC值(Area Under Curve):{auc:.4f}")
# 绘制ROC曲线
plt.figure(figsize=(8, 6))
# 绘制模型ROC曲线
plt.plot(fpr, tpr, color='darkred', lw=2, label=f'KNN模型(AUC = {auc:.4f})')
# 绘制随机猜测基准线(AUC=0.5)
plt.plot([0, 1], [0, 1], color='gray', lw=1, linestyle='--', label='随机猜测(AUC=0.5)')
# 设置标签与格式
plt.xlabel('假阳性率(FPR):良性误判为恶性的比例')
plt.ylabel('真阳性率(TPR):恶性正确预测的比例')
plt.title('KNN模型测试集ROC曲线')
plt.legend(loc='lower right')
plt.grid(alpha=0.3)
plt.savefig('测试集ROC曲线.png')
# =================预测概率分布可视化=================
import seaborn as sns
import matplotlib.pyplot as plt
# 提取良性样本(真实标签1)和恶性样本(真实标签0)的预测良性概率
# 真实恶性→预测良性的概率
malignant_proba = test_pred_df[test_pred_df['真实标签'] == '恶性']['良性概率']
# 真实良性→预测良性的概率
benign_proba = test_pred_df[test_pred_df['真实标签'] == '良性']['良性概率']
# 绘制预测概率核密度图
plt.figure(figsize=(10, 6))
# 绘制真实恶性样本的预测概率分布
sns.kdeplot(data=malignant_proba, fill=True, color='red', alpha=0.5, label='真实恶性(0)')
# 绘制真实良性样本的预测概率分布
sns.kdeplot(data=benign_proba, fill=True, color='green', alpha=0.5, label='真实良性(1)')
# 添加分类阈值线(默认0.5)
plt.axvline(x=0.5, color='black', linestyle='--', linewidth=2, label='分类阈值=0.5')
# 设置标签与格式
plt.xlabel('预测为良性(1)的概率')
plt.ylabel('概率密度')
plt.title('KNN模型测试集预测概率分布')
plt.xlim(0, 1) # 概率范围限定在0-1
plt.grid(alpha=0.3)
plt.legend()
plt.savefig('测试集预测概率分布.png')