真核基因组并非杂乱无章地分布在细胞核内,而是以高度有序的染色质结构存在。通过染色质构象捕获技术及其衍生的Hi-C、ChIA-PET等多种高通量手段,人们发现基因组在三维空间中形成了诸如A、B compartments,拓扑相关结构域(TADs)和染色质环等精细结构。这些结构直接参与了增强子与启动子之间的相互作用,与基因的激活和沉默密切相关,在转录调控过程中发挥着举足轻重的作用。然而,传统3D基因组数据库多聚焦单物种分析,难以实现跨物种的比较研究。
河北农业大学研究人员发表于《Plant Communications》标题为《An integrative 3D-genome database for plants》的数据库资源文章成功构建了3D-GDP数据库。该数据库打破单物种研究局限,整合了银杏、水稻、棉花等26种植物的3D基因组数据,提供从可视化到功能分析的"一站式"平台,为解析植物3D基因组进化与基因调控机制开辟新路径。

数据库链接
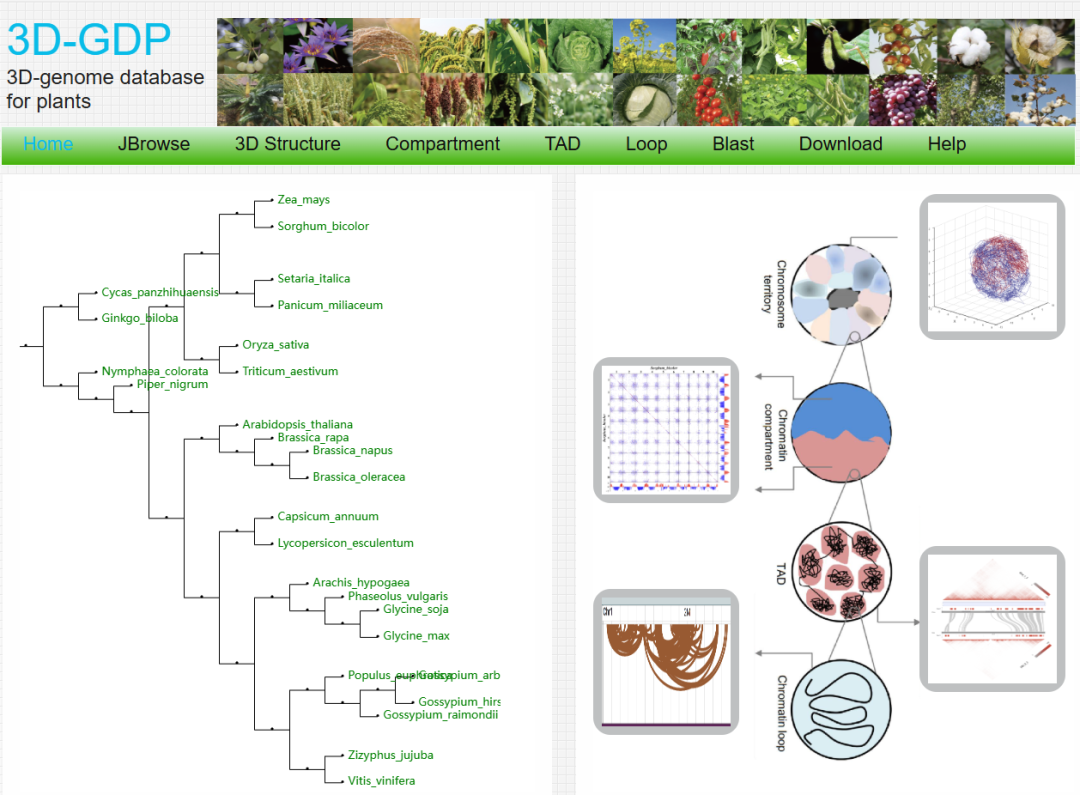
数据库首页
数据库数据规模
基因组覆盖
涵盖26种植物的383条染色体,注释1,528,029个基因,覆盖从裸子植物到双子叶植物的主要进化分支。
3D结构数据
包含约111,904个TADs(拓扑关联结构域)、8,678,833个染色质环,所有数据均来自公开的植物Hi-C数据。
注释信息
整合转录因子(TF)定位、蛋白互作网络、增强子预测(拟南芥专属)等信息,其中蛋白互作数据来自STRING数据库,功能注释基于GO和KEGG完成标准化处理。
数据库六大核心模块
3D Structure 模块
通过模拟染色质互作信息,直观展示每种植物染色质在细胞核内的空间分布,支持叠加注释(如A/B compartments、TAD位置、基因/TF/增强子定位);用户可选择物种、染色体,自定义添加注释图层,通过交互式界面缩放浏览,还可下载标注表格。
Compartment 模块
预测26种植物的A/B compartments(A区为活跃染色质,B区为异染色质),支持跨物种共线性与compartment结构的联合比较,通过热图和点图直观展示不同植物间的结构保守性。
TAD 模块
不仅可视化单物种TAD分布,还能通过"两物种比对""多物种比对"模式识别保守TAD,并预测其功能------包括分析TAD内基因的蛋白互作网络、计算互作率,以及基于GO/KEGG的功能富集。
Loop 模块
通过Circos图展示跨物种保守染色质环,支持用户查询环两端5kb内的同源基因,识别"结构保守-基因功能关联"的调控单元。
Blast 模块
用户可上传核酸/蛋白序列,选择目标物种后,数据库自动匹配高相似度序列所在的TAD,快速关联"基因序列-TAD结构",助力解析目标基因的3D调控背景。
JBrowse 模块
基于JBrowse2开发,支持实时缩放、平移,可同时展示基因组序列、基因注释、Hi-C接触热图、TAD/loop结构,兼容BEDPE、BED、HIC等格式的本地数据上传,方便研究者对比自有数据与数据库参考数据。
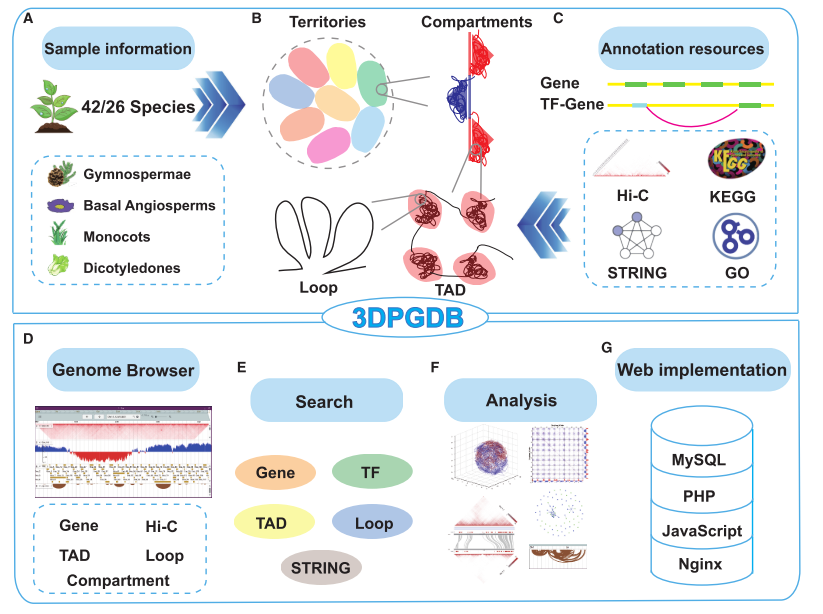
3D-GDP的构建
3D-GDP作为首个面向植物的多物种三维基因组比较平台,成功整合了26个物种的Hi-C数据与多组学注释,提供了从结构可视化、保守性分析到功能预测的一站式工具链,显著拓展了植物染色质结构研究的深度与广度。无论是解析植物3D基因组的进化规律,还是挖掘基因调控的结构基础,3D-GDP都将成为植物科研领域的核心工具。即刻访问数据库,开启你的 "三维基因组探索之旅" 吧!
参考文献
Yang Q, Wu T, Liu M, et al. An integrative 3D-genome database for plants[J]. Plant Communications, 2025, 6: 101346.
VGsoft Team(凌恩开发团队)于2015年成立,致力于为多组学相关生命科学研究,提供数据库和信息化解决方案。开发团队骨干由十年以上项目经验的资深软件工程师和生 信工程师构成,可提供灵活多样的数据库构建及信息化服务。服务项目包括物种数据库、多组学数据库、eDNA数据库、育种平台、生信云平台等,客户涵盖高校、科研院所、三甲医院及上市公司等,项目成果发表在Cell, Nucleic Acids Research, Molecular Therapy, Horticulture Research等国际著名期刊上。每一个研究领域都至少需要一个数据库,凌恩 生物开发团队秉持"数据驱动创新,客户赋能未来"的理念,提升科研价值,巩固数据成果,锁定领域影响。更多凌恩项目案例见:http://vgsoft.biozeron.com/bmstor/database/anima