0.需求
一个可怜的医学生朋友突然开始学python,从hello world开始的那种,学了一周终于学会了print()的一百种应用。但是她导师说明天就要学会使用Scanpy做项目出图,于是我带着圣光来了。
官方教程:装置------简洁
前提:已经安装好anaconda。
1.创建环境,安装依赖
(1)创建新的conda环境,我这里起名scanpy
bash
conda create -n scanpy(2)激活该环境,也就是进入到这个环境
bash
conda activate scanpy(3)根据官网,安装依赖
bash
conda install -c conda-forge scanpy python-igraph leidenalg注意是在当前的 conda 环境里安装,这里涉及到三个生物信息学/单细胞分析常用的 Python 包
scanpy:单细胞转录组分析主力库
python-igraph:图论算法(Scanpy 做聚类、PAGA 时需要)
leidenalg:Leiden 社区检测算法(Scanpy 默认的聚类算法之一)
【非必要:拉取版本代码】
如果涉及scanpy库的实现原理这些,可以用该方法获取到代码包去了解。
git仓库在这:GitHub - scverse/scanpy: Single-cell analysis in Python. Scales to >100M cells.
【如果进不去】换个镜像会快一点,但是有可能不可用:GitHub - scverse/scanpy: Single-cell analysis in Python. Scales to >100M cells.
【如果没git】官网是用很炫酷的方法直接git clone下来,但是医学生可以用更直观的方式,直接下载,不至于再学git。直接点进去链接,手动下载。
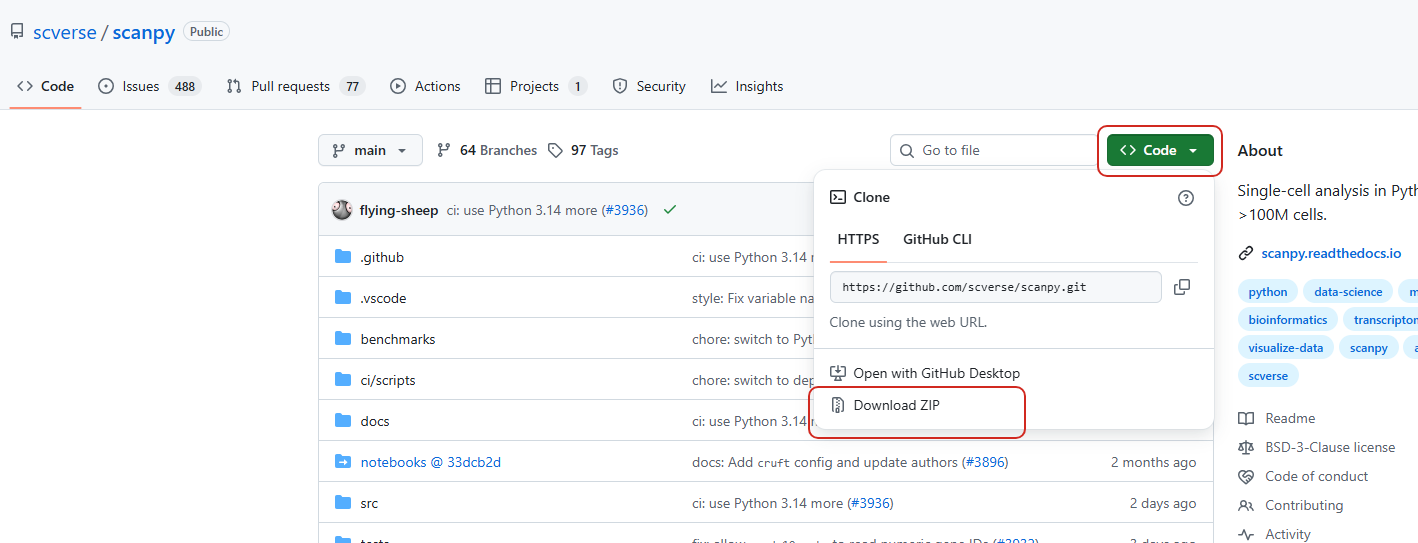
这个下载的zip包里就是要运行的代码文件夹,解压到自己喜欢的地方,然后用编译器打开。
【如果有git】直接在目标存放代码的路径下执行git命令(使用对应的镜像):
bash
git clone https://github.com/scverse/scanpy.git殊途同归,最后进入到主目录里:
bash
cd scanpy本地获取到整个目录结构:
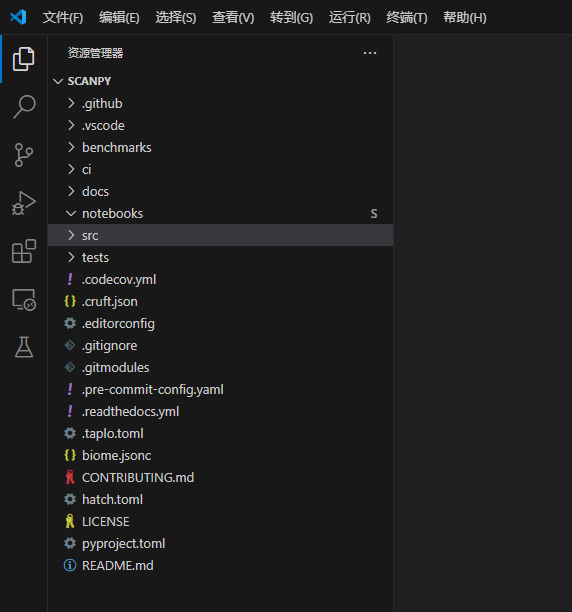
2.预处理与聚类
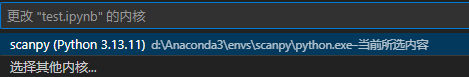
官方教程使用notebook,那我们也创建一个新的ipynb文件。注意内核选择,即前面所创建的这个环境。接下来都按照官方教程走。
2.1 基础准备
(1)引入依赖包
python
# Core scverse libraries
from __future__ import annotations
import anndata as ad
# Data retrieval
import pooch
import scanpy as sc【报错】No module named 'pooch',说明环境里没有这个包,那就在环境里装
bash
conda install -c conda-forge pooch(2)配置画图默认样式
一个全局配置,把以后所有 scanpy.pl.* 出的图,一次性设成尺寸50、背景纯白。
python
sc.settings.set_figure_params(dpi=50, facecolor="white")(3)下载数据
官方教程是联网下载了 figshare 上 DOI 对应的那个数据集。
python
EXAMPLE_DATA = pooch.create(
path=pooch.os_cache("scverse_tutorials"),
base_url="doi:10.6084/m9.figshare.22716739.v1/",
)
EXAMPLE_DATA.load_registry_from_doi()【报错】国内网络连 DOI 解析问题 。按照这个方法在本地下载:Scanpy教程-预处理和聚类数据集-CSDN博客
然后把下载后的2个h5文件放到缓存路径里。
【缓存路径的获取与创建】
python
import pooch, os
path = pooch.os_cache("scverse_tutorials")
print(path)
os.makedirs(path, exist_ok=True) 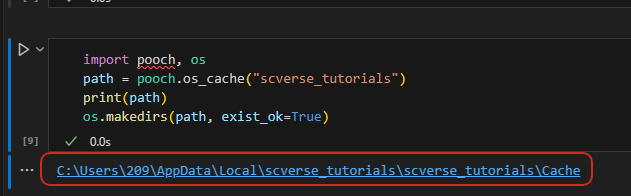
放完两个文件之后运行以下代码,pooch会从缓存里取到数据,就不会再联网下载了。文件现在被存放在 EXAMPLE_DATA 这个变量里。
python
registry = {
"s1d1_filtered_feature_bc_matrix.h5": "sha256:322c30a7a4905f7f113472442d4aa2c81a1ad736c86651f6c0b81e5b2ff94ac8",
"s1d3_filtered_feature_bc_matrix.h5": "sha256:ca40b287fac57ac2048f11f56f1b630c7c188718908fc217c6baf2e49bdb1982",
}
EXAMPLE_DATA = pooch.create(
path=pooch.os_cache("scverse_tutorials"),
base_url="", # 不需要联网
registry=registry,
)
# 测试获取文件路径
path1 = EXAMPLE_DATA.fetch("s1d1_filtered_feature_bc_matrix.h5")
path3 = EXAMPLE_DATA.fetch("s1d3_filtered_feature_bc_matrix.h5")
print("文件路径:")
print(path1)
print(path3)(4)加载与查看数据
从 EXAMPLE_DATA 里逐个抓取文件读取数据。
python
samples = {
"s1d1": "s1d1_filtered_feature_bc_matrix.h5",
"s1d3": "s1d3_filtered_feature_bc_matrix.h5",
}
adatas = {}
for sample_id, filename in samples.items():
path = EXAMPLE_DATA.fetch(filename)
sample_adata = sc.read_10x_h5(path)
sample_adata.var_names_make_unique()
adatas[sample_id] = sample_adata
adata = ad.concat(adatas, label="sample")
adata.obs_names_make_unique()
print(adata.obs["sample"].value_counts())
adata