超级增强子(SE)作为驱动细胞身份决定和关键致病基因表达的核心调控元件,一直是表观遗传学领域的研究热点,也是近年国自然的热点。然而,长期以来,相关的数据资源和注释工具主要集中于人类和小鼠模型。对于植物科学领域,尤其是主要农作物而言,缺乏一个系统性、综合性的超级增强子注释平台,这在很大程度上限制了我们从全基因组层面解析植物生长发育与逆境响应的调控网络。
近期,哈尔滨医科大学团队在Nucleic Acids Research发表了最新升级的SEdb 3.0。这一版本实现了跨越式的更新:它不仅将人类和小鼠的样本量扩充了一倍,更具里程碑意义的是,首次将拟南芥和玉米纳入数据库体系,正式打破了动植物在超级增强子研究上的数据壁垒。
此外,SEdb 3.0不再局限于位点信息的展示,而是通过整合eRNA、转录辅因子及染色质调节因子等多维数据,深入解析了SE的分子调控机制。在单细胞测序技术日益普及的背景下,SEdb 3.0 的发布为研究人员从更高维度理解细胞命运决定机制,以及挖掘作物重要农艺性状的调控靶点,提供了不可或缺的生物信息学工具。
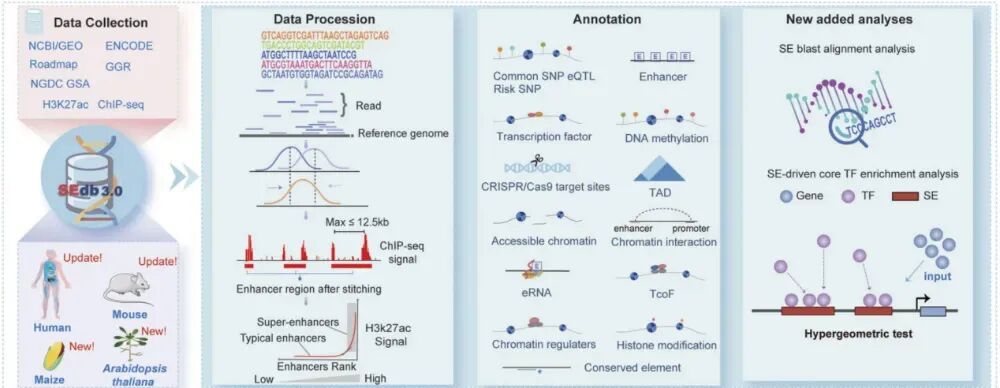
SEdb 3.0的核心升级亮点
0 1 跨界扩容(最大亮点)
新增了拟南芥和玉米两个植物物种!这标志着该数据库从医学/动物领域跨越到了植物科学领域。
02 数据量暴增
涵盖4个物种(人、鼠、拟南芥、玉米)。
收录了来自5387个ChIP-seq样本的~348万个超级增强子。
人类和小鼠的数据量相比2.0版本翻了一倍。
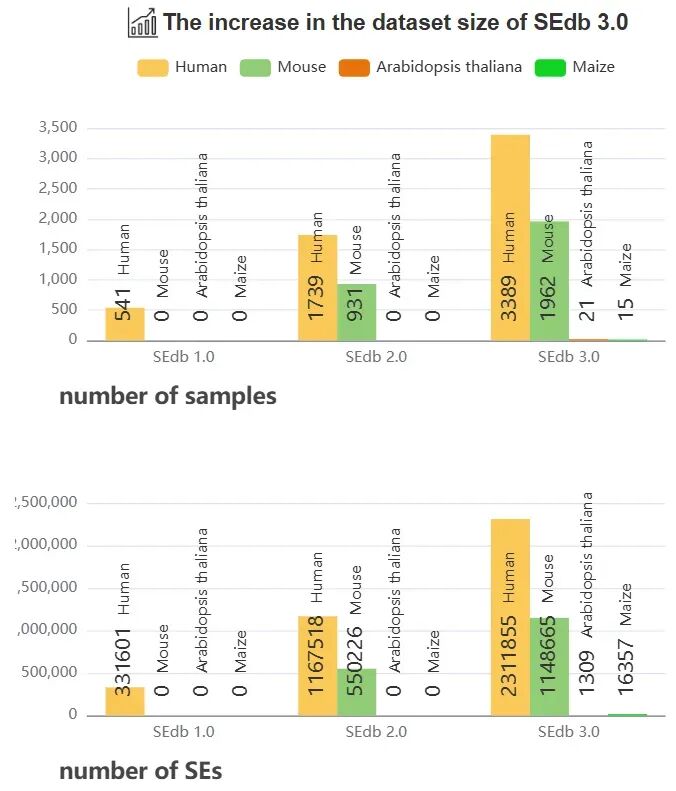
03 深度功能注释(机制层面)
新增了大量"表观遗传"和"转录调控"特征:
-
eRNAs:提供了SE转录活性的直接证据。
-
TcoFs & CRs:揭示了是谁(辅因子、染色质重塑复合物)在维持SE的开放状态和功能。
-
植物特有数据:整合了DHS(DNase I超敏感位点)、保守元件、植物组蛋白修饰等。
04 新增实用工具
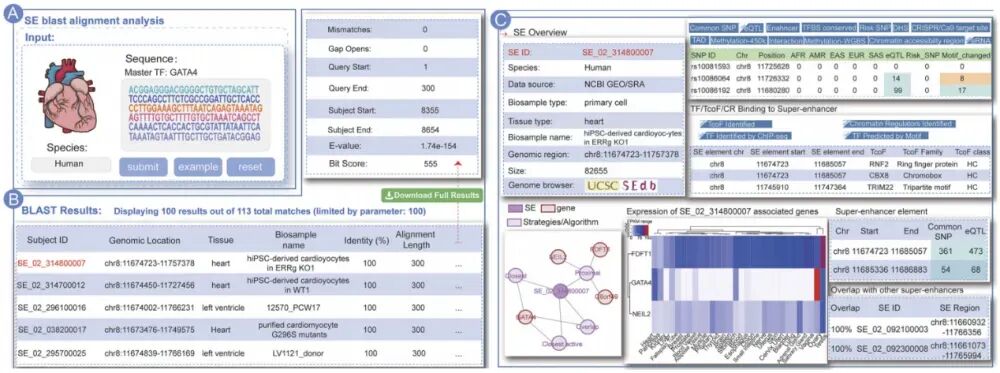
- SE Blast Alignment:我们可以上传一段序列,直接比对看看它是不是潜在的超级增强子。
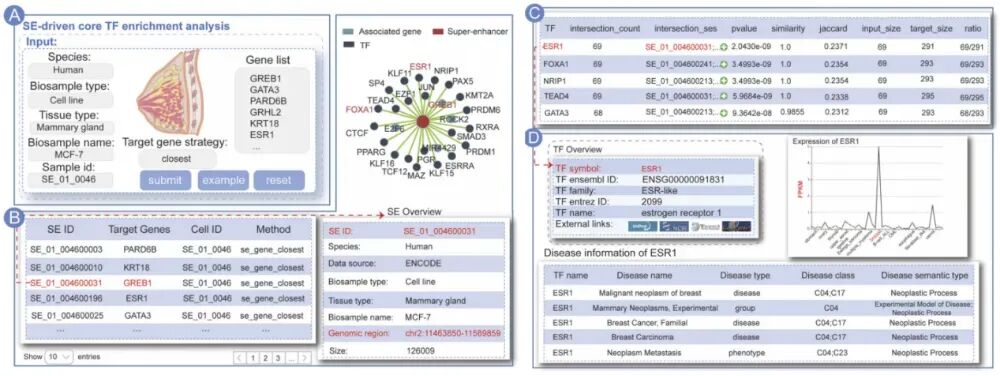
- SE-driven Core TF Enrichment:输入一组基因,自动分析背后的核心转录因子(Core TFs)是谁。
05 靶基因预测升级
- 靶基因预测升级:引入了ABC模型 (Activity-by-Contact) 和BETA算法,让"增强子-靶基因"的配对更准确。
使用教程
官网:http://www.licpathway.net:8081/sedb/

官网界面
网站包含多个板块:Data-Browse(数据浏览)、Search(搜索)、Analysis(分析)、Genome-Browser (基因组浏览器)、Download(下载)、Help(帮助)等。

主要板块介绍
0 1 数据浏览Data-Browse
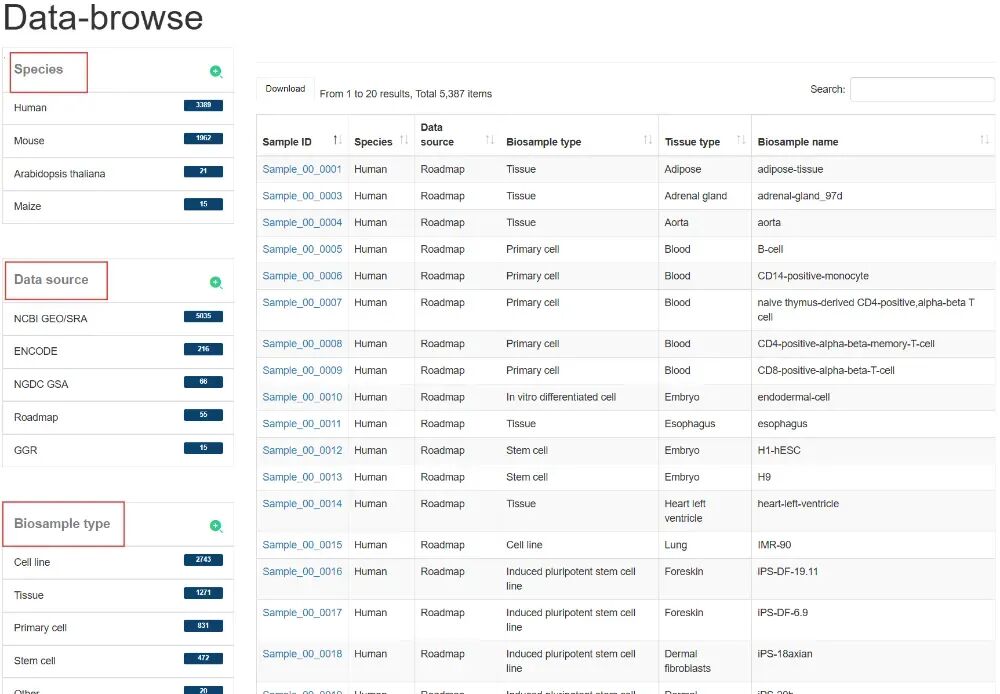
"数据浏览"页面让我们可以通过"物种"、"数据来源"、"生物样本类型"和"组织类型"快速搜索样本并自定义筛选条件。
02 搜索Search
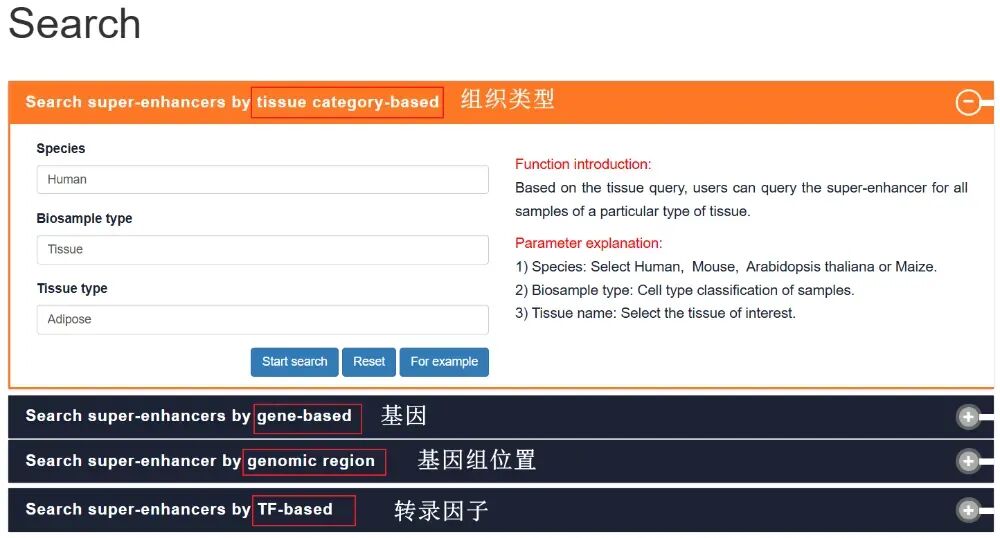
我们可以通过感兴趣的组织、基因、基因组位置和转录因子来确定超级增强子查询的范围。以组织类型为例,依次填写Human(人类)→Tissue(组织)→Adipos(脂肪),点击"Start search"。即可跳转搜索结果。
03 分析Analysis
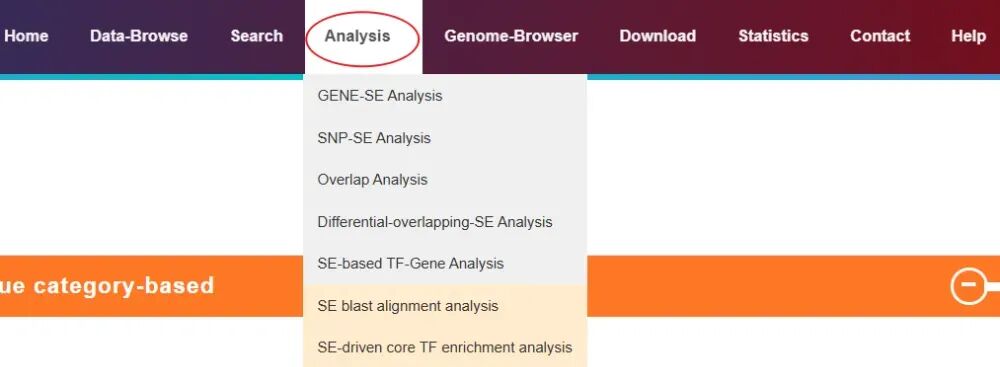
点击菜单栏的"Analysis",可以看到有多种类型的分析。可以根据研究目的,选择合适的分析。
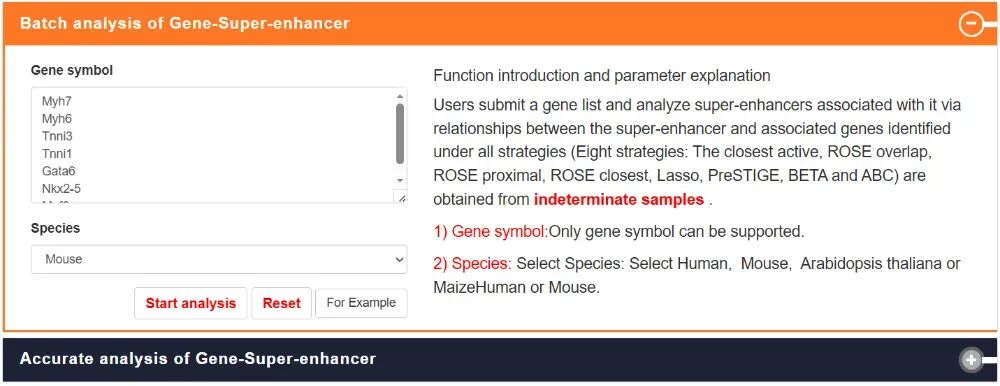
GENE-SE Analysis:可以进行基因-超级增强子的批量分析和基因-超级增强子的准确分析,提交基因并通过不同策略确定与其相关的超级增强子。
SNP-SE Analysis:分析超增强子区域的常见SNP,我们可以提交一个SNP并查找其出现的超级增强子、该超级增强子的注释信息及五大超群体的连锁不平衡(SNP)。
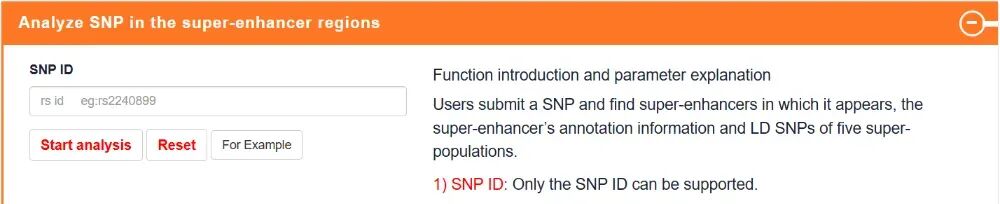
Overlap Analysis:重叠分析,提交bed文件,并通过设置bed文件中提交区域的重叠百分比,识别与提交区域有重叠关系的超级增强子。
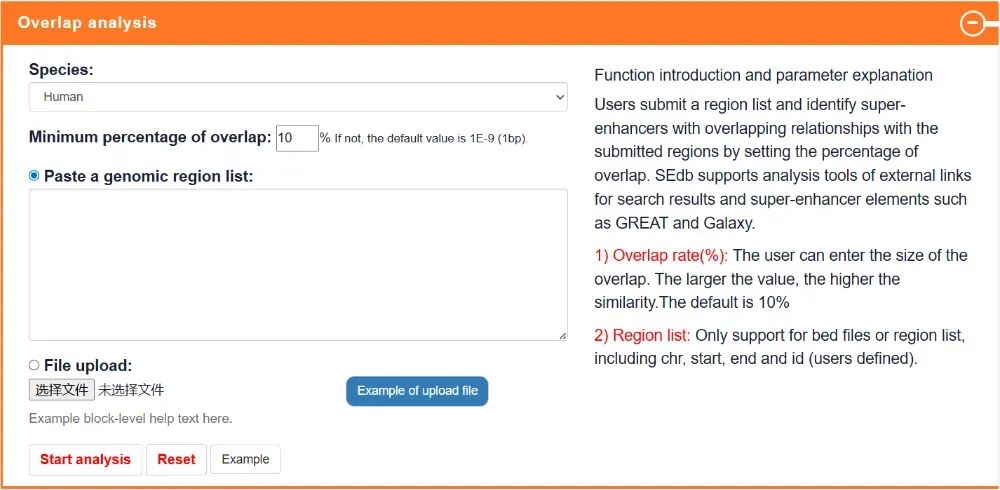
Differential-overlapping-SE Analysis:差异重叠超级增强子分析,在线分析两个提交样本之间的差异和重叠的超级增强子。
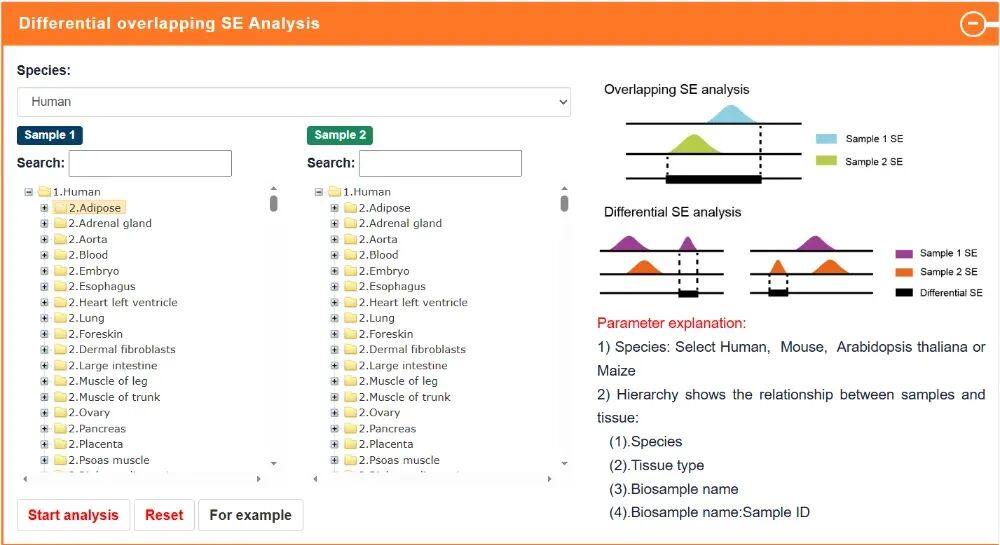
SE-based TF-GENE Analysis:基于超级增强子的转录因子-基因分析,显示与超级增强子相关的转录因子-基因对的数量、基因组区域和样本信息。我们可以提交两份转录因子和基因列表,同时选择物种为人类或小鼠,SEdb 3.0 将分析由超级增强子介导的全面转录因子-基因对。
SE blast alignment analysis:输入基因组序列或上传序列文件。SEdb 3.0将使用BLAST对超级增强子数据库进行序列比对,并检索最相似的超级增强子。
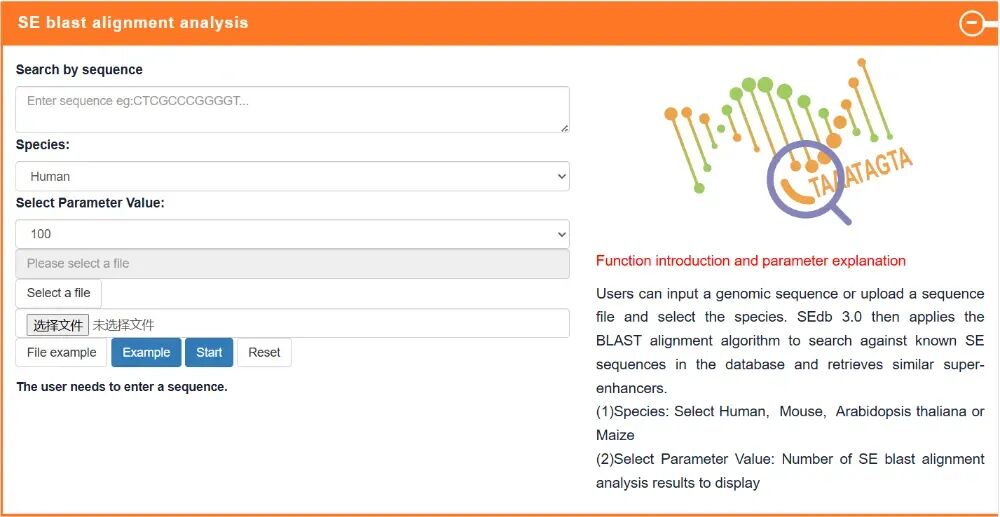
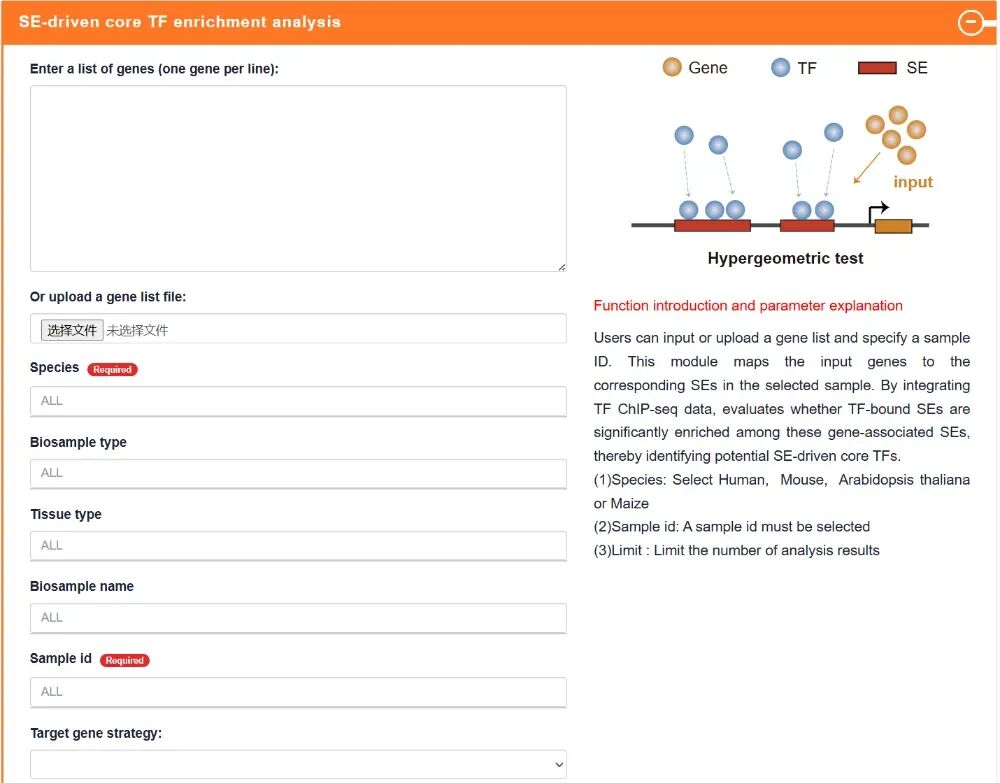
SE-driven core TF enrichment analysis:SE驱动的核心转录因子富集分析,输入基因列表或上传基因列表文件。识别可能作为输入基因集中超级增强子介导转录程序关键调控因子的转录因子。
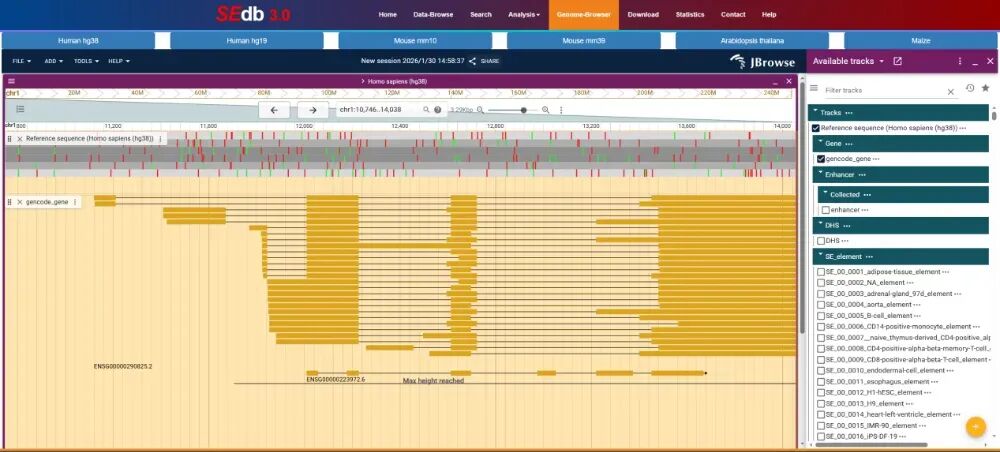
04 基因组浏览器Genome-Browser
基因组浏览器包含多个参考基因组,包括人类(Hg38和Hg19)、小鼠(Mm10和Mm39)、拟南芥(TAIR10)和玉米(B73)。 Hg38和Mm10作为人类和小鼠的默认参考基因组显示。 我们通过ChIP-seq数据和TAD观察超级增强子与邻近基因、基因组片段、SNP、常见SNP、风险SNP、DHS、增强子、保守TFBS、TFBS的接近程度。
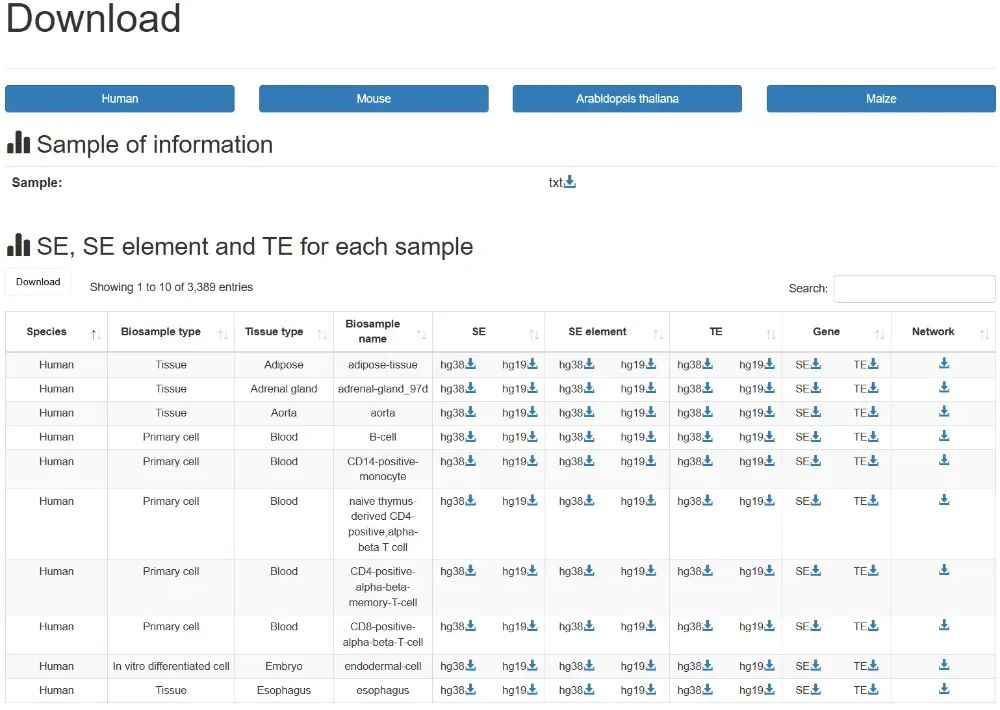
05 下载Download
数据库涵盖了多个参考基因组,包括hg19、hg38、mm10、mm39、B73和TAIR10。对于每个样本,我们可以下载超级增强子、超级增强子元件和典型增强子,以及超级增强子调控网络数据和与超级增强子及典型增强子相关的基因。此外,该数据库还提供全面的遗传和表观遗传注释,包括常见SNP、风险SNP、表达数量性状位点(eQTLs)、拓扑关联结构域(TADs)、增强子RNA(eRNAs)、转录辅因子(TcoFs)、染色质重塑复合物(CRs)、转录因子ChIP-seq 数据和基序(motif)数据等,从而推动多维度的超级增强子研究。不仅如此,所有搜索结果、超级增强子详情和分析输出都可以通过相应页面上的"下载"按钮直接下载。
如何使用数据库
很多小伙伴碰到的问题是:"我发现了一个表型相关的基因(如MYC或玉米的ZmPFKB),我想知道它是否受超级增强子调控?"那么我们今天以这个简单做个示范。
点击Search,选择第二个标签"Search Super-enhancers by gene-based",输入相应的基因、物种和策略,点击"Start search"。

跳转页面后,我们可以看到STX12相关的超级增强子列表。
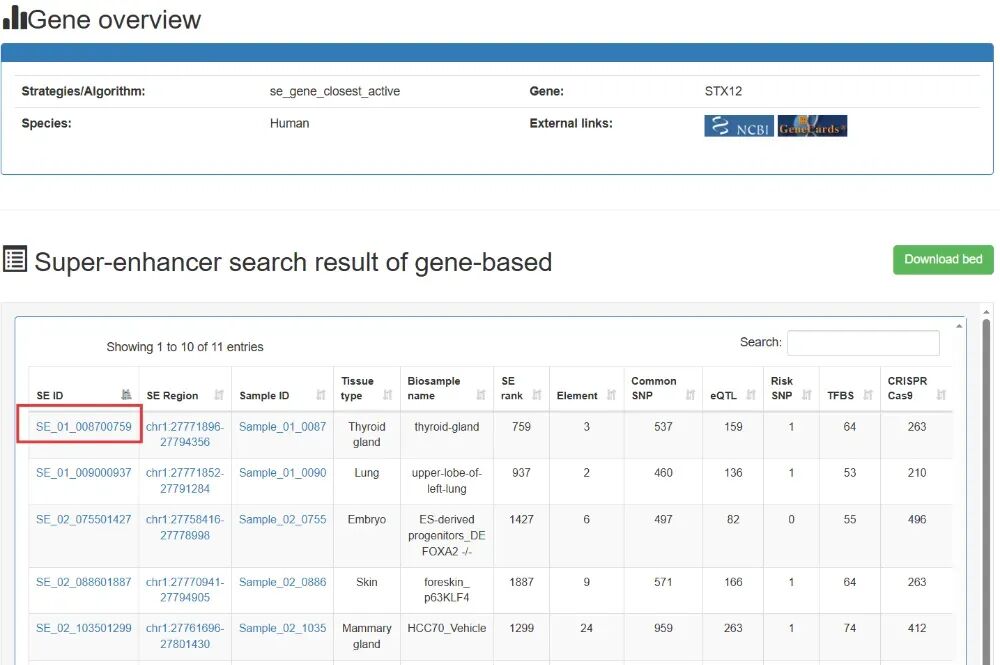
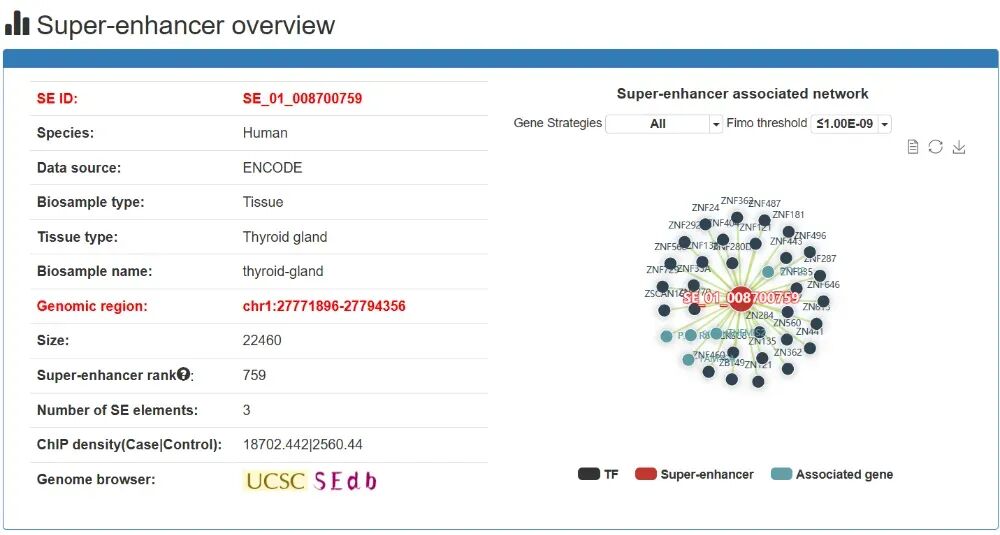
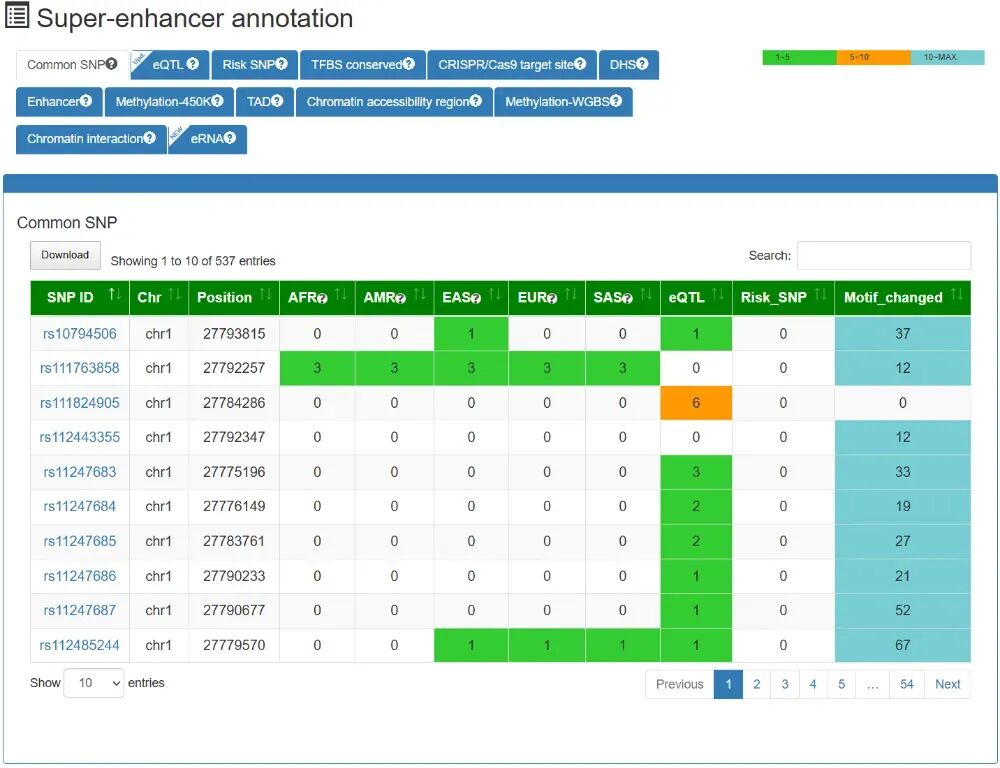
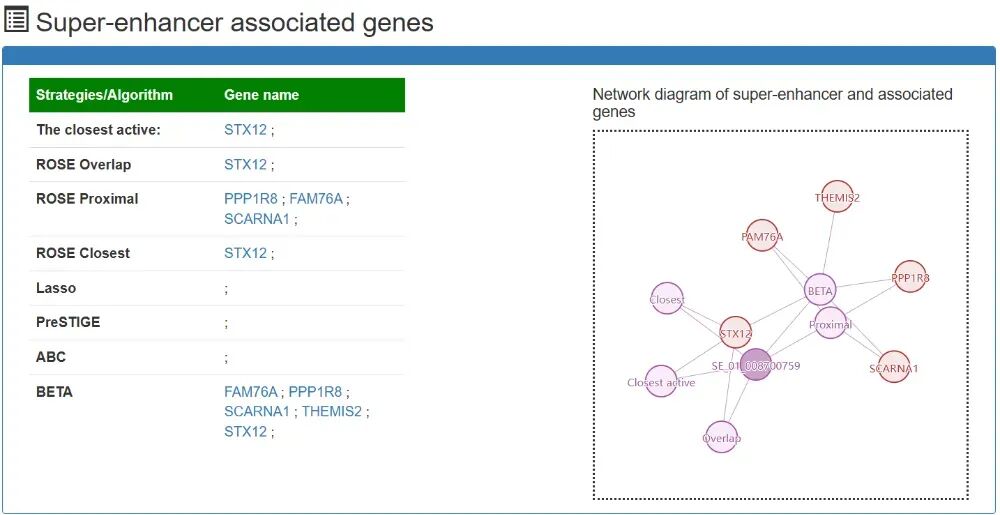
点击其中一个SE ID,可以看到这个超级增强子详细的信息,包含超级增强子概述、超级增强子注释信息、超级增强子相关基因、超级增强子相关基因的表达、结合到超级增强子上的TF/TcoF/CR和超级增强子元件(H3K27ac)。
- A. 超级增强子概述
- B. 超级增强子注释信息
- C. 超级增强子相关基因
- D. 超级增强子相关基因的表达
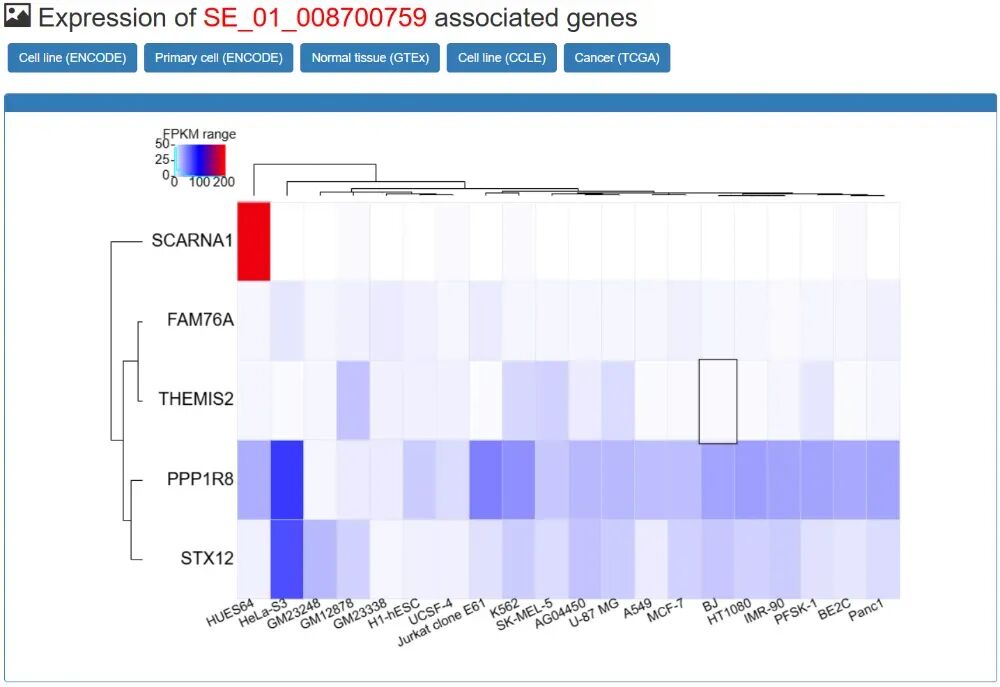
- E. 结合到超级增强子上的TF/TcoF/CR
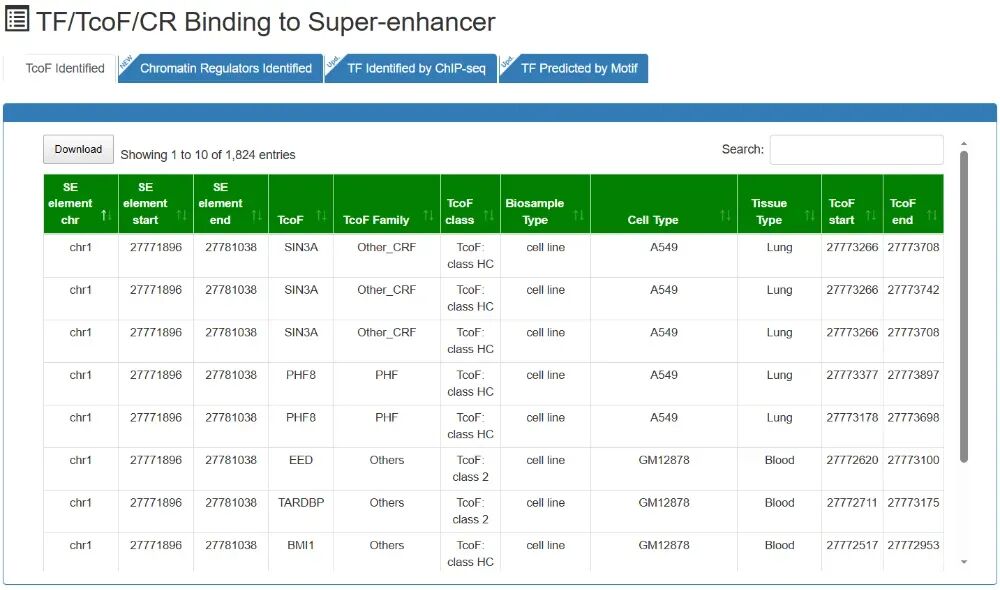
- F. 超级增强子元件(H3K27ac)
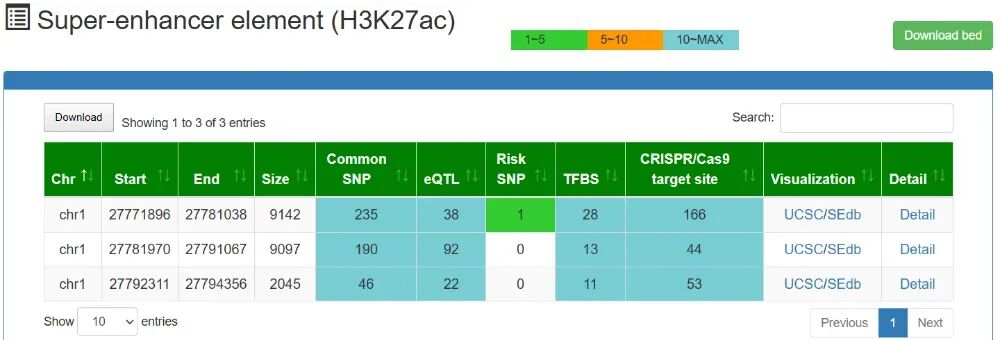
今天的介绍就到这里了,如需做增强子、转录因子、组蛋白修饰相关的研究欢迎联系我们,爱基百客提供领先的表观组学技术服务(ATAC-seq、ChIP-seq、CUT&Tag、WGBS等)