类似于 【AI基础】第三步:纯天然保姆喂饭级-安装并运行chatglm2-6b,有一些细节不一样。
此系列文章列表:
【AI基础】第一步:安装python开发环境-windows篇_下载安装ai环境python
【AI基础】第一步:安装python开发环境-conda篇_minicode怎么换虚拟环境
【AI基础】第三步:纯天然保姆喂饭级-安装并运行chatglm2-6b
一、安装miniconda
参考 【AI基础】第一步:安装python开发环境-conda篇_minicode怎么换虚拟环境-CSDN博客
二、安装CUDA、cuDNN和pyTorch
bash
> pip install protobuf transformers==4.40.0 cpm_kernels torch>=2.3.0 sentencepiece accelerate三、安装RUST
参考 【AI基础】第三步:纯天然保姆喂饭级-安装并运行chatglm2-6b 三、安装RUST
四、运行chatglm3
4.1 创建环境
bash
> conda create --name chatglm3
> conda activate chatglm3
> git clone https://github.com/THUDM/ChatGLM3
> cd chatglm34.2 调整
在chatglm3的依赖文件中,有vllm模块,此模块存在于linux系统中,在windows中安装时需要注释掉。

4.3 安装
bash
> pip install -r requirements.txt --verbose -i https://pypi.tuna.tsinghua.edu.cn/simple/- --verbose,会显示实时进度,避免下载大文件时耗时过长,造成卡死的假象。
- -i https://pypi.tuna.tsinghua.edu.cn/simple/,使用清华源,避免从官方下载造成速度慢。
 安装完成。
安装完成。
4.4 下载大模型文件
4.4.1 HuggingFace下载
下载地址:https://huggingface.co/THUDM/chatglm3-6b

4.4.2 国内下载
综合镜像站:
- 魔搭modelscope:chatglm3-6b 模型库-魔搭社区
- AI快站:AI快站 - HuggingFace模型免费加速下载 - chatglm3-6b
- 异型岛:互联高科 - chatglm3-6b
- HuggingFace镜像站:THUDM/chatglm3-6b · HF Mirror
4.4.3 下载完成
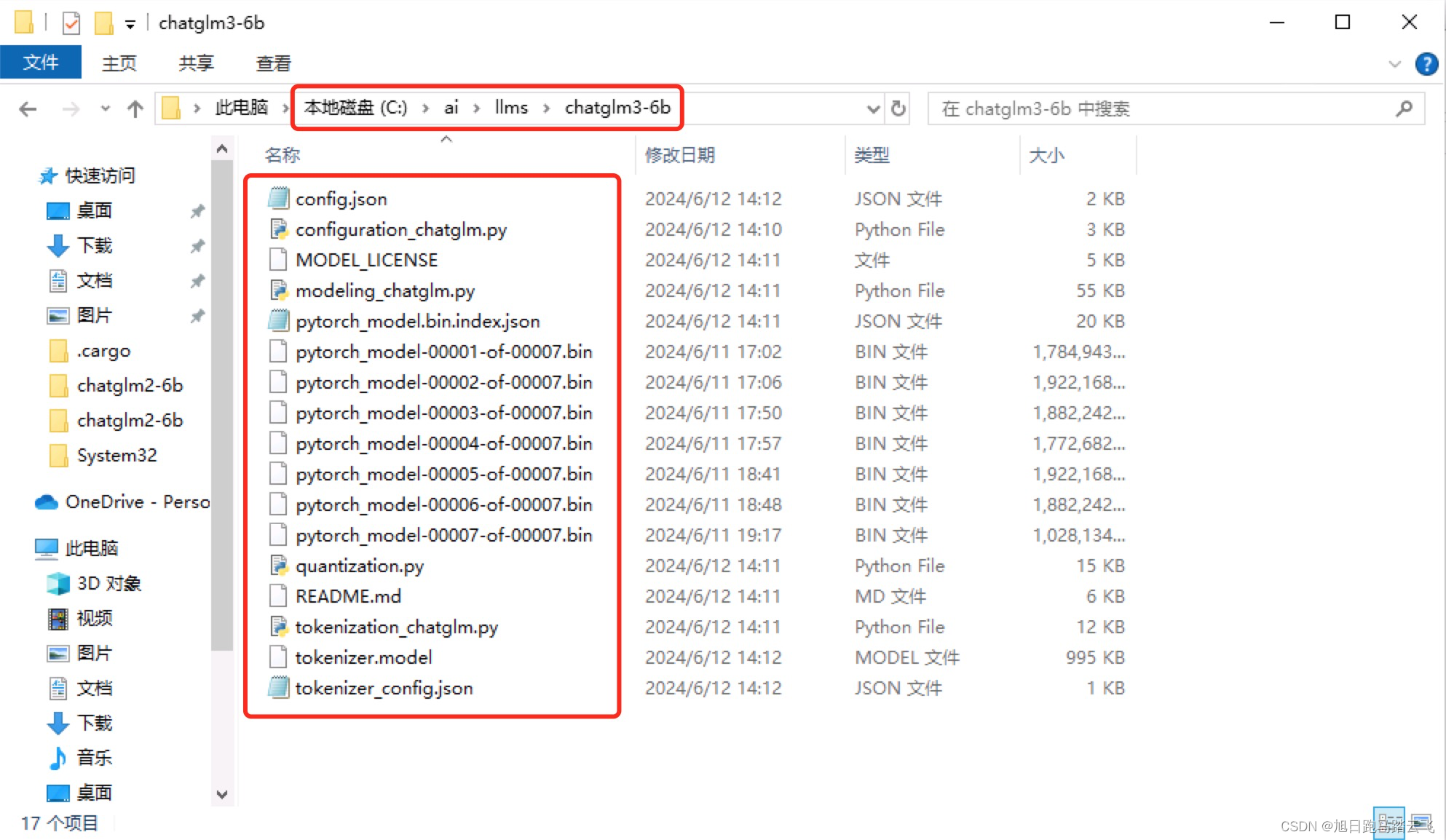
下载到本地的默认下载地址,文件结构如图(图中文件,一个都不能少):
4.5 运行代码
4.5.1 官方示例
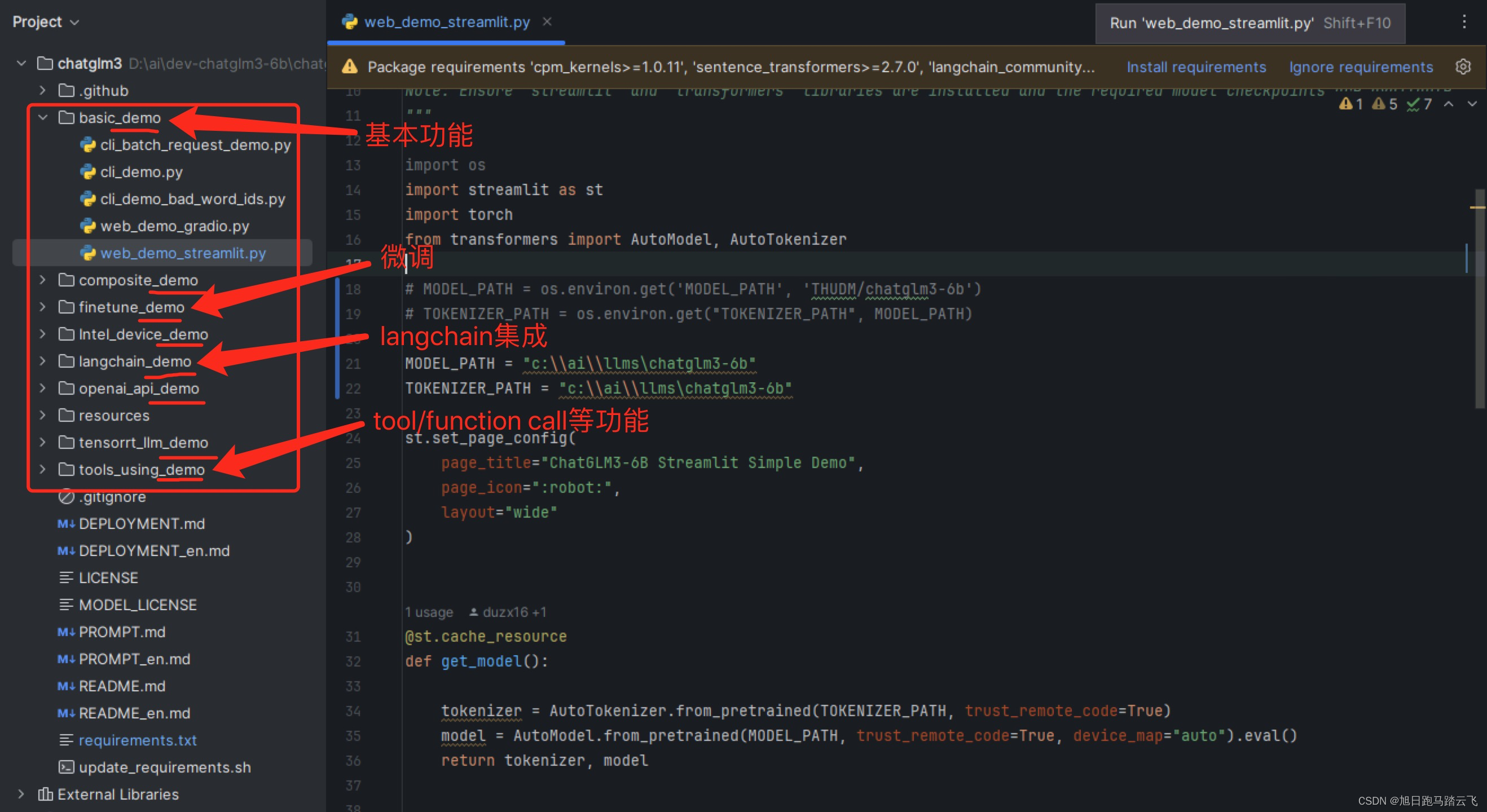
在 4.3 中下载的源码中我们可以看到多个以 _demo 为结尾的文件夹,这些都是官方示例。
4.5.2 运行网页端
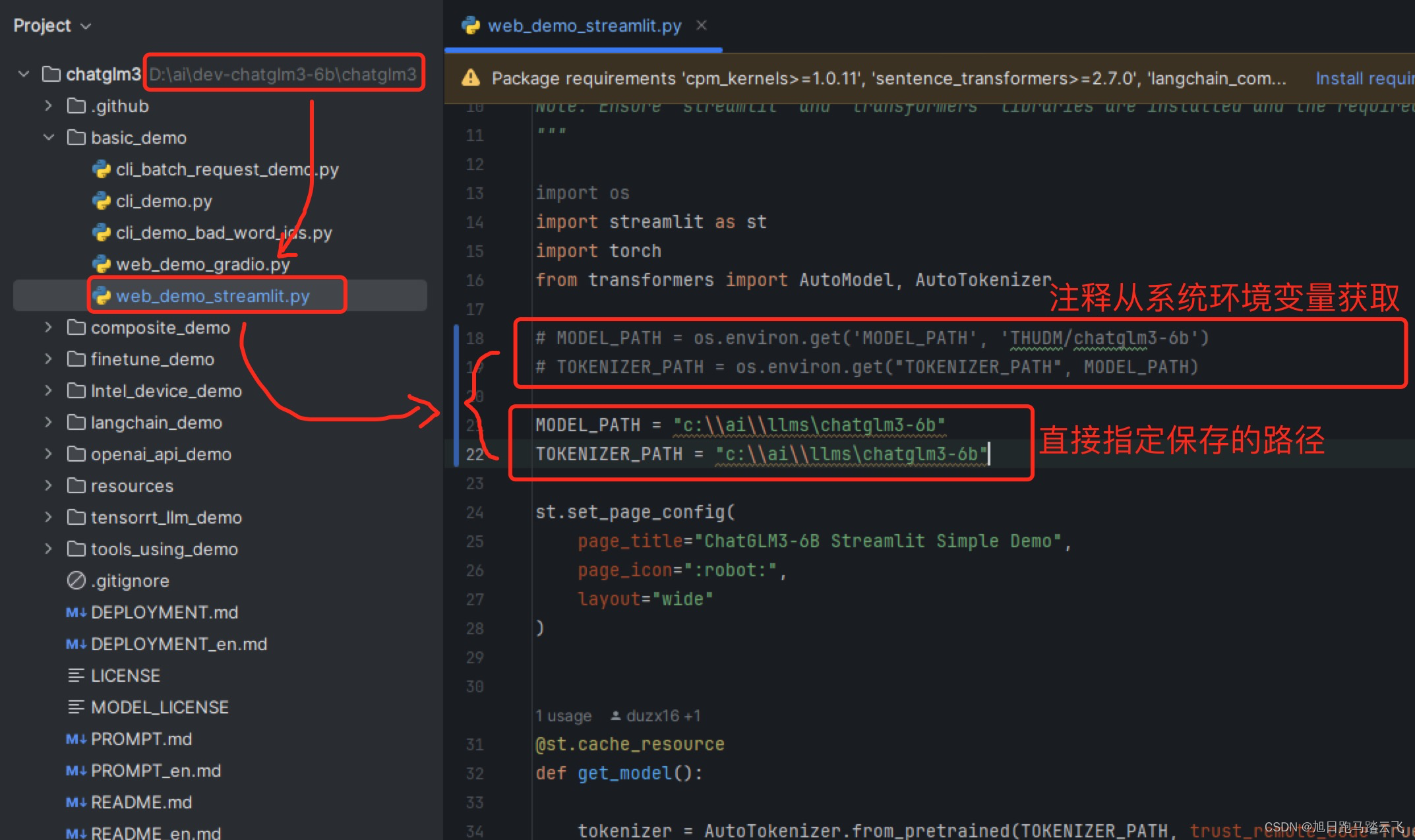
修改大模型路径:
运行网页端命令:
bash
> streamlit run basic_demo\web_demo_streamlit.py
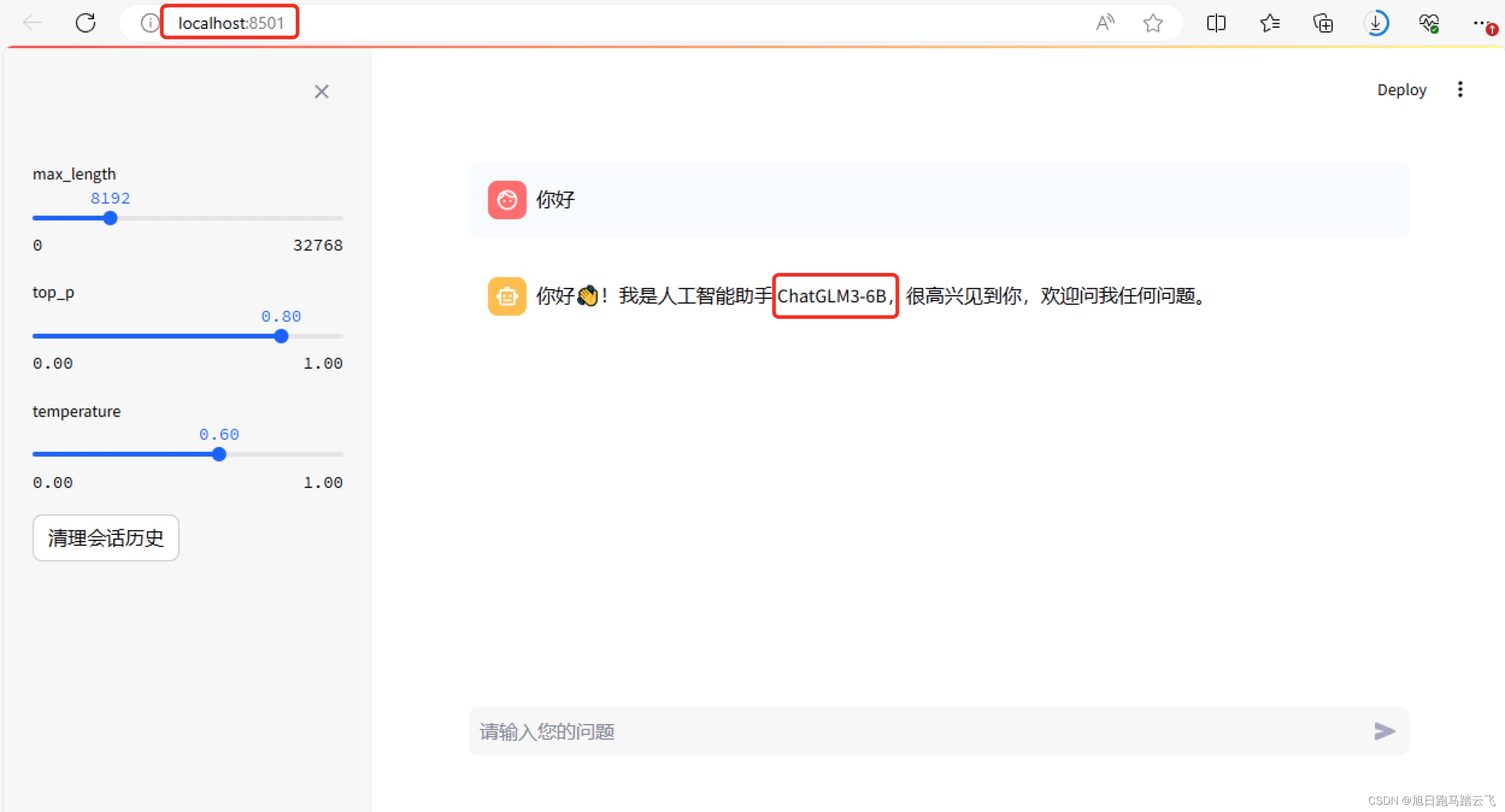
运行成功。