使用susieR鉴定多个因果变异位点只需要两个输入文件,一个输入文件是包含Zscore值的SNP位点(zscore.txt),另一个文件是LD matrix(LD.matrix.ld)。
zscore.txt 文件如下所示:
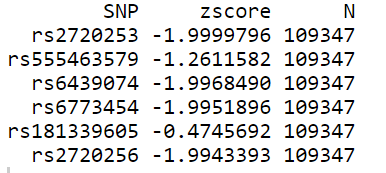
LD.matrix.ld 文件如下所示:

LD.matrix.ld 文件是通过plink生成的,使用到的命令如下:
bash
plink --bfile file --r2 --matrix --out LD.matrix其中,file是指包含zscore.txt文件中所有SNP的plink格式文件。注意,file.bim的SNP顺序要跟zscore.txt的SNP一列的顺序完全一致,不然后面运行susieR的时候会报错。
通过以上命令得到LD.matrix.ld 文件后,即可通过susieR包鉴定多个因果变异位点,如下所示:
r
install.packages("susieR")
rm(list=ls())
library(susieR)
library(data.table)
eq=read.table("zscore.txt", sep="\t", stringsAsFactors=FALSE,header=F)
dat <- fread("LD.matrix.ld")
dat1=as.matrix(dat)
fitted_rss3 <- susie_rss(c(eq$zscore), dat1, n=eq$N, L = 10)
susie_plot(fitted_rss3, y="PIP")
summary(fitted_rss3)$cs运行susie_plot(fitted_rss3, y="PIP")命令后得到如下图像:

其中,真正的因果变量以红色显示。95%的因果集通过三种不同的颜色(绿色、紫色、蓝色)来表示。
运行summary(fitted_rss3)$cs命令后得到如下结果:
# cs cs_log10bf cs_avg_r2 cs_min_r2
# 1 2 4.033879 1.0000000 1.0000000
# 2 1 6.744086 0.9634847 0.9634847
# 3 3 3.461470 0.9293299 0.7545197
# variable
# 1 653
# 2 773,777
# 3 362,365,372,373,374,379,381,383,384,386,387,388,389,391,392,396,397,398,399,400,401,403,404,405,407,408,415结果表示这三个因果信号已被三个因果集(CSs)捕获。注意的是,第三个因果集中包含许多变量,包括真正的因果变量403。