2025年7月2日 bioRxiv预印了一篇文章 In Situ Inference of Copy Number Variations in Image-Based Spatial Transcriptomics 提出iST-CNV方法,实现了从iST数据中推断CNV,并系统评估了技术限制与临床应用潜力。
引言:CNV与肿瘤研究的挑战
拷贝数变异(CNV)作为癌症驱动因素,在肿瘤异质性和治疗抵抗中起关键作用。传统基于测序的空间转录组技术(sST)虽能解析CNV,但受限于分辨率低(多为多细胞混合spot)和检测效率不足,难以实现单细胞精度的肿瘤克隆空间定位。近年来,成像空间转录组(iST,如CosMx、Xenium)凭借高分辨率(单细胞水平)和原位保留空间信息的能力崭露头角,但其基因覆盖度有限(通常数百至数千基因),CNV推断一直未被突破。
方法创新:RNA velocity启发的信号增强策略
1. 算法核心思想
该算法受RNA velocity模型中"细胞邻域信息传递"的启发,通过加权平均细胞及其邻近细胞的表达谱,增强低丰度基因信号并降低技术噪声。其核心假设是:
空间或转录组邻近的细胞可能处于相似的生物学状态,共享相似的CNV模式。
2. 数学建模与实现步骤
(1) 邻域图构建
-
输入数据:单细胞/空间表达矩阵(基因×细胞),通常已进行基础归一化和log转换
-
邻域定义:
-
基于k近邻图 (k-NN graph)或空间距离(针对iST数据)
-
距离度量:余弦相似度(转录组相似性)或欧氏距离(物理空间邻近性)
-
示例参数:CRC研究中使用100个转录组邻居(k=100)
-
(2) 权重计算
邻域细胞j对目标细胞i的贡献权重 ( w_{ij} ) 通过以下方式确定:
-
转录组相似性权重:其中 ( d_{ij} ) 是细胞i与j的表达谱距离,γ为衰减系数(默认γ=1)
-
空间距离权重(可选): 对iST数据可额外引入空间衰减因子,如高斯核函数:(( x_i )为细胞i的空间坐标,σ控制衰减范围)
(3) 表达谱平滑
目标细胞i的平滑后表达值 ( M_i ) 计算为:
其中 ( X_j ) 为邻域细胞j的归一化表达向量。实际操作中:
-
稀疏性处理:对零计数基因采用伪计数填充(如+1)
-
迭代平滑:可重复2-3次以增强信号(但需避免过度平滑)
(4) 下游CNV推断
平滑后的矩阵 ( M ) 输入至改进版inferCNVpy,关键调整包括:
-
参考细胞选择:使用病理注释的正常区域细胞或非上皮细胞作为基线
-
动态阈值:针对CNV增益(gain)和缺失(loss)分别设置截断值(如|LFC|>0.1)
3. 生物学与技术优势
(1) 解决iST数据痛点
-
低检测效率:CosMx平均仅973 counts/细胞,传统方法信噪比低
-
基因覆盖有限:即使WTx面板(~20k基因),单个基因仍稀疏
-
空间信息保留:避免解离式单细胞测序的空间信息丢失
(2) 与RNA速度的关联
-
共同理论基础:均利用细胞状态连续变化的假设(但RNA velocity关注剪切动力学,此处关注CNV稳定性)
-
关键差异 :
维度 RNA速度模型 CNV平滑模型 动态类型 转录瞬时变化(u/s RNA) 基因组结构变异(长期稳定) 时间尺度 分钟至小时级 克隆进化(月/年级) 输入数据 需未剪切/剪切mRNA 仅需成熟mRNA
4. 技术验证:性能评估与关键发现
1. 双平台一致性验证
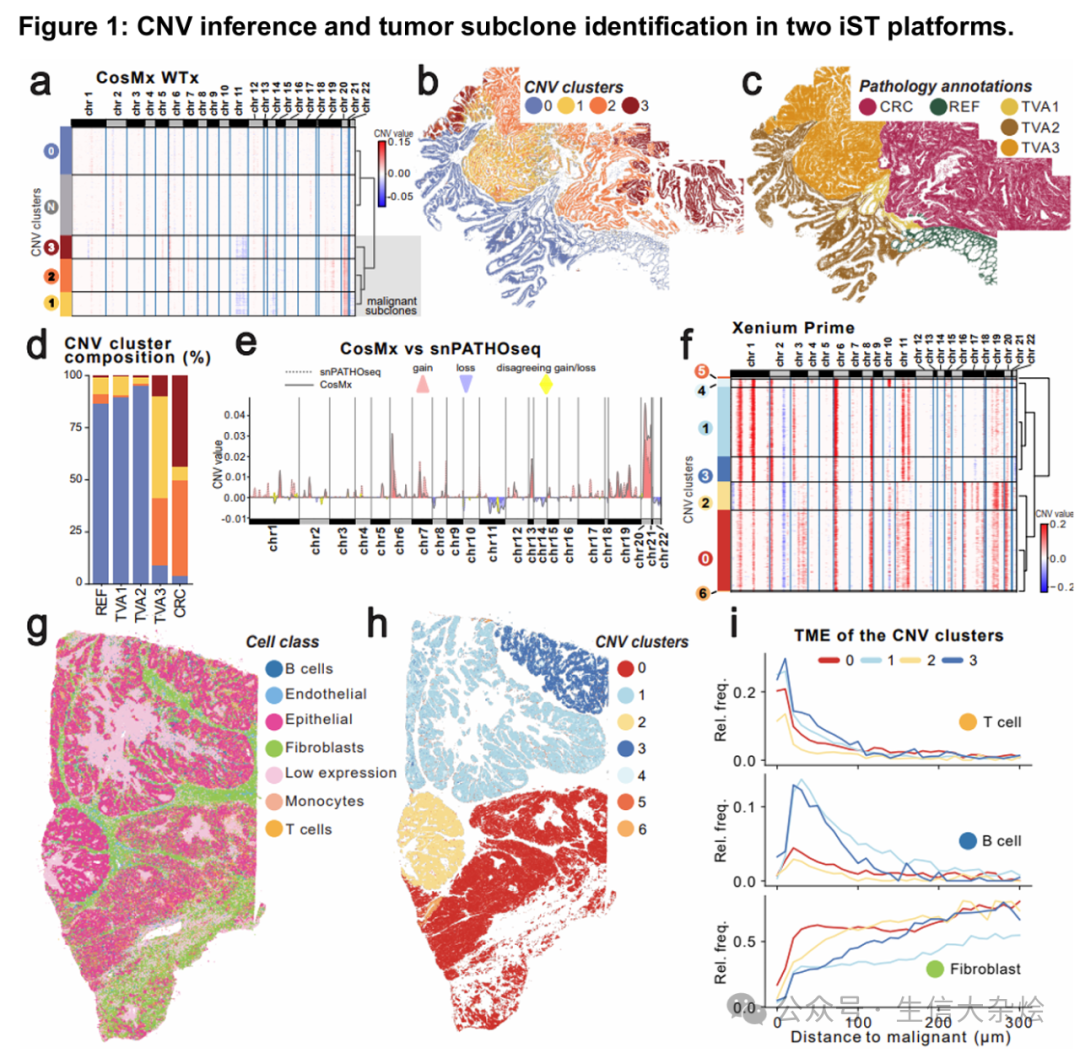
研究团队对结直肠癌(CRC)配对样本分别进行CosMx iST 和**单核PATHO-seq(snPATHO-seq)**分析,结果显示:
-
CNV图谱高度一致:均检测到CRC典型变异(如13q增益、8p缺失),且恶性亚克隆空间分布与病理注释(腺瘤/癌变区域)吻合(Dice系数0.837)。
-
三克隆进化模型:发现3个CNV定义的上皮亚克隆,其中一簇富集于晚期腺瘤(TVA3),提示早期肿瘤进展。
2. 技术限制的系统评估
通过模拟63种数据场景(不同基因面板大小、检测效率),揭示关键性能边界:
-
基因面板阈值:<1000基因的panel无法有效预测CNV(AUC<0.7),而≥2000基因时性能饱和(图2c)。
-
检测效率:CNV增益预测在1000 counts/细胞时达平台期,而缺失预测始终较差(AUC<0.8),可能与低表达基因的噪声有关。
-
CNV大小影响:大片段CNV(10-20 Mb)比小片段(1-5 Mb)更易检测(F1-score提升30%)。
3. 卵巢癌的空间微环境解析
在高级别浆液性卵巢癌(HGSOC)的Xenium数据中:
-
克隆-微环境互作:4个主要CNV亚克隆(占95.7%细胞)呈现空间区室化,其中克隆1/3周围富集T/B细胞,克隆0/2则与成纤维细胞共定位(与早期复发相关)。
-
临床关联性:8q24扩增(已知卵巢癌驱动因子)被精准检出,验证了方法的生物学相关性。
技术局限性
1. 检测灵敏度边界
-
基因面板依赖 :
<1000基因面板的CNV预测近乎随机(AUC<0.7),500基因面板完全失效
-
计数深度限制 :
CNV增益检测需≥1000 counts/细胞,缺失检测始终较差(AUC<0.8)
-
CNV大小影响 :
1-5 Mb小片段检出率比10-20 Mb大片段低30%(F1-score)
2. 生物学复杂性挑战
-
肿瘤异质性
稀有亚克隆(<5%细胞占比)检测困难,算法倾向于识别主导克隆
-
基质干扰
肿瘤纯度<50%时准确性下降30%,需联合病理注释优化参考细胞选择
总结
本研究首次证明iST数据可用于CNV推断,填补了单细胞空间基因组学的技术空白。其提出的平滑-聚类-空间映射流程为肿瘤异质性研究提供了新工具,尤其适用于探索克隆空间竞争、微环境互作等前沿问题。
参考文献
- Augusta Jensen, Helena L. Crowell, Anna Pascual Reguant, Irene Ruano, Sabine Tejpar, Holger Heyn, Mats Nilsson, Sergio Marco Salas bioRxiv 2025.07.02.662761; doi: https://doi.org/10.1101/2025.07.02.662761
作者在github开源了代码,接下来我们准备使用Xenium5k数据进行测试: