一、 为什么光谱分析需要"可解释性"?
光谱数据不仅是数字,更是物理和化学信息的载体。一个典型的光谱图包含成百上千个波长点,它们之间存在着复杂的共线性。传统的偏最小二乘法(PLS)虽然线性可解释,但在处理复杂混合物时往往力不从心。而神经网络、支持向量机等"黑盒"模型虽然精准,却容易捕捉到背景噪声或仪器漂移等虚假特征。
如果一个模型告诉你这批苹果很甜,但它是基于果皮上的光泽(可能打了蜡)而非果肉内部的糖分吸收峰做出的判断,那么这个模型就是不可靠的。SHAP 和 LIME 的任务,就是揭示模型决策背后的"化学逻辑",确保模型真的是在"看"化学键,而不是在"猜"噪声。
二、 LIME:局部观察者
LIME的策略非常巧妙,它不试图理解整个复杂模型的全局逻辑,而是专注于"当下"这一个样本。
想象一下,我们有一个用于检测橄榄油是否掺假的深度学习模型。当我们输入一个疑似掺假的样本光谱时,LIME 会在这个样本周围生成许多"扰动"样本(比如微调某些波段的吸光度),然后观察模型对这些扰动样本的预测变化。
LIME 就像是在黑暗的房间里打开了一束聚光灯。它告诉我们:"对于这个特定的橄榄油样本,模型极其关注 1400-1500 nm 这一小段波长。" 如果这一波段恰好对应水分子的 O-H 键伸缩振动,而纯橄榄油几乎不含水,那么化学家就能立即警觉:模型可能是在通过检测水分来判定掺假,这符合化学常识。LIME 将复杂的非线性决策边界在局部简化为一个简单的线性模型,让光谱分析师能一眼看穿模型在局部的"小心思"。
应用1. 识别"伪特征"与模型去噪
有时候模型在验证集上表现很好,但 LIME 可能会揭示它是"蒙对的"。
- 场景: 在拉曼光谱分类细菌时,模型准确率很高。但通过 LIME 对某个样本进行解释,发现模型判定其为"大肠杆菌"的主要依据竟然是光谱基线漂移产生的某个背景斜率,而不是细菌特有的指纹峰。
- 反向优化: 这种"事后发现"立刻指出了预处理的缺陷。它告诉我们需要重新回到预处理阶段,加强基线校正算法,或者在特征选择时强制剔除这段无意义的基线区域,从而迫使模型去学习真正的化学特征。
应用2. 边界样本的精细化处理
在光谱分类问题的决策边界附近,样本往往最难区分。
- 应用: 利用 LIME 分析那些处于分类边界的模糊样本。如果 LIME 显示,对于这些难分样本,模型极其依赖某几个特定的微弱波段,那么我们可以推断这几个波段是区分相似物质(如异构体)的关键。
- 策略: 在下一轮特征工程中,我们可以专门针对这几个微弱波段进行局部放大 或加权处理,甚至专门增加针对这些波段的信噪比增强算法,从而提升模型对疑难样本的识别能力。
三、 SHAP:公平分配大师
SHAP 源于合作博弈论,旨在计算每个特征(波长点)对最终预测结果的边际贡献。
比如,在土壤重金属污染的光谱监测中,预测铅(Pb)含量是一个难题,因为铅本身在近红外区没有直接吸收峰,通常是依靠与土壤有机质或铁氧化物的关联来间接反演。使用 XGBoost 等集成学习模型可以获得很好的预测效果,但很难解释。
巧妙之处:
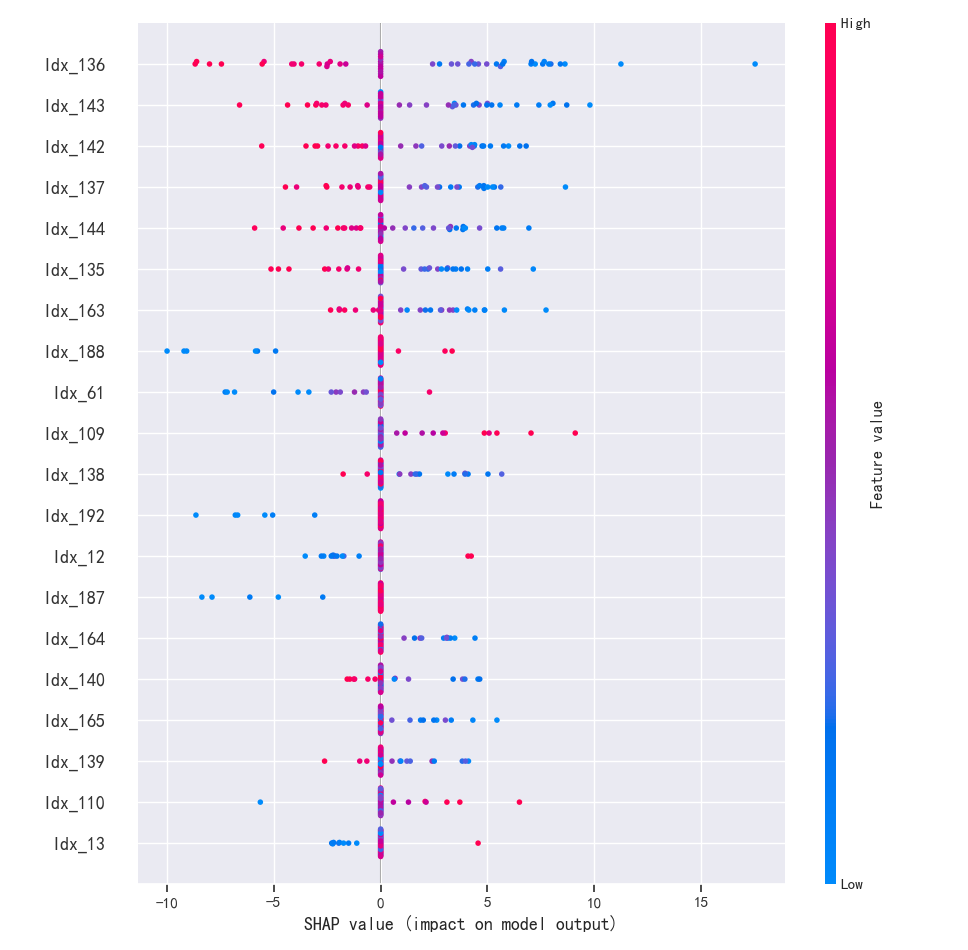
SHAP 值能绘制出一幅精美的**"特征贡献图"**。它不仅能告诉我们哪些波长最重要(比如与铁氧化物相关的波段),还能展示正负相关性:
- 红色点(高SHAP值):表示该波段吸光度越高,模型预测的铅含量越高。
- 蓝色点(低SHAP值) :表示该波段吸光度越低,预测值越低。
SHAP 的巧妙在于它的全局一致性 和局部精确性的统一。它能让分析师看到,模型虽然没有直接"看到"铅,但它确实紧紧抓住了与铅伴生的铁氧化物的光谱特征(如 700-900 nm)。这不仅验证了模型的可靠性,甚至可能启发科学家发现新的地球化学关联机制。
应用:迭代式特征波段筛选
SHAP 基于博弈论,能够计算出每一个特征(波长点)对模型预测结果的平均边际贡献。这种全局视角的"审计报告",是进行波段优选的绝佳依据,虽然是"事后分析",提取特征波段是在分析之前,但是如果用于模型鲁棒性分析,未来批次预测的话可以使用。
传统的波段选择方法(如竞争性自适应重加权采样 CARS)虽然有效,但往往依赖于随机采样。SHAP 提供了一种确定性的筛选逻辑:
- 第一步(训练全谱模型): 首先,我们将全波段光谱数据输入到非线性模型(如 XGBoost 或 LightGBM)中进行训练。
- 第二步(计算贡献度): 训练完成后,利用 SHAP 计算所有波长点的平均绝对 SHAP 值。这幅图谱实际上就是模型眼中的"光谱指纹"。
- 第三步(末位淘汰与重塑): 依据 SHAP 值排序,剔除那些贡献度极低甚至为零的波段(往往是噪声区间或无信息区间)。
- 第四步(闭环验证): 这才是关键。 我们仅保留高 SHAP 值的特征波段,重新训练一个新的简化模型。
四、代码实现
在预测任务中进行SHAP与LIME分析的代码如下:
python
import os
import matplotlib
matplotlib.use('TkAgg') # 保持您的设置
import csv
import numpy as np
import matplotlib.pyplot as plt
from sklearn.metrics import mean_squared_error, r2_score
from sklearn.cross_decomposition import PLSRegression
from scipy.signal import savgol_filter
import seaborn as sns
from BaselineRemoval import BaselineRemoval
# === 新增:引入解释性分析库 ===
import shap
import lime
import lime.lime_tabular
# 设置绘图样式
sns.set(context='notebook', style='darkgrid', palette='deep', font='sans-serif', font_scale=1, color_codes=False)
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
# 数据预处理函数
def preprocess_spectrum(spectrum_data, sg_w, sg_n, sg_d, lambda_, porder, norm_wavenumber, start_idx, end_idx):
# ....
# 读取数据函数
def read_data(file_path, sg_w, sg_n, sg_d, lambda_, porder, start_idx, end_idx, norm_wavenumber):
X = []
Y = []
if not os.path.exists(file_path): # 增加一点安全性
return np.array([]), np.array([])
with open(file_path, mode='r', newline='', encoding='ISO-8859-1') as file:
reader = csv.reader(file)
header = next(reader)
previous_od_value = None
current_spectra = []
for row in reader:
try: # 增加异常捕获防止坏行
spectrum_data = [float(val) for val in row[3:3 + 1024]]
od_value = float(row[1]) # OD值
spectrum_segment = spectrum_data[start_idx:end_idx]
preprocessed_spectrum = preprocess_spectrum(
spectrum_segment, sg_w, sg_n, sg_d, lambda_, porder, norm_wavenumber, start_idx, end_idx)
if previous_od_value is None or od_value != previous_od_value:
if current_spectra:
mean_spectrum = np.mean(current_spectra, axis=0)
X.append(mean_spectrum)
Y.append(previous_od_value)
current_spectra = [preprocessed_spectrum]
else:
current_spectra.append(preprocessed_spectrum)
previous_od_value = od_value
except Exception:
continue
if current_spectra:
mean_spectrum = np.mean(current_spectra, axis=0)
X.append(mean_spectrum)
Y.append(previous_od_value)
return np.array(X), np.array(Y)
# 训练集和测试集路径
train_file_paths = [
r"D:\pythonProject\KangMa\Data\LabeledData\LabeledData0807.csv",
r"D:\pythonProject\KangMa\Data\LabeledData\LabeledData0813.csv",
r"D:\pythonProject\KangMa\Data\LabeledData\LabeledData0907.csv",
r"D:\pythonProject\KangMa\Data\LabeledData\LabeledData0920.csv",
r"D:\pythonProject\KangMa\Data\LabeledData\LabeledData1012.csv"
]
test_file_paths = [
r"D:\pythonProject\KangMa\Data\LabeledData\LabeledData0820.csv",
r"D:\pythonProject\KangMa\Data\LabeledData\LabeledData0828.csv",
]
def main():
# 直接使用给定的最佳参数
best_params = {
'sg_w': 47,
'sg_n': 14,
'sg_d': 1,
'lambda_': 76.19968486314983,
'porder': 42,
'start_idx': 169,
'end_idx': 363,
'n_components': 9,
'norm_wavenumber': 1645.412436 # 保持与训练时一致
}
# 读取训练数据并预处理
X_train, Y_train = np.empty((0, best_params['end_idx'] - best_params['start_idx'])), np.empty((0,))
for fpath in train_file_paths:
X, Y = read_data(
fpath,
best_params['sg_w'], best_params['sg_n'], best_params['sg_d'],
best_params['lambda_'], best_params['porder'],
best_params['start_idx'], best_params['end_idx'],
best_params['norm_wavenumber']
)
if X.size > 0:
X_train = np.vstack([X_train, X]) if X_train.size else X
Y_train = np.concatenate([Y_train, Y]) if Y_train.size else Y
# 读取测试数据并预处理
X_test, Y_test = np.empty((0, best_params['end_idx'] - best_params['start_idx'])), np.empty((0,))
for fpath in test_file_paths:
X, Y = read_data(
fpath,
best_params['sg_w'], best_params['sg_n'], best_params['sg_d'],
best_params['lambda_'], best_params['porder'],
best_params['start_idx'], best_params['end_idx'],
best_params['norm_wavenumber']
)
if X.size > 0:
X_test = np.vstack([X_test, X]) if X_test.size else X
Y_test = np.concatenate([Y_test, Y]) if Y_test.size else Y
# 训练PLS模型
model = PLSRegression(n_components=best_params['n_components'])
model.fit(X_train, Y_train)
# 预测
Y_pred_train = model.predict(X_train)
Y_pred_test = model.predict(X_test)
# 计算误差指标
mse_train = mean_squared_error(Y_train, Y_pred_train)
r2_train = r2_score(Y_train, Y_pred_train)
mse_test = mean_squared_error(Y_test, Y_pred_test)
r2_test = r2_score(Y_test, Y_pred_test)
print(f"训练集MSE: {mse_train:.6f}, R²: {r2_train:.6f}")
print(f"测试集MSE: {mse_test:.6f}, R²: {r2_test:.6f}")
# ==============================================================================
# 绘图部分
# ==============================================================================
fig1 = plt.figure(figsize=(14, 14))
indices_train = np.arange(len(Y_train))
indices_test = np.arange(len(Y_test))
plt.subplot(2, 1, 1)
plt.plot(indices_train, Y_train, 'o-', label='训练集真实值', color='navy', alpha=0.7, markersize=10)
# 注意:这里也加了 flatten() 确保绘图兼容性,虽然 plt 通常能自动处理
plt.plot(indices_train, Y_pred_train.flatten(), 'o-', label='训练集预测值', color='orange', alpha=0.7, markersize=7)
plt.ylabel('值')
plt.legend()
plt.title('训练集真实值与预测值对比')
plt.grid(True)
plt.subplot(2, 1, 2)
plt.plot(indices_test, Y_test, 'o-', label='测试集真实值', color='navy', alpha=0.7, markersize=10)
plt.plot(indices_test, Y_pred_test.flatten(), 'o-', label='测试集预测值', color='orange', alpha=0.7, markersize=7)
plt.ylabel('值')
plt.legend()
plt.title('测试集真实值与预测值对比')
plt.grid(True)
# 这里不加 plt.show(),防止程序阻塞在这里,我们希望跑完后面的分析一起看
# ==============================================================================
# 新增:SHAP 和 LIME 分析模块 (Post-hoc Analysis)
# ==============================================================================
print("\n正在进行模型解释性分析 (SHAP & LIME)...")
# 构造特征名称 (对应波长索引)
feature_names = [f"Idx_{i}" for i in range(X_train.shape[1])]
# ------------------------------------------------------------------------------
# 1. SHAP (Global & Local Interpretation)
# ------------------------------------------------------------------------------
print(" -> 正在运行 SHAP 分析...")
# 为了加快速度,我们对训练数据进行 K-Means 聚类,只取 10 个代表性背景样本
# 如果数据量不大,可以直接用 X_train,但光谱数据通常建议聚类摘要
X_train_summary = shap.kmeans(X_train, 10)
# 定义预测包装函数,SHAP 需要 (n_samples, ) 的输出
def model_predict_wrapper(x):
return model.predict(x).flatten()
explainer_shap = shap.KernelExplainer(model_predict_wrapper, X_train_summary)
# 对测试集前 50 个样本进行解释 (全量计算太慢)
shap_samples_idx = min(50, len(X_test))
shap_values = explainer_shap.shap_values(X_test[:shap_samples_idx])
# SHAP Summary Plot (特征重要性排序)
# 这会创建一个新的 Figure
plt.figure()
plt.title("SHAP 摘要图 (特征重要性排序)")
shap.summary_plot(shap_values, X_test[:shap_samples_idx], feature_names=feature_names, show=False)
plt.tight_layout()
# ------------------------------------------------------------------------------
# 2. LIME (Local Interpretation for a specific instance)
# ------------------------------------------------------------------------------
print(" -> 正在运行 LIME 分析...")
explainer_lime = lime.lime_tabular.LimeTabularExplainer(
training_data=X_train,
feature_names=feature_names,
mode='regression',
verbose=False
)
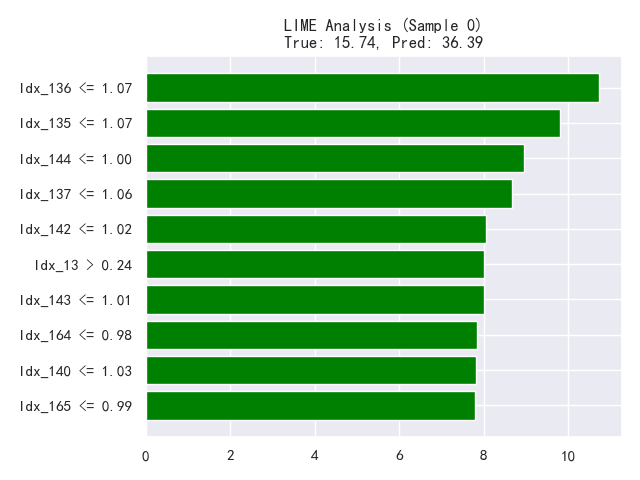
# 选择一个测试样本进行详细分析 (例如索引 0)
idx_lime = 0
if len(X_test) > idx_lime:
exp = explainer_lime.explain_instance(
data_row=X_test[idx_lime],
predict_fn=model_predict_wrapper,
num_features=10 # 只展示前 10 个最重要的影响波段
)
# 绘制 LIME 图
fig_lime = exp.as_pyplot_figure()
# === 修复点:安全获取预测值 ===
# 使用 float() 强制转换,无论 Y_pred_test 是标量、(N,1) 还是 (N,) 都能正常工作
pred_val = float(Y_pred_test[idx_lime])
true_val = float(Y_test[idx_lime])
plt.title(f"LIME Analysis (Sample {idx_lime})\nTrue: {true_val:.2f}, Pred: {pred_val:.2f}")
plt.tight_layout()
print("分析完成,正在显示结果...")
# === 最终统一显示 ===
# 这行代码会阻塞程序,直到手动关闭所有弹出的窗口
plt.show()
if __name__ == "__main__":
main()