由于多数疾病的产生与蛋白质功能异常直接相关,蛋白质在药物研发领域发挥着关键作用。研究人员在研发新药时,通常会将蛋白质作为核心药物靶点,使药物与部分结构稳定的蛋白质相结合以干预疾病进程。然而,将药物靶向缺乏明确结构、序列和构象偏好的天然无序蛋白(IDPs)仍然存在挑战。
利用抗体靶向的传统方法主要基于抗体对特定蛋白质的高度特异性结合能力,实现对目标蛋白质的识别和调控。但该靶向路径不仅需要进行大量实验操作,而且无序抗原极易在注射后降解失效。因此,占蛋白质组 50% 以上的天然无序区域(IDR)的蛋白质通常被判定为「不可用药」靶点,从未被用于药物开发。
在此背景下,荣获 2024 年诺贝尔化学奖的杰出计算生物学家、华盛顿大学蛋白质设计研究所所长 David Baker 及其团队提出了一种名为 Logos 的蛋白质设计策略,基于诱导契合(Induced Fit)的结合策略,设计了能够适应 39 种目标无序氨基酸序列的结合蛋白。 该研究在生成专门的扩展重复蛋白质骨架后,使用 RFdiffusion 模型进行推广。其中,骨架带有专门用于重复肽序列的口袋,使设计好的结合物-靶肽模版能够实现对无序蛋白质区域的通用识别。这意味着更多蛋白质能为新药研发提供靶点,有望加快癌症和阿尔兹海默症的研究。
相关研究成果以「Design of intrinsically disordered region binding proteins」为题,发表在 Science 。
研究亮点:
-
建立适合一般识别的模版结构库,实现对任何靶序列的结合适配构象诱导。
-
为 18 条合成肽段序列和 21 个具有广泛多样性与治疗潜力的天然无序区段(IDRs)设计结合蛋白,能够靶向与癌症有关的细胞外受体的无序区域并驱动蛋白质在细胞内的定位。
论文地址:
www.science.org/doi/10.1126...
关注公众号,后台回复「天然无序蛋白」获取完整 PDF
更多 AI 前沿论文:
模版库生成:通用肽识别
该研究结合物理设计方法和深度学习设计方法作为 IDRs 结合问题的解决方案。受限于肽单元与异质目标序列的不兼容性,研究从不同的重复蛋白结构入手,利用扩散模型将不同重复单元中的氨基酸结合口袋重组,并分化为不同的氨基酸及构象模版, 从而实现对序列的更广泛识别。
为识别天然无序蛋白中的肽,研究首先创建了骨架模版库。模版库具有两个特性:
-
每一个模板结构应能够「包裹」住拉伸态的肽链构象,并提供大量氢键和紧密堆积等相互作用的机会,从而实现对目标序列的高度特异性识别。
-
模版结构广泛,能够匹配任何靶序列,使至少一种模版能够诱导其成为确定、适配的结合构象。
生成骨架模版库的流程分为三个步骤:骨架生成、蛋白质活性口袋特化和蛋白质活性口袋组装。
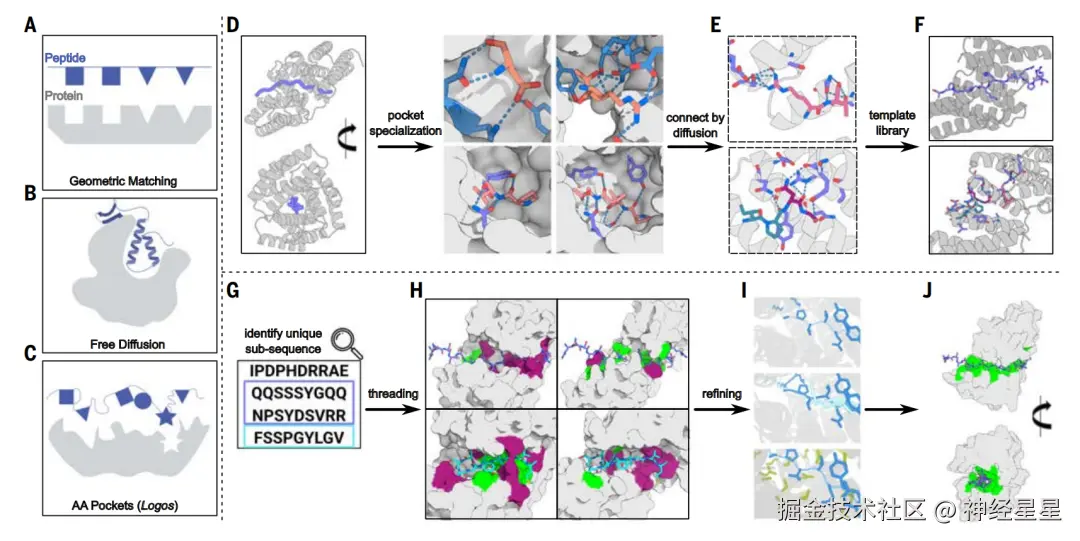
IDR 结合蛋白设计流程概述
骨架生成(Scaffold Generation)
在骨架生成阶段,研究团队选择以多种拉伸构象(extended conformations)为目标,而非局限于 polyproline II 构象,因为 polyproline II 构象主要出现在富含脯氨酸的肽段中。
在拉伸构象中,氨基酸的侧链会交替朝向相反的方向,这与双残基序列重复(two-residue repeat)的特征是一致的。所以,研究人员使用 Rosetta 设计方法,针对一系列双肽重复序列进行设计, 包括 LK 、 RT 、 YD 、 PV 和 GA(均为氨基酸单字母缩写),设计其在不同的拉伸构象下与这些肽段进行缠绕结合,使得每一个重复单元与一个双肽单元发生相互作用。
随后,研究人员通过荧光偏振实验对这些设计出的四重复单元版本的结合蛋白进行表征,结果显示:对于 LK 和 PV 重复肽段表现出纳摩尔级的结合能力;但对于更极性的 RT 和 YD 结合能力较弱,而对于高度柔性的 GA 则完全未检测到结合信号。
结合口袋特异化(Pocket Specialization)
在蛋白质活性口袋特异化步骤,研究人员利用扩散模型对口袋进行微调,以实现与特定目标肽序列更精确的匹配。
为提升模版匹配效率,研究改进了设计的结合口袋,在提高与目标序列匹配度的同时将相互作用的重复单元数量从 4 个增加至 5 个,以提高目标结构间的亲和力。将重复蛋白与肽骨架之间每个侧链双叉氢键周围的 4 到 9 个氨基酸保持固定,同时对设计结合蛋白之间的疏水相互作用进行多样化调整。
这种策略的优势在于,氢键的几何构型要求更严格,相比之下非极性疏水堆积的空间自由度更高,因此在设计中,与其从头反复采样氢键,不如直接以模板方式保留氢键更为高效。
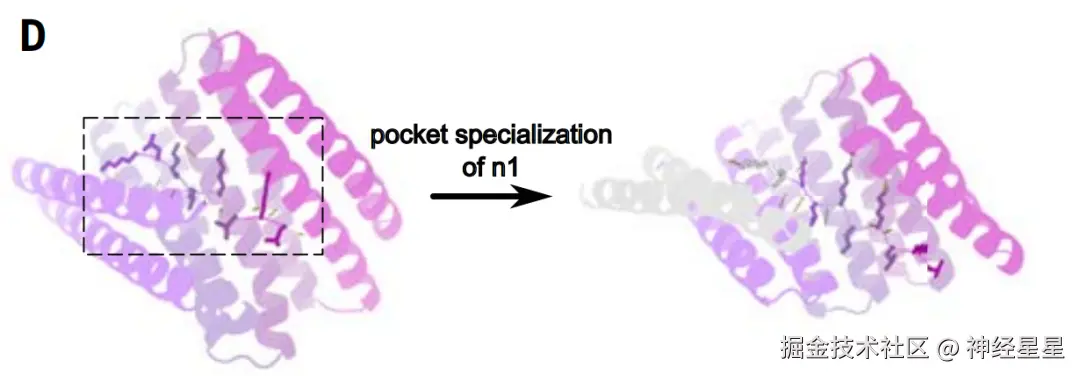
结合口袋特异化示例,优化并扩展了一个原本完全重复的四重骨架(左),生成了一个新的五重骨架(右)
新扩展的第五个重复结构以浅灰色显示
蛋白质结合口袋组装(Pocket Assembly)
在口袋组装步骤中,研究人员利用 RFdiffusion 模型在口袋之间创造接口,从而产生整体刚性结构并生成模版,将结合口袋组装到新的骨架中。 模版中的各种口袋根据不同顺序和几何形状排列,以在连续的扩展构象中与肽靶相互作用,对非重复序列进行更普遍的识别。
研究在得到与嵌合肽靶相互作用的嵌合蛋白模型后,对结合口袋进行了参数定位,并通过射频扩散将它们连接起来。研究使用该方法针对 7 个嵌合靶标生成了 70 个设计方案。从分离荧光素酶充足法和生物层干涉测量法实验的表征来看,在平均每个靶点仅测试了 10 个设计的情况下,7 个靶点中有 6 个点结合率达到了两位数纳摩尔。
为扩大模版库的规模以涵盖更广泛的序列,研究利用口袋组装技术构建了包含识别极性残基口袋的 36 个嵌合骨架,并生成了 1,000 个由设计好的结合蛋白和一个相应肽骨架组成的模版,其中,肽构象中的氨基酸能够与结合蛋白中设计好的口袋相匹配。
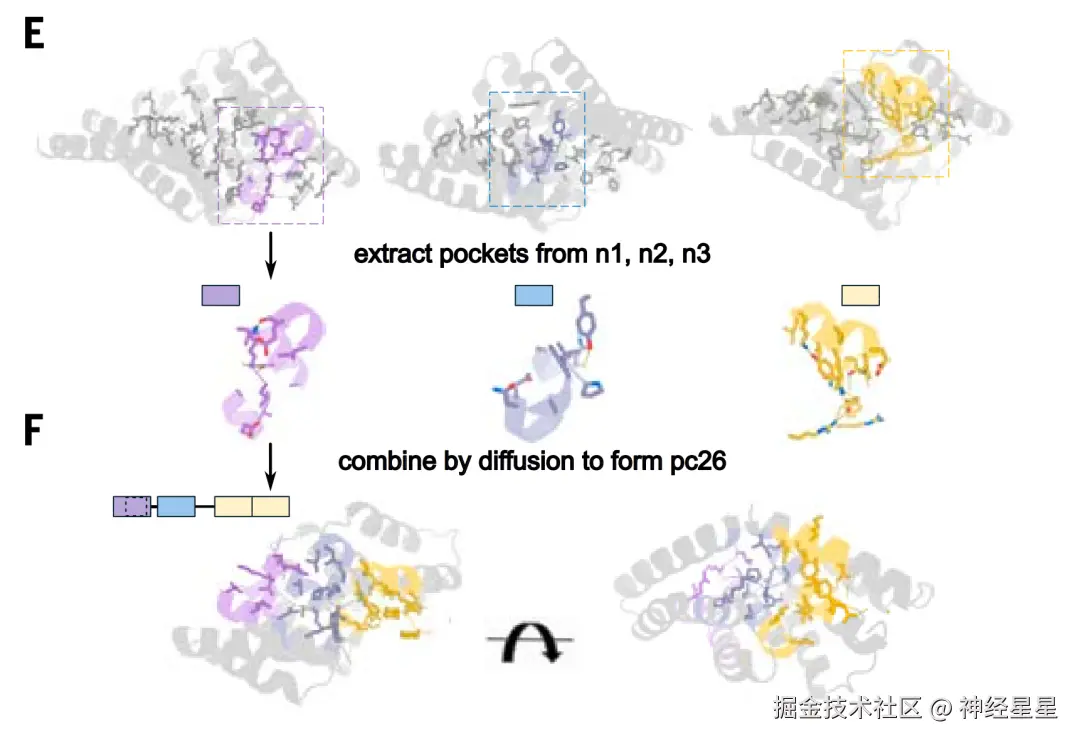
口袋组装实例
IDR 结合蛋白设计与优化
在建立模版库后,研究人员在模版库中插入天然无序区,利用模板库生成可结合非重复合成序列和任意天然无结构靶标的结合蛋白。该步骤分为线程(threading)匹配和结构优化(refinement)两部分。
线程匹配:确定最兼容的序列片段-模版对
在线程匹配中,研究将将目标序列穿入每个模版的骨架中,以识别最兼容的序列片段与模板配对。
一般来说,IDP 或 IDR 有大量可能的肽段可以作为靶标。为了找出 IDR 中最具靶向潜力的肽段,研究首先剔除了序列复杂度低的肽段和在蛋白质组中有多个近似匹配的肽段,以免此类靶标的结合剂产生交叉反应。 在将剩余氨基酸的独特序列片段映射到模版库的靶标骨架进行局部骨架重采样后,研究使用基于深度学习的蛋白质序列设计工具 ProteinMPNN 对结合蛋白的序列进行优化,并根据设计的结合蛋白与靶标序列之间的拟合度以及 AF2 预测值与模型之间的一致性进行评估。
在 AF2 指标不理想的情况下,继续使用 RFdiffusion 为特定目标定制骨架。随后,研究采用线程匹配为治疗相关的 IDP 、 IDR 和 IDP 片段生成结合体,每个靶标平均生成 28 种设计。
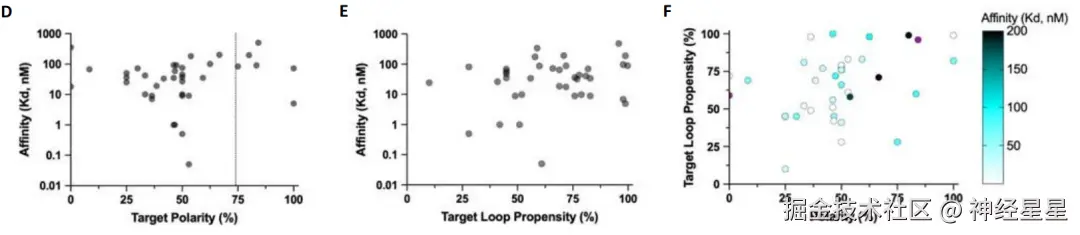
目标极性、最高亲和力与目标环倾向性关系
结构优化:提高结合蛋白与目标肽的匹配度
研究还对最佳匹配进行了优化,以增强设计的结合蛋白与目标肽之间的匹配度。研究选择了 DYNA_1b1 结合蛋白与强啡肽的解离常数进行测试,对合成靶标的最高命中率进行了射频扩散优化。结果显示,在 48 组设计中,有 45 组在筛选试验中显示出强亲和力,仅有 6 组设计的解离常数显示出弱亲和力。
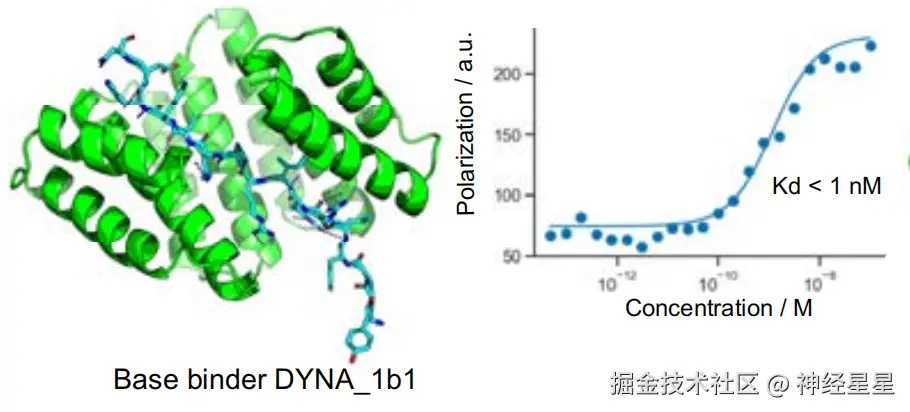
DYNA_1b1 扩展骨架构象与强啡肽 A 结合蛋白的设计模型
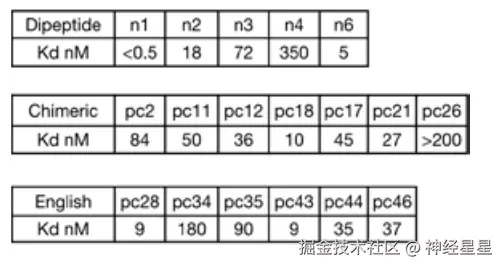
通过生物层干涉测量法测得的同源设计的结合蛋白-目标解离常数
强啡肽结构与结合蛋白正交性的有效性验证
为验证强啡肽结构在结合时的变化,研究检测了同位素标记的强啡肽 A 在溶液中未结合时、与 DYNA_1b1 结合时、与亲和力更高的 DYNA_2b2 结合时的核磁共振(NMR)光谱。
从核磁共振结果来看,游离的强啡肽 A 内在无序,但设计骨架包含的区域在结合后转变为有序。对于两种结合的复合物,核磁共振数据显示出扩展的结合态构象,与设计模型一致,证实了强啡肽作用在诱导无序蛋白和多肽进入非原生构象方面的有效性。
为了探索 Logos 的优化潜力,研究人员选择了一个与 dynorphin 结合的 binder------DYNA_1b1,其对 dynorphin 的结合常数(Kd)约为 1 nM 。研究人员基于 RFdiffusion 对排名靠前的设计进行了优化,在 48 个设计中,有 45 个在 5 nM 浓度下通过 BLI 筛选实验表现出强结合能力,其中有 6 个的 Kd 值通过 BLI 测得小于等于 100 pM;对其中两个优化设计(DYNA_2b1 和 DYNA_2b2)进行荧光偏振测量,结果表明它们的 Kd 分别低于 60 pM 和 100 pM,如下图 B 所示。
注:Dynorphin 是一种 κ-阿片受体(KOR)肽配体,与慢性疼痛相关。
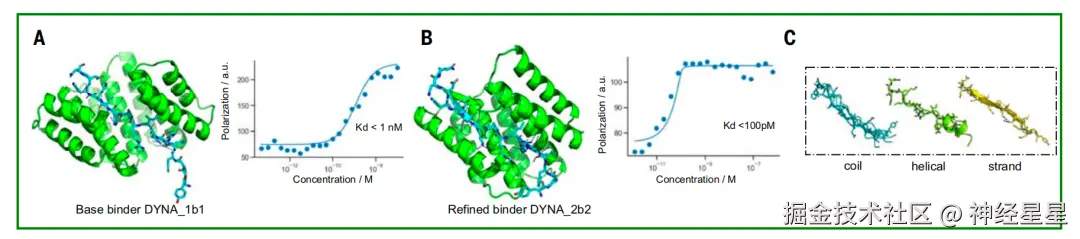
Dynorphin A 结合蛋白设计的结构特征分析
在 dynorphin A 的原始设计和优化设计中,该肽呈现出多种构象,包括无规卷曲、部分 β-链结构和部分 α-螺旋结构,如上图 C 所示。尽管 dynorphin A 和 B 的序列相似性达 62%,但它们各自的结合蛋白是互不交叉的,仅与各自的靶标结合。同时,与 dynorphin A 结合的设计蛋白 DYNA_1b7 的共晶结构与计算设计模型高度吻合,尤其是在核心结合界面处(上图 D-E)。 NMR 数据也进一步确认,原本无序的 dynorphin A 在与设计蛋白结合后,其骨架变得有序,再次印证了诱导契合机制的有效性(上图 F)。
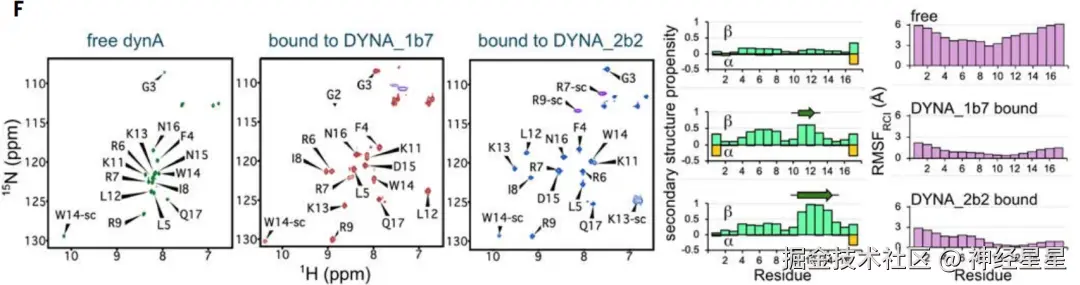
同位素标记的游离强啡肽 A 结合图谱
验证结合蛋白的功能性及正交性
研究采用 WASH 复合物及 PER 复合物为模型进行了免疫沉淀研究。其中,WASH 复合体包括 WASH 、 FAM21 、 CCDC53 、 SWIP 和 WASHC2 。测试显示,FAM21_1b1 从细胞裂解物中提取整个 WASH 复合物。
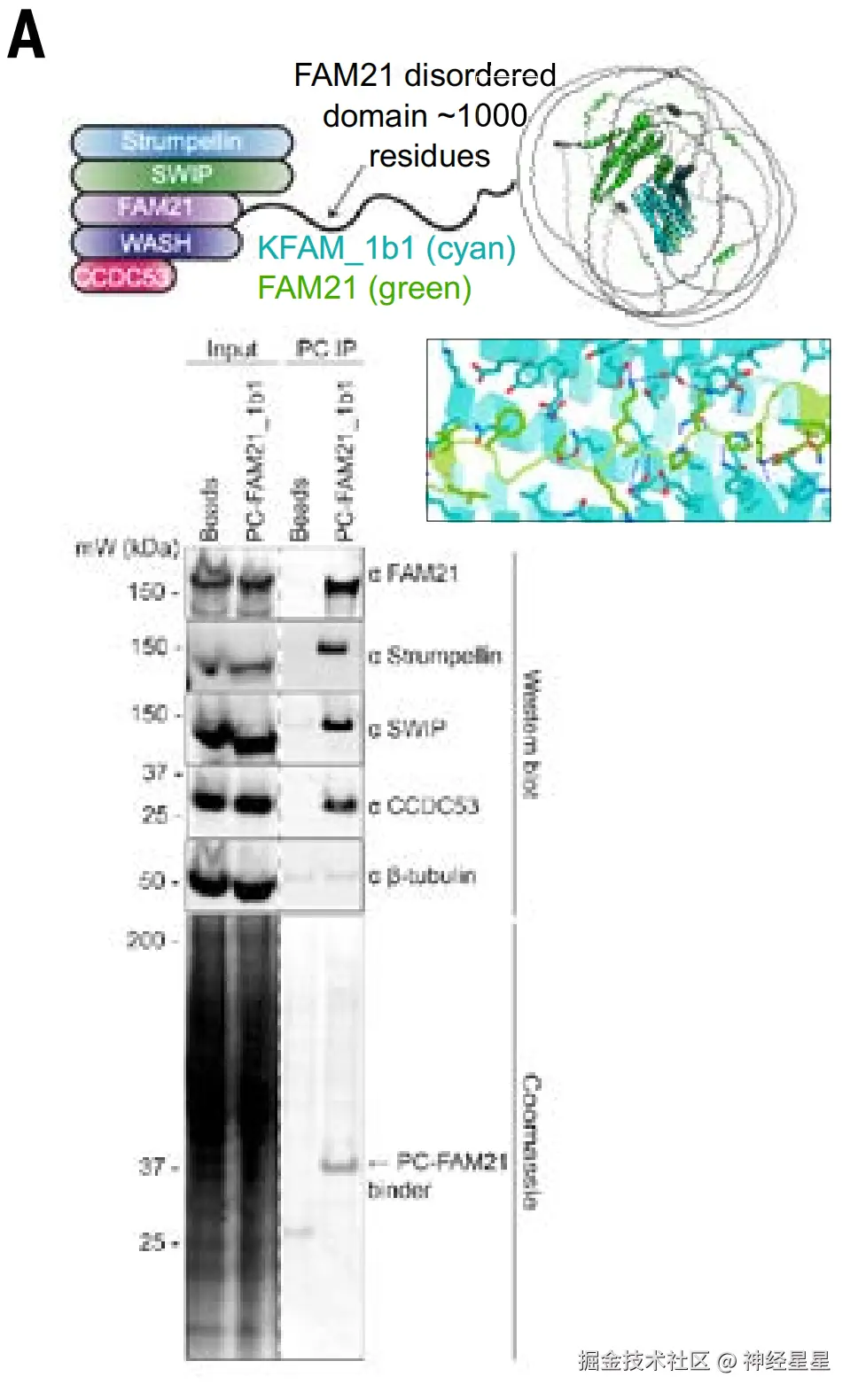
WASH 复合物包含带有长无序尾部的 FAM21 蛋白
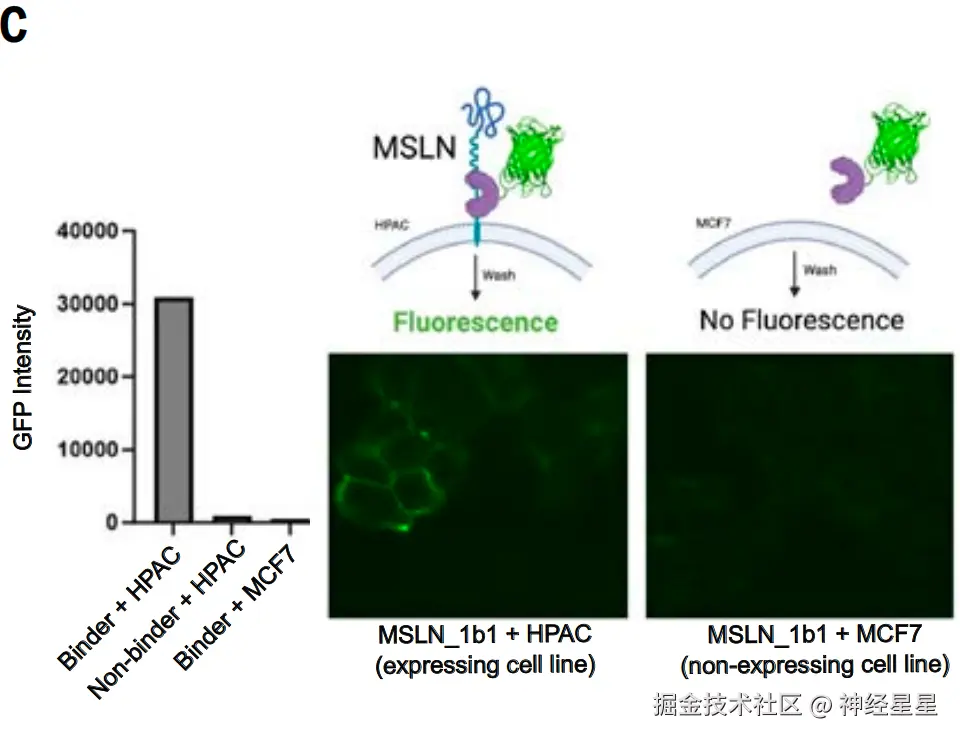
此外,其还研究了一种针对 MSLN 膜邻近区域(Juxtamembrane Region)设计的结合蛋白(MSLN_1b1),是否能够特异性结合表达该靶标的细胞(由于该区域的蛋白酶切割使得更远端的胞外结构域区域不太适合作为靶点)。
注:间皮素(Mesothelin,MSLN)是一种细胞表面糖蛋白,在多种癌症中被上调表达,因此在肿瘤靶向治疗中备受关注。
研究人员将绿色荧光蛋白(GFP)与 MSLN_1b1 融合,并与表达 MSLN 的细胞(人胰腺腺癌细胞系 HPAC)以及不表达 MSLN 的细胞系(密歇根癌症基金会乳腺癌细胞系 MCF7)共同孵育,同时设有一个不结合 MSLN 的 GFP-融合蛋白作为对照。
荧光显微镜显示,在 HPAC 细胞上,GFP-MSLN_1b1 融合蛋白在细胞连接处聚集,符合 MSLN 的定位特征;而在 MCF7 对照细胞中则没有观察到此现象。同时,对照结合蛋白在 HPAC 细胞中也未显示出结合信号,如下图 C 所示。因此,MSLN_1b1 能够特异性地识别并结合细胞表面的 MSLN 。
AI 驱动,解锁蛋白质靶向未来新图景
目前,AI 已经越来越广泛地参与蛋白质靶向研究,推动研究进入「多技术并行」的新阶段。除 David Baker 团队之外,宾夕法尼亚大学 George M. Burslem 、 Ophir Shalem 团队也在靶向蛋白的研究领域也实现了革命性突破。该团队在研究中提出了「蛋白质编辑」技术,成功利用分离内肽(split intein)系统,实现了在活体哺乳动物细胞中直接修改蛋白质合成后的氨基酸序列,首次实现在内源性蛋白中精准植入非标准氨基酸和化学标记(生物素、荧光团)。相关研究成果以「Intracellular protein editing enables incorporation of noncanonical residues in endogenous proteins」为题,发表在 Science 。
论文地址:
https://www.science.org/doi/10.1126/science.adr5499
此外,中国科学院遗传与发育生物学研究所高彩霞、华中农业大学李国田领衔的中外团队还引用了 David Baker 团队的蛋白质设计工作,开发了基于逆折叠模型的通用蛋白质工程方法 AiCE,基于 AI 驱动的蛋白质设计策略,成功优化了脱氨酶、核酸酶等 8 类蛋白质,并开发出新型碱基编辑器。研究论文以「Advancing protein evolution with inverse folding models integrating structural and evolutionary constraints」为题,发表在 Cell 。
论文地址:
https://www.cell.com/cell/abstract/S0092-8674(25)00680-4
从活细胞编辑到神经保护疗法,从糖基化创新到 AI 多链设计,随着 AI 在生物医学领域的持续发展,全球团队正以前所未有的多样化路径,攻克天然无序蛋白背后的生物医学难题。研究团队对天然无序区域靶向问题的探索,未来将为攻克癌症、阿尔茨海默病等疾病创造新的治疗路径。
参考链接: