ArchR TSS QC 绘图学习笔记
01 背景
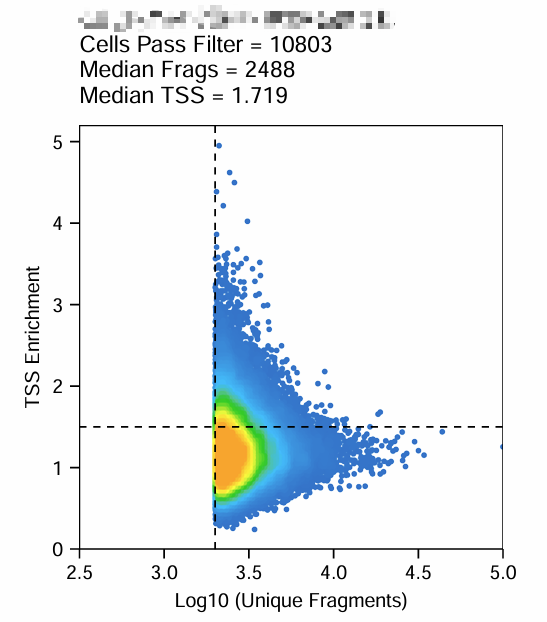
在单细胞 ATAC-seq 数据分析中,TSS(Transcription Start Site)富集度 和 Unique Fragments 是衡量每个细胞质量的重要指标。
- Unique Fragments (
nFrags):单细胞测序中每个细胞独特的插入片段数量。片段数量过低的细胞可能质量差。 - TSS Enrichment (
TSSEnrichment):每个细胞在转录起始位点附近的信号富集程度。高富集度通常说明染色质结构良好、信号清晰。
ArchR 的 QC 流程中会生成每个样本的 TSS_by_Unique_Frags.pdf 图,用于:
-
可视化细胞质量分布
- 横轴:Log10(unique fragments)
- 纵轴:TSS enrichment
-
设定细胞过滤阈值
- 可以通过虚线查看哪些细胞满足过滤标准:
nFrags >= 2000,TSS >= 1.5
- 可以通过虚线查看哪些细胞满足过滤标准:
在 ArchR 官方的
CreateArrowFiles()函数里,这个 PDF 是自动生成的。如果你的运行没有生成,可以参考源码,自行生成。链接:ArchR GitHub
02 完整代码
r
library(ArchR)
library(ggplot2)
#-------------------- 路径设置 --------------------
qc_dir <- "xxxxx/02.qc/QualityControl"
out_dir <- file.path(qc_dir, "TSS_QC_Plots")
dir.create(out_dir, showWarnings = FALSE)
#-------------------- 匹配RDS文件 --------------------
rds_files <- list.files(
qc_dir,
pattern = "Pre-Filter-Metadata\\.rds$",
recursive = TRUE,
full.names = TRUE
)
cat("找到的RDS文件:\n")
print(rds_files)
#-------------------- 设置过滤阈值 --------------------
filterFrags <- 2000
filterTSS <- 1.5
#-------------------- 循环读取并画图 --------------------
for (rds_path in rds_files) {
# 读取 Metadata
Metadata <- readRDS(rds_path)
sampleName <- gsub("_Pre-Filter-Metadata\\.rds$", "", basename(rds_path))
message("正在处理样本:", sampleName)
# 根据最小片段数量过滤
Metadata <- Metadata[Metadata$nFrags >= 2000, ]
# 计算 Keep 标记(满足过滤条件的细胞)
Metadata$Keep <- (Metadata$nFrags >= filterFrags & Metadata$TSSEnrichment >= filterTSS)
nKeep <- sum(Metadata$Keep)
medFrags <- median(Metadata$nFrags[Metadata$Keep])
medTSS <- median(Metadata$TSSEnrichment[Metadata$Keep])
#-------------------- 输出 PDF --------------------
pdf_path <- file.path(out_dir, paste0(sampleName, "-TSS_by_Unique_Frags.pdf"))
keep_idx <- which(Metadata$Keep)
pdf(pdf_path, width = 4, height = 4)
ggPoint(
x = pmin(log10(Metadata$nFrags), 5) + rnorm(length(Metadata$nFrags), sd = 1e-5),
y = Metadata$TSSEnrichment + rnorm(length(Metadata$nFrags), sd = 1e-5),
colorDensity = TRUE,
xlim = c(2.5, 5),
ylim = c(0, max(Metadata$TSSEnrichment[keep_idx], na.rm = TRUE) * 1.05),
baseSize = 6,
continuousSet = "sambaNight",
xlabel = "Log10 (Unique Fragments)",
ylabel = "TSS Enrichment",
title = paste0(sampleName, "\nCells Pass Filter: ", nKeep,
"\nMedian Frags: ", medFrags,
"\nMedian TSS: ", medTSS),
rastr = TRUE
) +
geom_hline(yintercept = filterTSS, lty = "dashed", size = 0.25) +
geom_vline(xintercept = log10(filterFrags), lty = "dashed", size = 0.25)
dev.off()
}
message("✅ 所有样本的 TSS QC 图已生成到: ", out_dir)
#-------------------- 示例:细胞类型可视化 --------------------
pdf("xxxxxx/03.cca/celltype.pdf",
width = 25, height = 20)
DimPlot(data, group.by = "all_celltype", raster = FALSE) +
sc_theme(colortype = 'discrete', label.size = 10, legend.size = 12, base_size = 20)
dev.off()03 代码解读
1. 路径与文件准备
r
qc_dir <- "...QualityControl"
out_dir <- file.path(qc_dir, "TSS_QC_Plots")
dir.create(out_dir)- 设置 QC 文件夹路径。
- 创建输出文件夹用于存储 PDF。
2. 匹配 RDS 文件
r
rds_files <- list.files(qc_dir, pattern = "Pre-Filter-Metadata\\.rds$", recursive = TRUE, full.names = TRUE)- 查找每个样本的 Pre-Filter Metadata。
- 每个 RDS 文件包含每个细胞的
nFrags和TSSEnrichment信息。
3. 循环处理每个样本
r
Metadata <- readRDS(rds_path)
sampleName <- gsub("_Pre-Filter-Metadata\\.rds$", "", basename(rds_path))- 读取 RDS。
- 提取样本名,便于命名 PDF。
4. 过滤细胞
r
Metadata$Keep <- (Metadata$nFrags >= filterFrags & Metadata$TSSEnrichment >= filterTSS)- 标记哪些细胞满足 QC 阈值。
- 计算通过过滤的细胞数
nKeep和中位数指标medFrags、medTSS。
5. 绘制 PDF
r
ggPoint(
x = pmin(log10(Metadata$nFrags), 5) + rnorm(...),
y = Metadata$TSSEnrichment + rnorm(...),
colorDensity = TRUE
)- 横轴:Log10(unique fragments)
- 纵轴:TSS enrichment
rnorm(..., sd = 1e-5):轻微抖动,避免点完全重叠。colorDensity = TRUE:高密度点显示为颜色梯度。- 添加虚线表示过滤阈值。
6. 输出图和消息
r
pdf(pdf_path, width = 4, height = 4)
dev.off()
message("✅ 所有样本的 TSS QC 图已生成到: ", out_dir)- 每个样本生成单独的 PDF。
- 最终提示所有样本已完成。
04 总结
-
为什么需要这个 PDF:
- 可视化每个样本中每个细胞的质量分布。
- 直观选择过滤阈值(fragment 数量和 TSS 富集)。
-
ArchR 官方做法:
CreateArrowFiles()内部会生成该 PDF。- 如果没有生成,可以通过源码逻辑自行绘制。
-
学习价值:
- 理解单细胞 ATAC QC 指标。
- 学习如何用
ggPoint绘制高密度散点图。 - 掌握批量处理多个样本的思路。