最近一段时间,我一直在探索 AI 蛋白质设计。起初只用 RFdiffusion 生成蛋白质骨架,但渐渐觉得"光有骨架还不够"------想要真正获得可表达、可折叠的功能性序列,还需要后续的序列设计与结构验证工具。
于是,我陆续安装并配置了 ProteinMPNN(用于序列设计)和 AlphaFold2(用于结构预测),把整个流程跑通了!现将安装与初步使用过程记录下来,希望能帮到同样在 macOS(特别是 Apple Silicon M 系列芯片)上折腾的朋友们。
1.ProteinMPNN安装
1.1 克隆仓库
bash
mkdir proteinMPNN
cd proteinMPNN
git clone https://github.com/dauparas/ProteinMPNN.git 1.2 创建 Conda 环境
bash
conda create -n proteinMPNN
conda activate proteinMPNN
conda install pytorch torchvision torchaudio -c pytorch 1.3 测试运行
进入项目目录,查看帮助:
bash
cd ProteinMPNN #cd 到下载好的ProteinMPNN的文件夹
python protein_mpnn_run.py -h # 该命令将显示脚本使用说明和可用的输入参数。尝试对 PDB 结构 5L33 进行全序列重设计(无任何约束):
bash
mkdir output_try
python protein_mpnn_run.py --pdb_path inputs/PDB_monomers/pdbs/5L33.pdb --out_folder output_try/5L33_redesign --num_seq_per_target 10 --sampling_temp "0.1" --seed 9 --batch_size 1 --model_name v_48_020 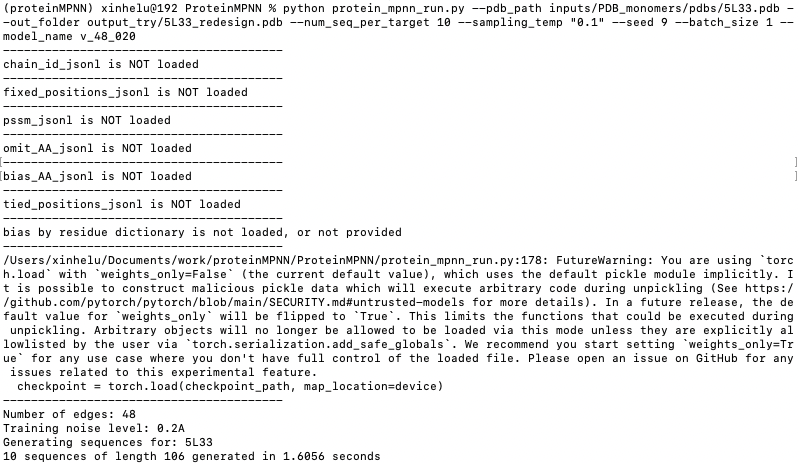
运行成功后,输出文件夹中会包含 10 条新设计的序列(FASTA 格式),每条附带打分和恢复率信息。
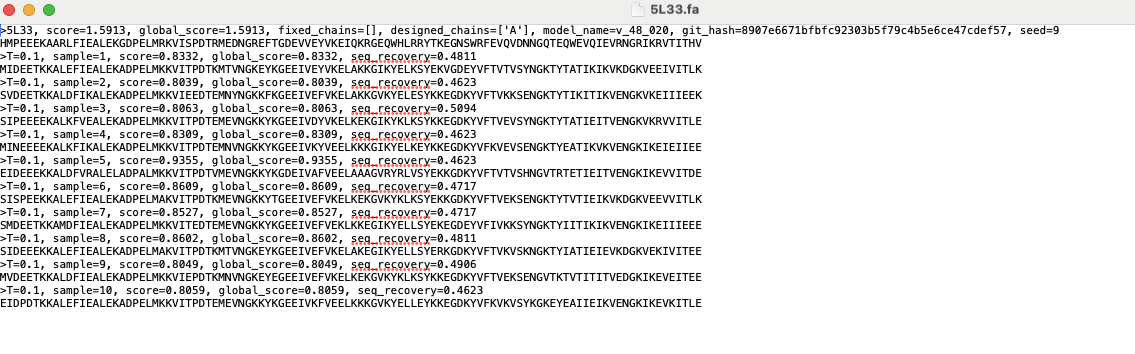
假如我们输入RFdiffusion生成的只有甘氨酸(G)和位置信息的蛋白质骨架输入,ProteinMPNN就会生成有序列的fasta格式啦!接下来基于AlphaFold2对生成结构的合理性打分进行筛选,就可以进行表达验证!
2.AlphaFold2安装
由于原版 AlphaFold2 在本地部署复杂,我选择了轻量高效的 ColabFold(由 sokrypton 团队开发),它兼容 Apple Silicon,并支持模板、Amber 优化等高级功能。
2.1 安装 Homebrew(如未安装)
/bin/bash -c "$(curl -fsSL https://raw.githubusercontent.com/Homebrew/install/HEAD/install.sh)"如果无法安装brew,可自行前往官网下载安装(https://brew.sh)
2.2 安装依赖工具与ColabFold
(虽然写了m1,但经测m4也可以使用,应该m系列芯片通用)
bash
/bin/bash -c "$(curl -fsSL https://raw.githubusercontent.com/Homebrew/install/HEAD/install.sh)"
#输入mac解锁密码
brew install wget cmake gnu-sed brewsci/bio/hh-suite kalign mmseqs2
brew install --cask miniforge
wget https://raw.githubusercontent.com/YoshitakaMo/localcolabfold/main/install_colabbatch_M1mac.sh
chmod 777 ./install_colabbatch_M1mac.sh
bash ./install_colabbatch_M1mac.sh
#有的时候一次无法安装成功,可多次安装最后一行代码2.3 配置环境变量并验证安装
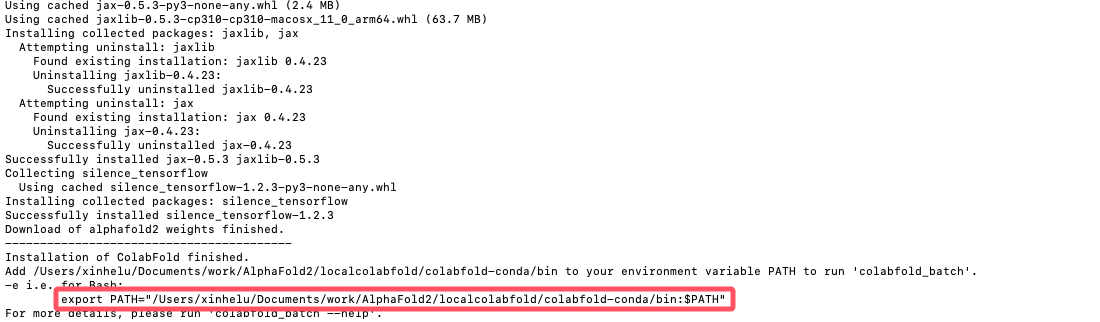
安装完成后,终端会提示你添加 PATH。例如:
bash
#最后一行代码生成的环境
export PATH="/Users/xinhelu/Documents/work/AlphaFold2/localcolabfold/colabfold-conda/bin:$PATH"
#验证安装(查看帮助,需要几秒钟时间)
colabfold_batch --help(如果安装遇到其他问题,可以看看文末的ColabFold安装参考)
2.4 测试
预测人类 Oncostatin-M(P13725)
下载 FASTA 文件:https://rest.uniprot.org/uniprotkb/P13725.fasta
放入工作目录后运行:
验证代码
./localcolabfold/colabfold-conda/bin/colabfold_batch --amber --templates --num-recycle 3 --model-order 1,2,3,4,5 ./P13725.fasta FFA2output蛋白序列:
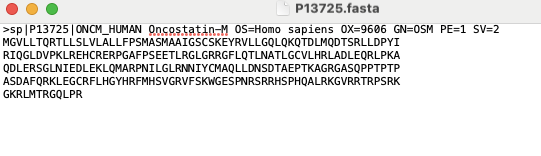
结果将保存在 FFA2output 文件夹中,包含预测结构(PDB)、置信度(pLDDT)等。
proteinMPNN安装参考(稍作改动,mac):
1.https://blog.csdn.net/qq_41025456/article/details/145738451
ColabFold安装参考(mac):
1.https://marcdeller.com/alphafold-local-install-on-mac/
3.https://github.com/YoshitakaMo/localcolabfold
蛋白设计交流QQ群:438723520
作者:bioinforiver
2025年12月4日
本内容仅供学习交流,未经许可不许转载。
(本人系原作者,原文见:https://mp.weixin.qq.com/s/5kCuz0wNfTkTuC5vv6bT8A)