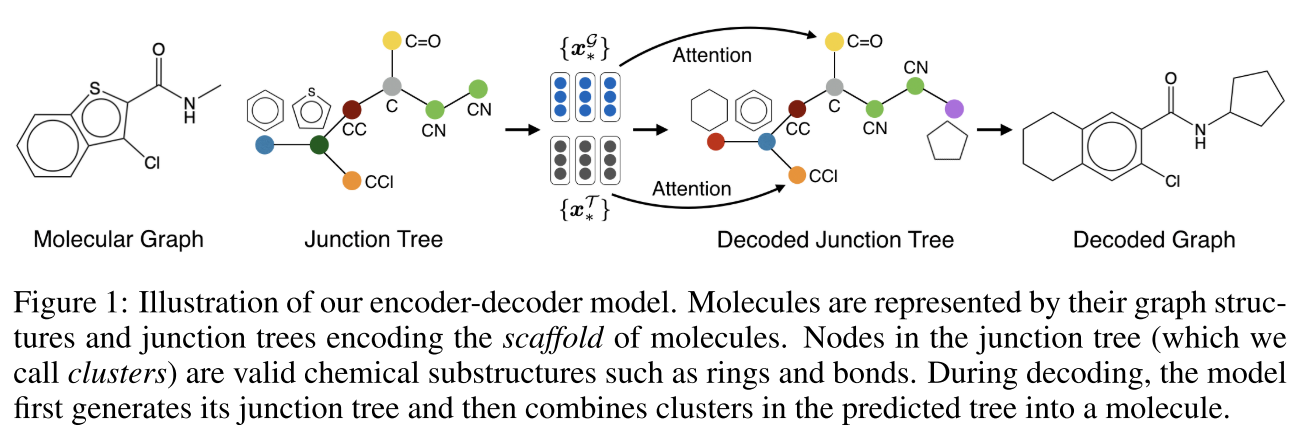
论文题目:Learning Multimodal Graph-to-Graph Translation for Molecular Optimization(学习用于分子优化的多模态图到图转换)
会议:ICLR2019
摘要:我们认为分子优化是一个图到图的转换问题。目标是学习从一个分子图映射到另一个具有更好性质的基于成对分子的可用语料库的分子图。由于分子可以以不同的方式进行优化,因此每个输入图都有多个可行的翻译。因此,一个关键的挑战是对不同的翻译输出进行建模。我们的主要贡献包括用于学习不同图翻译的连接树编码器-解码器以及用于对齐分子分布的新型对抗性训练方法。在我们的模型中,不同的输出分布是通过调节翻译过程的低维潜在向量显式实现的。我们在多个分子优化任务上评估了我们的模型,并表明我们的模型优于以前最先进的基线。
分子优化的图到图翻译 - 用AI设计更好的药物分子
引言
药物发现的核心挑战之一是如何优化分子结构以获得更好的药物性质。今天我们解读Wengong Jin等人在ICLR 2019发表的论文《Learning Multimodal Graph-to-Graph Translation for Molecular Optimization》。这篇论文将分子优化重新定义为图到图的翻译问题,并提出了创新的多模态学习方案。
问题定义与动机
传统方法:匹配分子对分析(MMPA)
MMPA的工作流程:
- 收集分子对(X, Y),其中Y比X具有更好的化学性质
- 提取图转换规则(如"将甲基替换为乙基可提高溶解度")
- 应用规则生成新分子
MMPA的局限:
- 需要实现数百万条规则才能覆盖复杂模式
- 规则需要人工设计和维护
- 难以捕捉复杂的结构变化
新视角:图到图翻译
将分子优化视为机器翻译任务:
- 输入:性质较差的分子图X
- 输出:性质更好的分子图Y
- 训练数据:分子对(X, Y)
核心挑战:
- 如何生成有效的分子图(而非线性序列)
- 如何建模多样化的输出(一个分子可通过多种方式优化)
- 如何保证生成分子的化学有效性
Junction Tree表示
为什么不直接用图?
分子图的直接生成面临挑战:
- 需要保证化学有效性(原子价、环结构等)
- 解码过程难以并行
- 难以控制整体骨架结构
Junction Tree(连接树)方法:
将分子分解为两个层次:
- Junction Tree(骨架):节点是化学有效的子结构(如苯环、键等)
- Molecular Graph(细节):完整的分子图
示例:对于一个包含苯环和其他基团的分子:
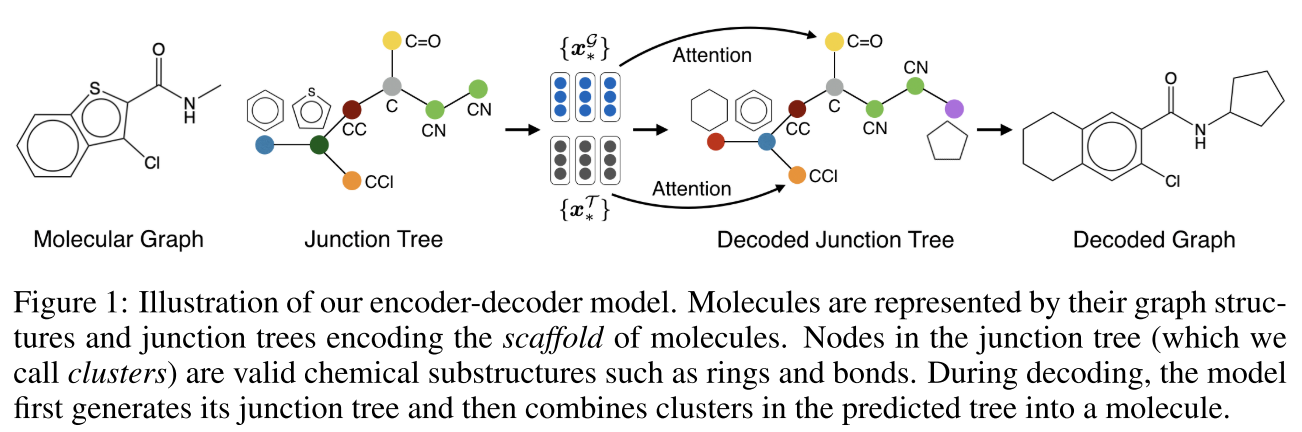
- Junction tree节点:苯环、C=O基团、CCl基团、CN基团等
- 边:表示这些子结构如何连接
优势:
- 保证生成的子结构化学有效
- 从粗到细的解码过程
- 提供多尺度表示
模型架构详解
1. 统一编码器(Tree and Graph Encoder)
对树和图使用相同的消息传递网络架构。
消息传递(T轮迭代):
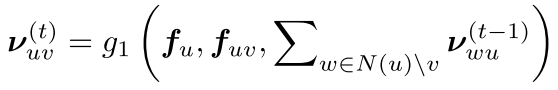
其中:
- f_u: 节点特征
- f_{uv}: 边特征
- N(u): u的邻居
节点表示聚合:
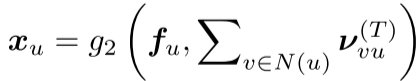
这产生两组向量:
- Tree向量:

- Graph向量:

关键改进:与之前的工作不同,这里:
- 不假设预定义的根节点
- 消息异步更新(无人为顺序)
- 避免了顺序偏差
2. Junction Tree解码器
使用树递归神经网络生成junction tree,采用深度优先遍历顺序。
核心组件:
Tree GRU(更新消息):
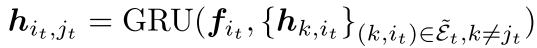
拓扑预测(扩展新节点还是回溯?):
计算预测隐状态:
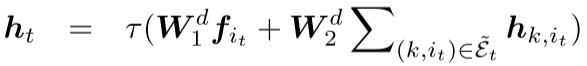
通过注意力机制聚合源信息:
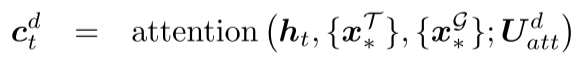
预测概率:
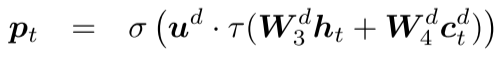
标签预测(新节点的化学子结构类型):
同样使用注意力:
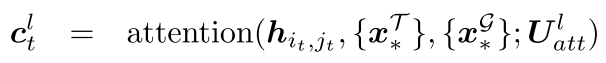
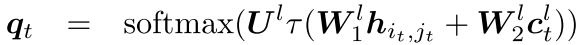
注意力机制的作用:
- 动态关注源分子的相关部分
- 分别处理树和图层面的信息
- 提供跨层次的信息流
3. Graph解码器
给定预测的junction tree  ,组装成完整分子图。
,组装成完整分子图。
挑战:同一个junction tree可能对应多个分子(如图2所示的3种组装方式)
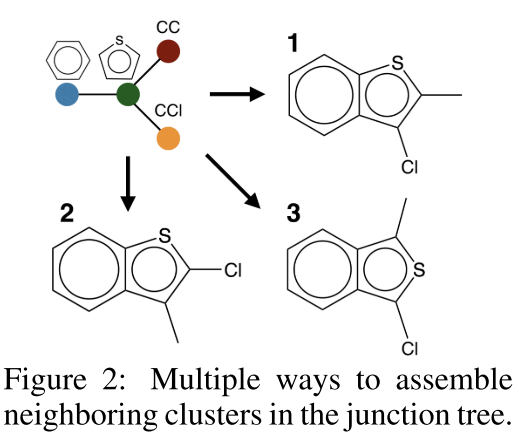
解决方案 :为每个树节点i评分所有可能的局部连接方式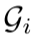
评分函数:
- 对候选图G_i运行图消息传递得到原子表示
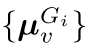
- 求和池化:
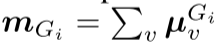
- 与源图向量计算点积:
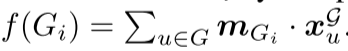
训练目标:
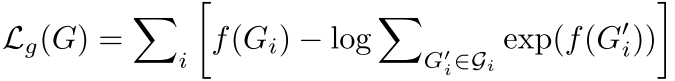
多模态学习
目标 :学习映射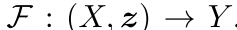 ,其中
,其中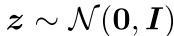 是潜在编码
是潜在编码
为什么需要潜在编码?
- 一个分子有多种优化路径(多模态)
- 需要显式建模输出的多样性
- 潜在编码控制生成的不同"风格"
1. Variational Junction Tree Encoder-Decoder (VJTNN)
潜在编码推断:
计算分子X和Y之间的差异向量:
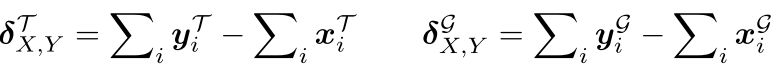
这个差异向量编码了从X到Y的结构变化。
通过变分推断得到潜在编码:
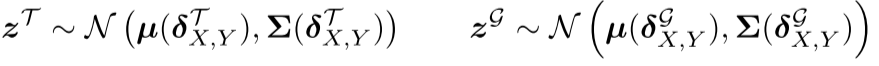
融合潜在编码:

"扰动"后的向量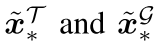 输入解码器。
输入解码器。
训练目标(VAE损失):
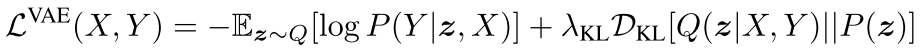
其中:
- 重构损失:鼓励准确生成Y
- KL散度:正则化潜在分布接近先验
关键技术细节:
- 潜在编码维度:|z| = 8(低维避免模型忽略输入X)
2. 对抗性骨架正则化(Adversarial Scaffold Regularization)
动机:确保从先验生成的分子符合目标域分布
挑战:离散的树/图解码阻碍梯度传播
解决方案:在连续表示上进行对抗训练
连续表示构造:
在树解码过程中,累积软(连续)信息:
- 根节点标签分布
 (softmax,非one-hot)
(softmax,非one-hot) - 隐藏消息
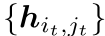 (连续向量)
(连续向量) - 使用预测的概率(而非硬决策)加权消息
修改的树GRU(算法2):

其中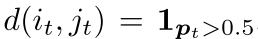 是二值门控。
是二值门控。
梯度处理:使用straight-through estimator
- 前向传播:使用阈值函数(离散)
- 反向传播:使用hard sigmoid(连续)
最终表示 :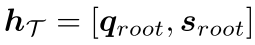
其中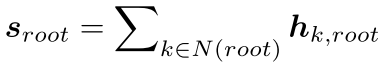 是入边消息的和。
是入边消息的和。
判别器:多层前馈网络D(.)
对抗训练(Wasserstein GAN with Gradient Penalty):
训练流程:
- 每个生成器更新前,判别器训练N=5次
- 交替优化判别器和生成器
- 最终模型:VJTNN + GAN
实验设置
数据集构造:
所有任务都从ZINC数据集构造训练对(X, Y),满足:
- 相似性约束:
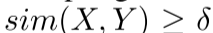 (Tanimoto相似度)
(Tanimoto相似度) - 性质改进显著
任务1: Penalized logP
- 目标:提升penalized logP分数
- 测试集:与JT-VAE相同
任务2: QED (Drug Likeness)
- 目标:将QED ∈ [0.7, 0.8]的分子提升到[0.9, 1.0]
- 挑战:目标范围仅占ZINC数据集的6.6%
- 训练集:88K对
- 测试集:800个分子
任务3: DRD2 (生物活性)
- 目标:将DRD2活性从p \< 0.05提升到p \> 0.5
- 挑战:活性分子仅占1.9%
- 训练集:34K对
- 测试集:1000个分子
基线方法:
- MMPA:使用mmpdb工具包提取转换规则
- JT-VAE:在潜在空间梯度上升优化
- VSeq2Seq:基于SMILES的变分seq2seq(Bahdanau attention)
- GCPN:强化学习方法,迭代添加/删除原子和键
模型配置:
- 隐藏维度:300
- 潜在编码:|z| = 8
- 树编码器迭代:6次
- 图编码器迭代:3次
- 总参数:3.9M
- 优化器:Adam(学习率0.001,每epoch衰减0.9)
- 训练轮数:20 epochs
实验结果深度分析
1. Penalized logP任务
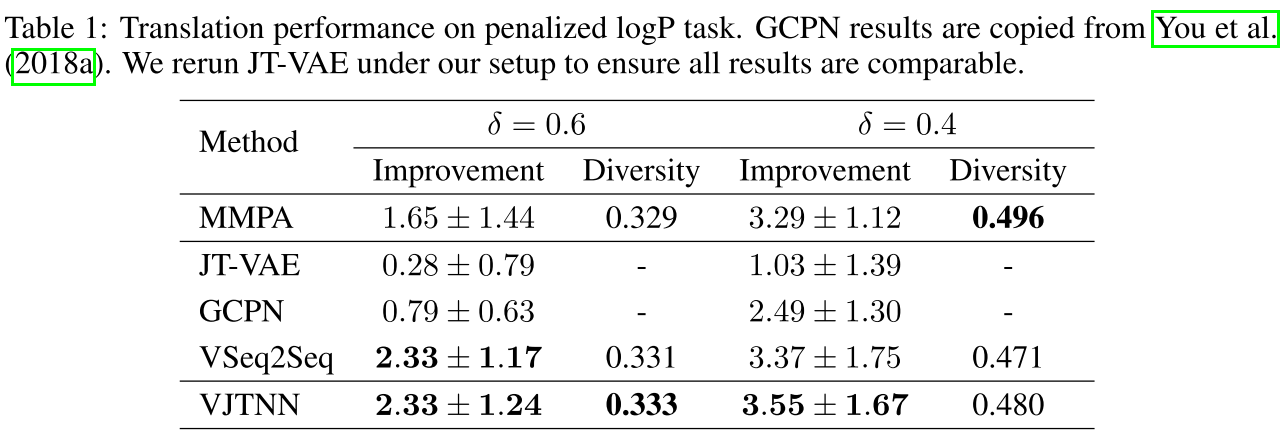
| 方法 | 改进(δ=0.6) | 多样性 | 改进(δ=0.4) | 多样性 |
|---|---|---|---|---|
| MMPA | 1.65 ± 1.44 | 0.329 | 3.29 ± 1.12 | 0.496 |
| JT-VAE | 0.28 ± 0.79 | - | 1.03 ± 1.39 | - |
| GCPN | 0.79 ± 0.63 | - | 2.49 ± 1.30 | - |
| VSeq2Seq | 2.33 ± 1.17 | 0.331 | 3.37 ± 1.75 | 0.471 |
| VJTNN | 2.33 ± 1.24 | 0.333 | 3.55 ± 1.67 | 0.480 |
关键发现:
- VJTNN相比GCPN提升42% (2.49 → 3.55, δ=0.4)
- 相比JT-VAE提升245% (1.03 → 3.55)
- 多样性略低于MMPA但保持竞争力
- 监督学习方法(VJTNN, VSeq2Seq)明显优于非监督/强化学习方法
2. QED任务
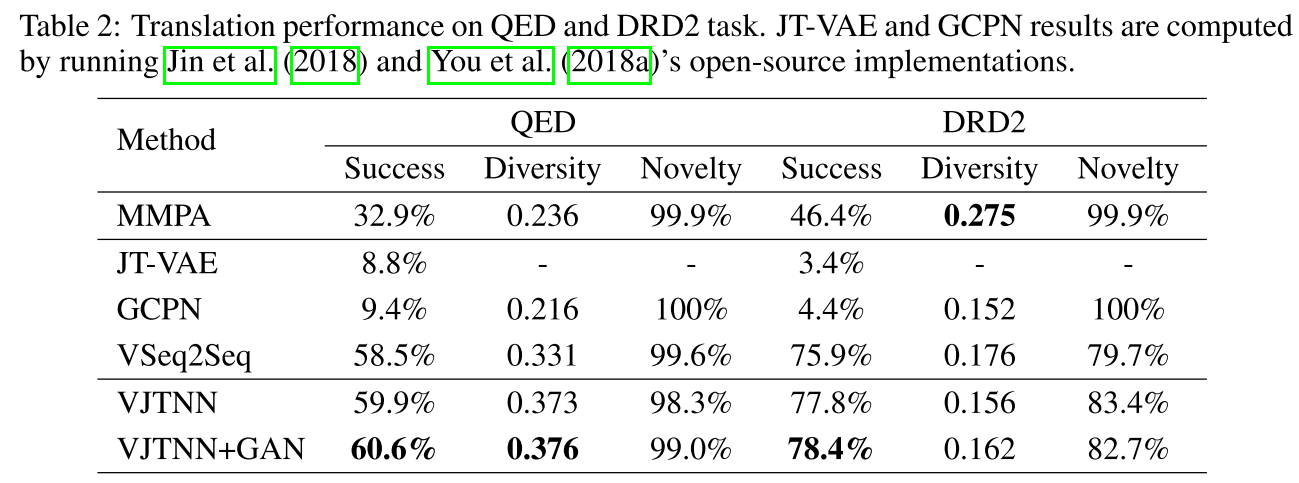
关键发现:
- VJTNN+GAN成功率是GCPN的6.4倍 (9.4% → 60.6%)
- 相比JT-VAE提升587% (8.8% → 60.6%)
- 对抗训练小幅提升成功率(59.9% → 60.6%)和多样性(0.373 → 0.376)
- 98%+的新颖性表明模型能发现新化合物
3. DRD2任务
关键发现:
- VJTNN+GAN成功率是GCPN的17.8倍 (4.4% → 78.4%)
- 相比JT-VAE提升2206% (3.4% → 78.4%)
- 序列方法(VSeq2Seq)新颖性较低(79.7%),可能过拟合训练数据
- VJTNN在准确性和新颖性之间取得更好平衡
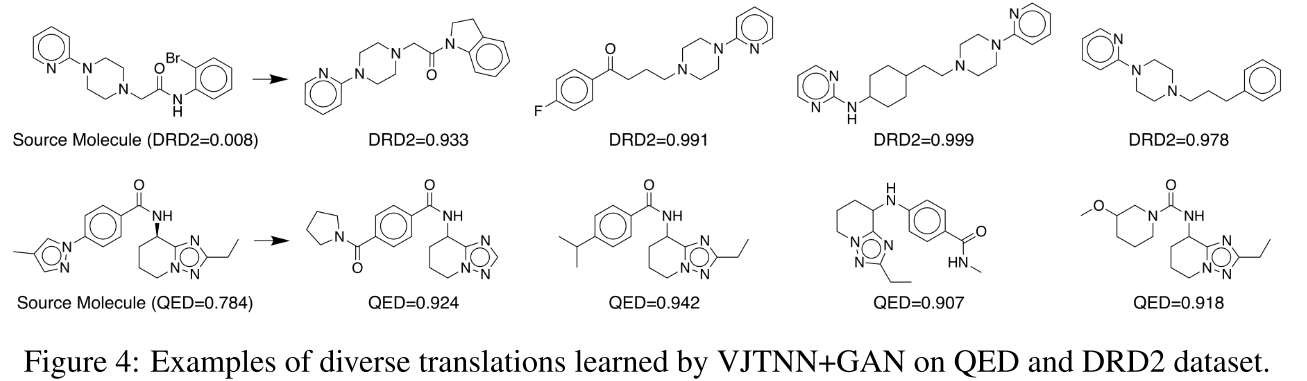
4. 多样性定性分析
图4展示了同一源分子的多种翻译结果:
QED示例(源分子QED=0.784):
- 翻译1:添加环状结构,QED=0.824
- 翻译2:修改侧链,QED=0.942
- 翻译3:调整官能团位置,QED=0.918
DRD2示例(源分子DRD2=0.008):
- 翻译1:添加氮杂环,DRD2=0.933
- 翻译2:修改苯环取代基,DRD2=0.991
- 翻译3:改变连接方式,DRD2=0.999
- 翻译4:添加双键和环,DRD2=0.978
这些示例展示了模型确实学到了多种有效的分子优化策略。
总结
这篇论文将分子优化问题重新定义为图到图翻译,并提出了创新的多模态学习框架。核心贡献包括:
理论贡献:
- 将MMPA转换为端到端学习问题
- Junction tree分层表示
- 变分学习+对抗训练的组合方案
技术贡献:
- 统一的树/图编码器
- 带注意力的树解码器
- 连续表示上的对抗正则化
实验贡献:
- 三个分子优化任务上的SOTA性能
- 大幅超越强化学习基线
- 验证了多样性和新颖性
影响力:
- 启发了后续大量分子生成工作
- 对抗正则化技术被广泛采用
- Junction tree表示成为标准选择
对于药物发现和分子设计领域的研究者,这是一篇必读的论文。它不仅提供了实用的工具,还展示了如何将机器学习技术创造性地应用于化学问题。