🎯 一、总体目标(Why)
把
n条线段(用中心点(cx, cy)表示)分成k个空间上紧凑的组(簇),使得同一组内的线段彼此靠近。
这是典型的 K-Means 聚类问题 ,但只关注位置信息,忽略长度、角度等其他特征。
实际case跑出来的结果如下:
1. 高斯分布的直线类聚
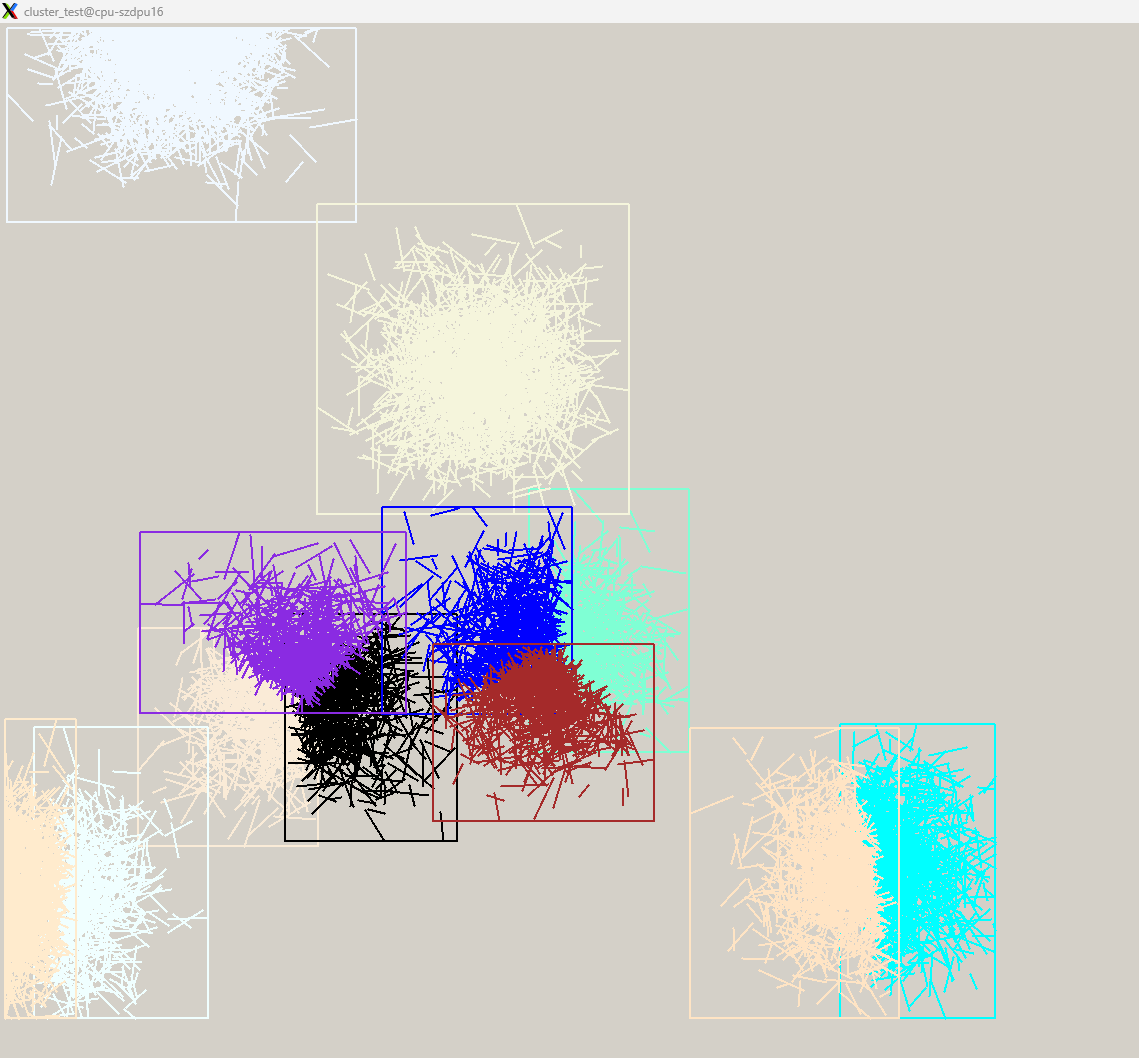
2. 均匀分布的直线类聚

🔧 二、函数输入输出(What)
QVector<int> kmeansPositionOnly(
const QVector<Feature>& feature_list, // 输入:每条线的特征(我们只用 .m_cx, .m_cy)
int k, // 想分多少个簇(box)
int max_iters = 1000 // 最多迭代 1000 次(防死循环)
)返回值 :labels[i] 表示第 i 条线属于哪个簇(取值 0 到 k-1)。
🧱 三、核心思想(How)------ K-Means 的两步迭代
K-Means算法思想如下:
- 随机选取K个中心点。(KMEANS++会在选取一个中心点后更倾向于去选择离选定中心点更远的)
- 计算其他点离哪个中心点更近,就算做哪一簇。
- 计算每个新簇的新中心点(取平均)。
- 重新调整除中心点外的归属情况。
- 直到每次分簇情况相同或者中心点收敛。
K-Means 是一个迭代优化算法,每轮做两件事:
| 步骤 | 名称 | 做什么 |
|---|---|---|
| 1️⃣ | 分配(Assignment) | 对每个点,找离它最近的簇中心,把它"分给"那个簇 |
| 2️⃣ | 更新(Update) | 对每个簇,重新计算其中心(所有点的平均位置) |
重复这两步,直到:
- 所有点的归属不再变化(收敛)
- 达到最大迭代次数(安全退出)
Z-score 标准化(或归一化)
1. 什么是方差(Variance)?
假设有一组数:x₁, x₂, ..., xₙ
-
先算平均值:
μ=n1∑i=1nxi
-
再算每个数离平均值有多远(偏差):
(xi−μ)
-
把所有偏差平方后求平均(避免正负抵消):
Var(x)=n1∑i=1n(xi−μ)2
👉 这个 Var(x) 就是方差。
- 方差越大 → 数据越分散
- 方差越小 → 数据越集中
2. 什么是"单位方差"?
就是让这个方差 等于 1。
怎么做到?------ 除以标准差(standard deviation)
因为:
- 标准差 = √方差 → 记作
σ - 如果你对每个数据做变换:
zi=σxi−μ
那么新数据 z₁, z₂, ..., zₙ 的:
- 均值 = 0
- 方差 = 1 ← 这就是"单位方差"
这个过程叫 Z-score 标准化(或归一化)。
📚 四、代码逐段详解
✅ 第 1 部分:边界检查 & 初始化
int n = feature_list.size();
if (n == 0 || k <= 0) return QVector<int>(); // 空输入或无效 k,直接返回
if (k > n) k = n; // 不能分比点还多的簇💡 例如:只有 3 条线,却想分 10 个 box?最多只能分 3 个(每条一个簇)。
QVector<int> labels(n, 0); // labels[i]:第 i 条线当前属于哪个簇(初始全为 0)
QVector<Feature> centroids(k); // 存储 k 个簇的"中心点"(质心)
// 初始化质心:取前 k 个点作为初始中心
for (int i = 0; i < k; ++i)
{
centroids[i] = feature_list[i];
}⚠️ 这是简单初始化(不是最优的 K-Means++),但在实践中对结果影响有限,尤其当数据量大时。
🔁 第 2 部分:主循环 ------ 迭代优化(最多 max_iters 轮)
for (int iter = 0; iter < max_iters; ++iter)
{
bool changed = false; // 标记本轮是否有任何点换了簇▶ 步骤 A:分配阶段(Assign points to nearest centroid)
for (int i = 0; i < n; ++i)
{
int best_k = 0;
double best_dist = dist2PositionOnly(feature_list[i], centroids[0]);
// 遍历所有 k 个簇中心,找最近的
for (int j = 1; j < k; ++j)
{
double d = dist2PositionOnly(feature_list[i], centroids[j]);
if (d < best_dist)
{
best_dist = d;
best_k = j;
}
}
// 如果归属变了,记录下来
if (labels[i] != best_k)
{
labels[i] = best_k;
changed = true;
}
}✅
dist2PositionOnly(a, b)只计算(a.cx - b.cx)^2 + (a.cy - b.cy)^2,不涉及 len/ang。
🎯 目标:让每个点都"就近归队"。
▶ 提前终止:如果没人换队,说明已稳定!
if (!changed) break; // 收敛!无需继续迭代▶ 步骤 B:更新阶段(Recompute centroids)
QVector<Feature> new_centroids(k); // 临时累加和
QVector<int> counts(k, 0); // 每个簇有多少个点
// 遍历所有点,按当前标签累加到对应簇
for (int i = 0; i < n; ++i)
{
int cid = labels[i]; // 当前点属于哪个簇
new_centroids[cid].m_cx += feature_list[i].m_cx;
new_centroids[cid].m_cy += feature_list[i].m_cy;
counts[cid]++;
}
// 正式更新质心 = 平均位置
for (int i = 0; i < k; ++i)
{
if (counts[i] > 0)
{
centroids[i].m_cx = new_centroids[i].m_cx / counts[i];
centroids[i].m_cy = new_centroids[i].m_cy / counts[i];
// 注意:len/ang 不参与聚类,所以不更新(保持原值也无影响)
}
else
{
// ❗ 处理"空簇":某个簇没被分配任何点
centroids[i] = feature_list[qrand() % n]; // 随机选一个点作为新中心
}
}💡 "空簇"问题 :当
k设得太大,或初始中心太集中,某些簇可能没人"认领"。解决方案:随机重置,避免该簇永远为空导致算法失效。
📤 第 3 部分:返回结果
return labels; // 每个点的最终簇编号这个 labels 向量会被用于:
- 分组线段
- 计算每个组的包围盒(Box)
- 输出最终的聚类结果
🌟 五、关键设计亮点(针对你的需求)
| 特性 | 说明 |
|---|---|
仅用位置 (cx, cy) |
忽略长度、角度,确保空间邻近的线被分在一起 |
| 标准化已在调用前完成 | 所以这里直接使用归一化后的坐标,保证 x/y 尺度一致 |
| 处理空簇 | 随机重置,提升鲁棒性 |
| 提前收敛 | 一旦稳定就退出,节省计算 |
| 最大迭代保护 | max_iters=1000 防止极端情况卡死(实际通常 10~50 轮就收敛) |
📊 六、举个小例子(k=2)
假设 4 条线的中心点(已标准化):
- L0: (-1.0, -0.9)
- L1: (-0.8, -1.1)
- L2: (0.9, 1.0)
- L3: (1.1, 0.8)
- 初始质心:C0 = L0, C1 = L1
- 第1轮分配 :
- L0, L1 → C0
- L2, L3 → C1(因为离 C1 更近?其实要看距离)
- 更新质心 :
- C0 = avg(L0,L1) ≈ (-0.9, -1.0)
- C1 = avg(L2,L3) ≈ (1.0, 0.9)
- 第2轮分配 :结果不变 → 收敛!
- 返回 labels = [0, 0, 1, 1]
✅ 成功分成左右两组!
✅ 七、总结:这段代码的实现思路
- 目标明确 :按空间位置将线段聚成
k组。 - 算法选择:使用经典 K-Means(分配 + 更新迭代)。
- 特征简化 :只用
(cx, cy),忽略干扰项。 - 健壮性保障 :
- 边界检查
- 空簇处理
- 提前收敛
- 最大迭代限制
- 工程友好:接口简单,结果可直接用于后续 Box 构建。
你现在完全掌握了这段代码的设计哲学与实现细节 !
它是一个轻量、高效、可靠的 K-Means 实现,专为你"可控数量 box"的需求量身定制 ✅
如需进一步优化(如 K-Means++ 初始化、多线程加速、支持权重等),也可以在此基础上扩展。
整体代码如下:
cpp
struct Feature
{
double m_cx, m_cy, m_len, m_ang;
Feature(double cx = 0.0, double cy = 0.0, double len = 0.0, double ang = 0.0)
: m_cx(cx),
m_cy(cy),
m_len(len),
m_ang(ang)
{
}
};
static inline double dist2PositionOnly(const Feature& a, const Feature& b)
{
double dx = a.m_cx - b.m_cx;
double dy = a.m_cy - b.m_cy;
return dx * dx + dy * dy;
}
static void standardizePositionOnly(QVector<Feature>& feature_list)
{
int n = feature_list.size();
if (n == 0)
return;
double mean_cx = 0.0, mean_cy = 0.0;
for (int i = 0; i < n; ++i)
{
mean_cx += feature_list[i].m_cx;
mean_cy += feature_list[i].m_cy;
}
mean_cx /= n;
mean_cy /= n;
double var_cx = 0.0, var_cy = 0.0;
for (int i = 0; i < n; ++i)
{
double dx = feature_list[i].m_cx - mean_cx;
double dy = feature_list[i].m_cy - mean_cy;
var_cx += dx * dx;
var_cy += dy * dy;
}
var_cx /= n;
var_cy /= n;
double std_cx = qSqrt(var_cx);
double std_cy = qSqrt(var_cy);
if (std_cx < 1e-12)
std_cx = 1.0;
if (std_cy < 1e-12)
std_cy = 1.0;
for (int i = 0; i < n; ++i)
{
feature_list[i].m_cx = (feature_list[i].m_cx - mean_cx) / std_cx;
feature_list[i].m_cy = (feature_list[i].m_cy - mean_cy) / std_cy;
}
}
static QVector<int> kmeansPositionOnly(const QVector<Feature>& feature_list, int k, int max_iters)
{
int n = feature_list.size();
if (n == 0 || k <= 0)
return QVector<int>();
if (k > n)
k = n;
QVector<int> labels(n, 0);
QVector<Feature> centroids(k);
qsrand(QTime::currentTime().msec());
for (int i = 0; i < k; ++i)
{
centroids[i] = feature_list[qrand() % n];
}
for (int iter = 0; iter < max_iters; ++iter)
{
bool changed = false;
for (int i = 0; i < n; ++i)
{
int best_k = 0;
double best_dist = dist2PositionOnly(feature_list[i], centroids[0]);
for (int j = 1; j < k; ++j)
{
double d = dist2PositionOnly(feature_list[i], centroids[j]);
if (d < best_dist)
{
best_dist = d;
best_k = j;
}
}
if (labels[i] != best_k)
{
labels[i] = best_k;
changed = true;
}
}
if (!changed)
break;
QVector<Feature> new_centroids(k);
QVector<int> counts(k, 0);
for (int i = 0; i < n; ++i)
{
int cid = labels[i];
new_centroids[cid].m_cx += feature_list[i].m_cx;
new_centroids[cid].m_cy += feature_list[i].m_cy;
counts[cid]++;
}
for (int i = 0; i < k; ++i)
{
if (counts[i] > 0)
{
centroids[i].m_cx = new_centroids[i].m_cx / counts[i];
centroids[i].m_cy = new_centroids[i].m_cy / counts[i];
}
else
{
centroids[i] = feature_list[qrand() % n];
}
}
}
return labels;
}
static Box computeBoundingBox(const QVector<Line>& lines)
{
if (lines.isEmpty())
return Box(0, 0, 0, 0);
double xmin = std::numeric_limits<double>::max();
double ymin = std::numeric_limits<double>::max();
double xmax = -std::numeric_limits<double>::max();
double ymax = -std::numeric_limits<double>::max();
for (const Line& line : lines)
{
xmin = qMin(xmin, qMin(line.p1().x(), line.p2().x()));
ymin = qMin(ymin, qMin(line.p1().y(), line.p2().y()));
xmax = qMax(xmax, qMax(line.p1().x(), line.p2().x()));
ymax = qMax(ymax, qMax(line.p1().y(), line.p2().y()));
}
return Box(xmin, ymin, xmax, ymax);
}
QVector<ClusterResult> Algorithm::clusterLines(const QVector<Line>& lines, int k, int max_iters)
{
QVector<ClusterResult> results;
if (lines.isEmpty() || k <= 0)
return results;
QVector<Feature> features;
features.reserve(lines.size());
for (int i = 0; i < lines.size(); ++i)
{
const Line& line = lines[i];
double cx = (line.p1().x() + line.p2().x()) * 0.5;
double cy = (line.p1().y() + line.p2().y()) * 0.5;
features.append(Feature(cx, cy, 0.0, 0.0));
}
standardizePositionOnly(features);
QVector<int> labels = kmeansPositionOnly(features, k, max_iters);
QVector<QVector<Line>> groups(k);
for (int i = 0; i < lines.size(); ++i)
{
int cid = labels[i];
if (cid < 0 || cid >= k)
cid = 0;
groups[cid].append(lines[i]);
}
for (int i = 0; i < k; ++i)
{
if (groups[i].isEmpty())
continue;
Box box = computeBoundingBox(groups[i]);
results.append(ClusterResult(box, groups[i]));
}
return results;
}