Gepard:叶绿体基因组组装的高效点阵图分析工具
有四分体结构的叶绿体
五种肉苁蓉属植物叶绿体基因组-文献精读25_肉苁蓉全基因组-CSDN博客
13个黄杞属叶绿体基因组-文献精读68_genome varscan插件报错-CSDN博客
GetOrganelle(组装叶绿体)安装与使用-bioinfomatics tools 34-CSDN博客
01 工具背景:什么是 Gepard?
Gepard 的全称是Genome Pair Rapid Dotter (基因组配对快速点阵分析工具),是一款专为生物序列比对设计的点阵图(Dotplot)绘制工具,由 Jan Krumsiek 等团队于 2007 年开发并发表在《Bioinformatics》期刊,核心基于后缀数组(Suffix Array) 算法实现高效的序列比对分析,是基因组组装、序列相似性验证领域的经典工具,尤其在细胞器基因组(叶绿体、线粒体)组装中应用广泛。
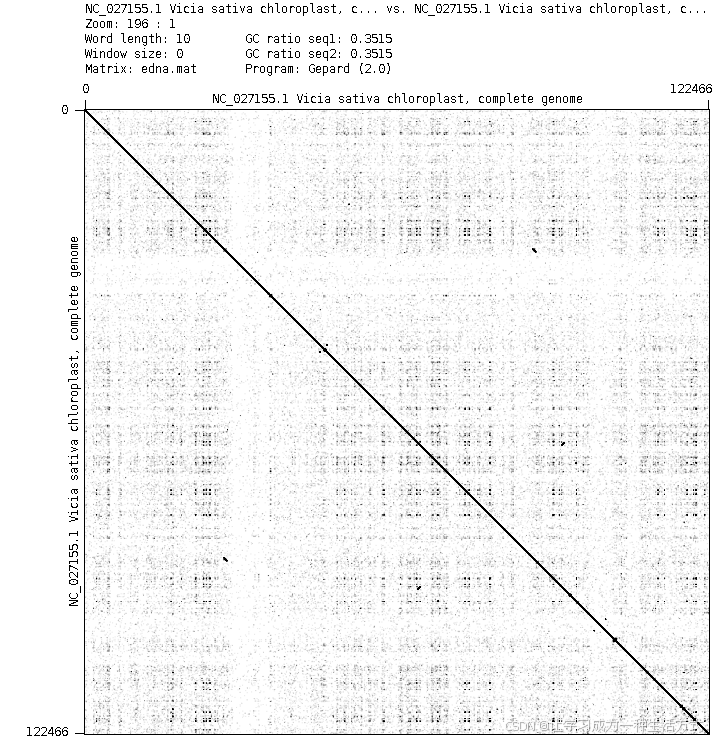
补充背景:点阵图(Dotplot)是什么?
点阵图是生物序列分析的基础可视化手段,以二维坐标系呈现两条序列(或一条序列自比对)的相似性:横轴和纵轴分别代表待分析的序列,坐标系中的每个点代表对应位置的序列片段存在匹配,连续的点形成的斜线代表序列的连续相似区域,缺口、断点则代表序列的插入、缺失或变异。对于短而紧凑的叶绿体基因组(大小约 120-160kb),点阵图能直观反映序列的重复区域、组装准确性、反向重复序列(IR 区,叶绿体基因组标志性结构)完整性,是组装后验证的关键步骤。
02 核心价值:为何叶绿体组装要用到 Gepard?
叶绿体基因组作为植物的重要细胞器基因组,具有结构保守、存在反向重复区(IRa/IRb)、单拷贝区(LSC/SSC) 等特征,组装过程中易因重复序列导致拼接错误(如重复区漏拼、方向颠倒),而 Gepard 恰好能解决这些问题:
- 验证组装准确性:将组装得到的叶绿体基因组序列自比对,通过点阵图的连续斜线判断序列是否存在拼接断点、缺失;
- 识别重复区域:清晰展示叶绿体基因组的 IR 区边界,验证 IR 区的长度和序列一致性;
- 检测序列变异:对比样品与参考叶绿体基因组,快速定位 SNP、Indel 或结构变异区域;
- 高效适配小基因组:Gepard 将点阵图计算的时间复杂度优化至Θ(m*log n)(m、n 为待比对序列长度),对叶绿体这类小基因组能实现秒级比对,远快于传统点阵图工具。
简单来说,Gepard 是叶绿体基因组组装 "从序列到正确结构" 的关键验证工具,也是后续基因组注释、进化分析的基础保障。
03 Gepard 核心特性
- 操作友好:提供交互式可视化界面,无需复杂的命令行参数,可直接调整比对参数、可视化效果;
- 速度极快:基于后缀数组算法,大幅降低计算复杂度,支持基因组尺度的序列比对(适配叶绿体 / 线粒体 / 细菌等小基因组,也支持真核生物核基因组局部区域分析);
- 创新的客户 - 服务器模式:可直接基于在线序列 / 注释数据计算点阵图并按功能注释着色,无需提前下载序列或注释文件,节省本地存储空间;
- 多格式支持:兼容 FASTA、GenBank 等主流生物序列格式,输出的点阵图可导出为高清图片,直接用于科研论文作图;
- 命令行 + 图形界面双模式 :既提供
gepardcmd命令行工具(适合批量分析、脚本化流程),也提供交互式 GUI 界面(适合手动调整、可视化分析),满足不同分析需求; - 配套教程完善 :工具内置
futorial.html文件,包含详细的使用步骤和参数说明,新手易上手。
04 安装方法(两种便捷方式,无需修改代码)
Gepard 的安装无需修改任何源码,推荐两种生物信息学领域的标准安装方式,可根据自身电脑环境(Windows/Mac/Linux)选择,均为官方推荐的稳定版本(2.1.0)。
4.1 Docker 安装(推荐跨平台 / 无环境冲突)
Docker 是轻量级容器工具,能彻底解决 "环境依赖冲突" 问题,安装后可直接运行 Gepard,无需配置其他依赖,适合跨平台使用或本地环境复杂的情况。
前提条件
本地已安装并运行 Docker(Docker 安装教程:Docker 官方教程,Windows/Mac 可安装 Docker Desktop,Linux 可通过包管理器安装)。
安装 & 运行命令(直接复制执行,无需修改)
# 拉取Gepard 2.1.0稳定版镜像(biocontainers官方镜像,兼容所有系统)
docker pull quay.io/biocontainers/gepard:2.1.0--hdfd78af_0
# 运行Gepard命令行工具(gepardcmd,核心分析命令)
docker run quay.io/biocontainers/gepard:2.1.0--hdfd78af_0 gepardcmd4.2 Conda 安装(适合生物信息学常规环境)
Conda 是生物信息学最常用的包管理工具,适合本地已配置 Conda(Anaconda/Miniconda)环境的用户,安装步骤简洁,运行速度更快。
前提条件
本地已安装 Conda(Miniconda 安装教程:Miniconda 官方教程,轻量更推荐)。
conda和mamba安装与使用-生物信息学工具30_conda 安装mamba-CSDN博客
安装 & 运行命令(直接复制执行,无需修改)
bash
# 创建专属Gepard环境,从bioconda源安装(自动解决所有依赖)
conda create --name gepard -c bioconda gepard -y
conda install bioconda::gepard
# 激活Gepard环境(每次运行前需执行)
conda activate gepard
# 运行Gepard命令行工具
gepardcmd补充:退出 Conda 环境
conda deactivate05 常用命令行
bash
java -cp /mnt/data/getorganelle/share/gepard/dist/Gepard-2.1.jar org.gepard.client.cmdline.CommandLine -seq ./embplant_pt.K105.complete.graph1.1.path_sequence.fasta ./embplant_pt.K105.complete.graph1.1.path_sequence.fasta -matrix /mnt/data/getorganelle/share/gepard/resources/matrices/edna.mat -outfile ./1-1.fasta.png
java -cp /mnt/data/getorganelle/share/gepard/dist/Gepard-2.1.jar org.gepard.client.cmdline.CommandLine -seq ./embplant_pt.K105.complete.graph1.2.path_sequence.fasta ./embplant_pt.K105.complete.graph1.2.path_sequence.fasta -matrix /mnt/data/getorganelle/share/gepard/resources/matrices/edna.mat -outfile ./1-2.fasta.png没有四分体结构的叶绿体

有四分体结构的叶绿体
五种肉苁蓉属植物叶绿体基因组-文献精读25_肉苁蓉全基因组-CSDN博客
13个黄杞属叶绿体基因组-文献精读68_genome varscan插件报错-CSDN博客
GetOrganelle(组装叶绿体)安装与使用-bioinfomatics tools 34-CSDN博客
06 引用
Jan Krumsiek, Roland Arnold, Thomas Rattei, Gepard: a rapid and sensitive tool for creating dotplots on genome scale, Bioinformatics , Volume 23, Issue 8, 15 April 2007, Pages 1026--1028, https://doi.org/10.1093/bioinformatics/btm039