小罗碎碎念
这篇文章报道了一种基于机器学习模型的自动化方法,用于在常规组织病理学图像中检测和分类胃肠道癌症中的三级淋巴结构,并验证了其与患者生存预后的关联。

在这项多中心诊断/预后研究中,开发了一种基于机器学习的计算工具,用于自动化检测和定量评估常规H&E幻灯片上的TLSs,并证实了其在胃肠癌中的生存率关联。
提出的TLSs评分系统可能补充现有的分期系统,并在风险分层中发挥作用。未来的前瞻性验证研究可能会确认这些结果的重复性和广泛适用性。
一作&通讯
| 角色 | 作者姓名 | 单位(中文翻译) |
|---|---|---|
| 第一作者 | Zhe Li | 西北工业大学计算机科学与技术学院 |
| 第一作者 | Yuming Jiang | 斯坦福大学医学院放射肿瘤科 |
| 第一作者 | Bailiang Li | 斯坦福大学医学院放射肿瘤科 |
| 通讯作者 | Ruijiang Li | 斯坦福大学医学院放射肿瘤科 |
本文介绍了一种用于检测和分类胃肠道癌症中三级淋巴结构(TLSs)的可解释机器学习模型的开发和验证。研究表明,该模型能够自动、准确地评估TLSs,并且TLSs的定量评分与患者的生存率显著相关。
-
引言:
- TLSs是与癌症预后良好和免疫治疗响应改善相关的异位淋巴器官。
- 目前TLSs的评估方法存在局限性,如观察者间变异性高、专门成像技术复杂且成本高。
- 本研究旨在基于常规组织病理学图像开发一种自动化的TLSs评估机器学习模型。
-
方法:
- 研究设计遵循TRIPOD报告指南,使用了来自TCGA和SMU的数据集。
- 使用卷积神经网络(ResNet18)进行肿瘤与正常组织的分割。
- 使用基于掩膜区域的卷积神经网络(Mask R-CNN)进行单细胞成像分析,分割和分类单个核细胞。
- 通过图像处理和机器学习模型对TLSs进行分割和分类,并计算每个肿瘤的定量TLS评分。
-
结果:
- 机器学习模型在检测和分类TLSs方面表现出高准确性,TLS1、TLS2和TLS3的分类准确率分别为97.7%、96.3%和95.7%。
- 在6种癌症类型中,TLSs的检测率为40.0%(食管癌)到75.6%(胃癌)。
- TLS评分与生存率显著相关,高TLS评分组的患者生存率显著高于低TLS评分组和无TLSs组。
- TLS评分是独立的预后因素,即使在调整了临床病理变量和肿瘤浸润淋巴细胞后,TLS评分仍与生存率显著相关。
-
讨论:
- 本研究是首个基于常规H&E染色图像开发自动化定量TLS评分系统的大型研究,并在所有主要类型的胃肠道癌症中验证了其临床相关性。
- TLSs的存在与多种癌症类型的良好预后和免疫治疗响应改善相关。
- 该模型的自动化评估方法可以标准化和量化TLSs的评估,克服了传统人工评估的局限性。
-
结论:
- 本研究开发了一种基于机器学习的计算工具,用于自动检测和定量评估常规H&E染色切片中的TLSs,并确认了TLSs与胃肠道癌症生存率的关联。
- 提出的TLS评分系统可能补充当前的癌症分期系统,并有助于风险分层的精细化。
这篇论文展示了机器学习在病理学图像分析中的潜力,并为胃肠道癌症的预后评估提供了新的工具。
代码&数据
代码链接和数据集
数据集
-
The Cancer Genome Atlas (TCGA)
- TCGA-ESCA: 食管癌数据集
- TCGA-STAD: 胃癌数据集
- TCGA-COAD: 结肠癌数据集
- TCGA-READ: 直肠癌数据集
- TCGA-LIHC: 肝细胞癌数据集
- TCGA-PAAD: 胰腺癌数据集
- 这些数据集的数字化H&E全视野图像可以从The Cancer Imaging Archive获取。
-
Southern Medical University (SMU)
- SMU-STAD: 胃癌数据集
- 该数据集包含332名患者的332张数字化H&E全视野图像,由南方医科大学南方医院收集。
代码链接
文章代码未开源,但提到了一些关键的代码和方法:
-
ResNet18 和 Mask R-CNN
- 这些是用于图像分割和分类的深度学习模型。虽然文章中没有提供具体的代码链接,但这些模型的实现可以在公开的深度学习库中找到,如TensorFlow或PyTorch。
- 例如,ResNet18和Mask R-CNN的实现可以在PyTorch的官方GitHub仓库中找到:
-
分类和回归树 (CART) 算法
- 用于TLS分类的机器学习模型。CART算法的实现可以在Python的scikit-learn库中找到:
-
数据分析工具
- 文章提到使用了
lifelines包进行生存分析。lifelines包可以在Python的PyPI上找到:
- 文章提到使用了
一、绪论
第三级淋巴结构(TLSs)是在非淋巴组织中形成的异位淋巴器官,常见于慢性炎症和肿瘤部位。尽管其形成机制尚未完全明了,但TLSs在抗肿瘤免疫反应中扮演着关键角色。研究表明,TLSs的存在与多种癌症类型的良好预后和对免疫治疗的改善反应相关(参考文献4-7)。
目前,检测TLSs的常用和公认方法是采用多重免疫组化或免疫荧光技术对免疫细胞谱系标志物进行组织染色(参考文献8-10)。然而,多重成像因成本高昂、操作复杂、视野受限以及难以规模化,其应用主要限于研究环境。相比之下,苏木精-伊红(H&E)染色在临床病理学中普遍使用,是临床标准。因此,在H&E染色的组织切片上检测TLSs可能成为基于多重成像方法的实用替代。
已有研究基于病理学家的评估,在常规H&E染色的切片上对TLSs进行评价(参考文献11)。但这种方法的耗时耗力以及手动和定性评估的局限性,特别是观察者间的变异性,导致了对此方法的需求(参考文献12)。目前,亟需验证的方法来实现对H&E图像中TLSs的标准化和定量评估。
机器学习技术在提取数字病理学数据中的临床相关信息方面日益得到应用(参考文献13-15)。多项研究证明了深度学习在基于H&E图像进行癌症自动诊断、分级、遗传改变预测和预后评估方面的可行性(参考文献16-28)。本研究旨在开发一个可解释的机器学习模型,用于在整张H&E切片图像中自动检测、计数和分类TLSs,并进一步评估TLSs在多种胃肠道癌症中的预后价值。
二、方法
2-1:研究设计
本研究提出了一种基于机器学习的计算成像分析流程,用于在常规H&E染色的整张切片图像中全自动和定量评估TLSs(包括计数和特征描述)。进一步评估了TLSs在六种常见人类消化系统癌症:食管癌、胃癌、结肠癌、直肠癌、肝癌和胰腺癌中的预后意义。
eFigure 1 描述了一个用于自动化检测、分类和定量评估三级淋巴结构(TLS)的计算流程。

这个流程是基于人工智能技术的,并且专门针对用苏木精-伊红(hematoxylin-eosin, H&E)染色的全切片图像(whole-slide images, WSIs)。
-
ResNet18模型:
- 作用: 这个模型用于从全切片图像中分割出肿瘤区域。ResNet18是一个深度残差网络,它通过学习图像中的特征来区分肿瘤和非肿瘤区域。
- 输入: 完整的H&E染色的全切片图像。
- 输出: 图像中肿瘤区域的二维分割图(mask),这有助于后续步骤中对特定区域内的细胞进行分析。
-
Mask RCNN模型:
- 作用: 这个模型在由ResNet18模型确定的肿瘤区域内进一步进行淋巴细胞的分割。Mask RCNN是一种先进的目标检测算法,它不仅可以识别图像中的对象,还可以生成对象的精确掩码。
- 输入: 由ResNet18模型输出的肿瘤区域。
- 输出: 肿瘤区域内每个细胞的分割图,特别是淋巴细胞,为TLS的识别和分类提供基础。
-
分类和回归树(CART)算法:
- 作用: 这个算法用于根据从Mask RCNN获得的淋巴细胞分割图来分类个别TLS。CART是一种决策树学习技术,它可以将数据分成不同的类别,这里用于将TLS分为不同的成熟状态。
- 输入: 淋巴细胞的分割图以及可能的一些图像特征,如TLS的面积、圆度和淋巴细胞密度的偏斜度。
- 输出: 每个TLS的分类标签,根据其成熟度将其分为TLS1、TLS2或TLS3。
整个计算流程是自动化的,意味着一旦设置了模型和算法参数,它就可以在不需要人工干预的情况下处理全切片图像,从而提高效率和减少人为误差。此外,这种基于人工智能的方法可以提供TLS的定量评分,有助于进一步的临床研究和可能的预后评估。
简而言之,首先使用具有深度残差学习的卷积神经网络(ResNet18)来分割整张切片图像中的肿瘤与正常组织。接着,通过基于掩码区域的卷积神经网络(R-CNN)进行单细胞成像分析,以分割和将单个细胞核分类为三种细胞类型:淋巴细胞、肿瘤细胞和其他非恶性细胞。
基于淋巴细胞密度图,进行图像处理并训练机器学习模型以获得TLSs的分割和分类。最后,计算每个肿瘤的定量TLS得分,并将其与患者预后相关联,并与基因表达谱进行相关性分析。
关于图像预处理、ResNet18和Mask R-CNN的训练以及与分子特征的相关性分析的详细描述,请参考补充材料1的eMethods。
2-2:患者与数据集
在这项国际多中心研究中,回顾性地收集并分析了1924名患者的整张H&E切片图像和临床数据。
包括了7个独立队列:TCGA食管癌(TCGA-ESCA)、胃癌(TCGA-STAD)、结肠癌(TCGA-COAD)、直肠癌(TCGA-READ)、肝癌(TCGA-LIHC)、胰腺癌(TCGA-PAAD)和SMU-STAD。
TCGA队列的数字化H&E整张切片图像公开可用。从癌症成像档案库中检索了1813张整张切片图像,并对1592名患者的1660张图像进行了分析。TCGA中的大多数患者有1张切片;对于有多张切片的患者,分析了所有可用切片。
此外,从中国广州南方医院的SMU收集了332名胃癌患者的332张数字化H&E整张切片图像。所有样本均来自未经治疗的原发性肿瘤切除标本。仅包括由甲醛固定、石蜡包埋的肿瘤切片生成的诊断性切片。详细的纳入和排除标准在补充材料1的eFigure 2中描述。

2-3:TLSs的分割与分类
本研究开发了一个计算流程,用于自动识别、分割和分类单个TLSs。
首先,基于先前获得的细胞核分割和淋巴细胞掩码,作者计算了16×16μm²网格上每个单位面积的淋巴细胞数量,生成了淋巴细胞密度图。然后,对这些密度图应用阈值处理和形态学图像处理(开启操作),并在排除过小的淋巴细胞簇(即面积<0.0384 mm²)后,获得了最终的TLS分割掩码。
在本研究中,根据成熟程度定义了TLSs的3个等级:淋巴样聚集物、初级滤泡和带有生发中心的次级滤泡。作者训练了一个机器学习模型来按等级分类单个TLSs。
为此,作者从TCGA-STAD队列中选择了45名患者,并由一名病理学家(Y.J.)手动标记了总共865个TLSs,将其分类为3个TLS等级之一。整个数据集被随机划分为训练集(379个TLSs)和测试集(486个TLSs)。
考虑到TLS2和TLS3倾向于具有圆形形状并且通常比TLS1大,且TLS3具有独特的生发中心以及较低的淋巴细胞密度,作者为每个TLS计算了3个特征:面积、圆度(即面积乘以4π与周长的平方之比)和每个TLS的淋巴细胞密度的偏度。
使用这些数据,作者使用分类和回归树(CART)算法训练了一个模型。作者使用Python编程语言版本3.6.11的scikit-learn包训练CART,使用默认参数设置(criterion=gini;splitter=best;min_samples_split=2)。使用训练集的5折交叉验证确定树的最大深度为4。
鉴于TLS3的相对重要性,TLS1、TLS2和TLS3的类权重经验性地设置为1、2和3。
2-4:TLSs的定量评分分析
首先,作者计算了每个等级(TLS1、TLS2和TLS3)的TLS总面积和肿瘤面积。
对于每位患者,计算了三个单独的TLS得分,即每个等级的TLS总面积除以肿瘤面积。接着,作者定义了总体TLS得分为TLS面积除以肿瘤面积的线性加权总和,如下所示:
T L S 得分 = ( w 1 × a r e a T L S 1 + w 2 × a r e a T L S 2 + w 3 × a r e a T L S 3 ) / a r e a t u m o r TLS得分 = (w1 × areaTLS1 + w2 × areaTLS2 + w3 × areaTLS3)/areatumor TLS得分=(w1×areaTLS1+w2×areaTLS2+w3×areaTLS3)/areatumor
其中,areaTLS1、areaTLS2、areaTLS3和areatumor分别是TLS1、TLS2、TLS3和肿瘤的总面积,w1、w2和w3是对应的权重。
为了确定最优权重,作者在TCGA-STAD队列中进行了TLS1得分、TLS2得分和TLS3得分的Cox回归分析,以评估总体生存率。
2-5:统计分析
作者使用总体准确性和混淆矩阵来评估TLS分类的性能。
通过TLS得分与生存结果的关系来评估单个和总体TLS得分的预后价值。
总体生存期定义为从诊断到死亡或最后一次随访的时间。无进展生存期定义为从诊断到疾病进展、死亡或最后一次随访的时间。使用Cox比例风险模型进行单变量和多变量分析。多变量分析中包括了临床和病理变量,如肿瘤分期和分级。使用Kaplan-Meier分析和log-rank检验来评估按风险组分层患者的生存情况。作者使用Harrel一致性指数(C指数)作为评估预后预测性能的指标。双侧P值小于0.05被认为具有统计学意义。
数据分析在2021年6月至2022年3月之间进行,使用Python编程语言中的lifelines包版本0.25.11。
三、结果
3-1:患者特征
本研究共纳入1924名患有胃肠道癌症的患者,跨越7个队列。
患者的年龄中位数(四分位距)从SMU-STAD队列的57岁(49-64岁)到TCGA-COAD队列的68岁(58-77岁);性别比例从TCGA-COAD队列的男性患者214名(52.3%)到TCGA-ESCA队列的男性患者134名(86.5%)。
在大多数癌症类型中,AJCC分期II和III的疾病占据了大多数诊断(从TCGA-LIHC队列的164名(45.2%)到TCGA-PAAD队列的148名(84.6%))。TCGA和SMU的胃癌数据集在临床病理特征分布上相似。
7个队列的基线特征在表中进行了总结。

3-2:准确检测肿瘤和肿瘤浸润淋巴细胞的分类
ResNet18模型在肿瘤检测中实现了超过0.99的样本外曲线下面积(95% CI,0.98-1.00)。
作者从TCGA-STAD数据集的20名随机选择的胃癌患者中手动分割了15368个细胞核,并在140个图像块中对每个细胞核进行了肿瘤细胞、淋巴细胞或其他细胞的标记。
Mask R-CNN模型在细胞核检测中实现了91.1%的准确率(95% CI,90.6%-91.5%),精确率和召回率分别为97.1%(95% CI,96.8%-97.4%)和93.7%(95% CI,93.3%-94.1%)。
在细胞核分类中,训练数据集和测试数据集在肿瘤细胞上分别观察到95.8%(95% CI,95.5%-96.1%)和95.37%(95% CI,95.0%-95.7%)的高准确率,在淋巴细胞上分别为98.4%(95% CI,98.2%-98.6%)和95.7%(95% CI,95.4%-96.0%)(补充材料1的eFigure 3)。

3-3:TLS的准确分类
图1展示了将每个TLS分类为TLS1、TLS2或TLS3的最优决策树。

所提出的模型在训练数据集中准确率高,TLS1的分类准确率为373/379(98.4%;95% CI,97.1%-99.7%),TLS2为371/379(97.9%;95% CI,96.5%-99.3%),TLS3为369/379(97.4%;95% CI,95.8%-99.0%)。在测试数据集中也观察到了高TLS分类准确率(TLS1:97.7%;95% CI,96.4%-99.0%;TLS2:96.3%;95% CI,94.6%-98.0%;TLS3:95.7%;95% CI,93.9%-97.5%)。
图2A和B展示了训练和测试数据集中TLS分类的混淆矩阵。

一些额外的TLS分类图像显示在补充材料1的eFigure 4中,其中TLS1、TLS2和TLS3分别用红色、蓝色和绿色轮廓线表示,这有助于区分不同成熟阶段的TLS。

3-4:基于整张切片图像的TLS计数和定量评估
eFigure 5(补充材料1)显示了至少检测到一个TLS的肿瘤百分比。

在所有6种癌症类型中,TLS1比其他类型的TLS更常见,在TCGA-STAD中有高达69%的肿瘤至少检测到一个TLS1(242/353肿瘤[68.6%])。检测到任何类型TLS的肿瘤比例从TCGA-ESCA的62/155(40.0%)到TCGA-STAD的267/353(75.6%),表明TLS在胃肠道癌症中高度流行。
图3A显示了按等级分布的TLS得分。

作者还计算了TLS密度,即单位肿瘤面积内的TLS数量,其分布与TLS得分相似(补充材料1的eFigure 6)。

在6种癌症类型中,SMU-STAD队列的定量得分和TLS密度最高,而TCGA-READ的得分最低。所有患者的平均(标准差)TLS大小为肿瘤面积的0.009(0.023),而检测到任何类型TLS的患者的平均(标准差)TLS大小为肿瘤面积的0.016(0.029)(图3A)。在罕见情况下,TLS大小占肿瘤面积的5%至10%。三个单独TLS得分之间的皮尔逊相关系数较小到中等,表明这些得分可能具有互补性(补充材料1的eFigure 7)。
为了总结每位患者的TLS得分,作者线性组合了三个单独的TLS得分。
最优的相应权重分别为TLS1 0.81,TLS2 0.84,TLS3 1.00。图3B显示了7个队列中总体TLS得分的分布。

与单个TLS得分相似,胃癌的总体TLS得分最高,而肝癌的TLS得分最低;食管癌、结肠癌、直肠癌和胰腺癌的TLS得分介于两者之间。然后,作者评估了TLS得分与肿瘤分期或分级之间的关系;作者没有观察到除了TCGA-STAD和TCGA-READ癌症之外的其他关联,其中II和III期疾病具有更高的TLS得分(补充材料1的eTable 1)。

作者还评估了具有多个切片的患者TLS得分的跨切片变异。TLS得分的平均(标准差)变异系数(即TLS得分标准差与平均值的比率)从TCGA-LIHC的0.30(0.35)到TCGA-STAD的0.57(0.39),表明跨切片的变异从小到大(补充材料1的eTable 2)。

3-5:TLS分数在癌症类型中的预后结果分析
本研究评估了TLS分数在6种癌症类型和7个队列中的预后结果。
总体TLS分数将患者分为3个不同的风险组(图4;补充材料1的eFigure 8)。

TLS分数较高的患者与TLS分数较低的患者相比,总体生存率显著提高(总体危险比[HR],0.27;95% CI,0.18-0.41;P < .001);这些患者再次比未检测到TLS的患者有更好的生存率(HR,0.65;95% CI,0.56-0.76;P < .001)。与未检测到TLS的患者相比,TLS分数较高的患者的生存差异更大(HR,0.18;95% CI,0.12-0.27;P < .001)。
总体TLS分数的预后结果在无进展生存期中也呈现出相同的模式(补充材料1的eFigure 9)。

TLS分数较高的患者与TLS分数较低的患者相比,无进展生存期显著提高(HR,0.56;95% CI,0.43-0.72;P < .001),后者又比未检测到TLS的患者有更好的无进展生存期(HR,0.72;95% CI,0.63-0.83;P < .001);TLS分数较高的患者也比未检测到TLS的患者有更好的无进展生存期(HR,0.41;95% CI,0.32-0.52;P < .001)。
在所有7个队列中,3个单独的TLS分数在单变量分析中与所有癌症的总体生存率相关,除了TCGA-ESCA、COAD和PAAD中的TSL2以及样本数量最少的直肠癌(补充材料1的eTable 3)。

例如,对于TCGA-STAD,TLS1的HR为0.55(95% CI,0.41-0.75;P = .001),TLS2为0.53(95% CI,0.34-0.82;P = .005),TLS3为0.18(95% CI,0.07-0.51;P = .001)。在多变量分析中,所有3个分数在组合数据集中仍然显著,其中TLS3是生存的最强预测因子(所有数据集合并:HR,0.25;95% CI,0.15-0.42;P < .001)。
与单独的TLS分数相比,总体TLS分数在生存预测中实现了更高的准确性(补充材料1的eFigure 11)。

重要的是,TLS分数在预后预测中的表现优于TLS密度,这表明定量评分可能比简单的TLS计数提供更多信息(补充材料1的eFigure 12)。

为了研究不同个体TLS分数的权重,作者使用SMU-STAD、TCGA-PAAD或合并数据集重新训练了一个线性模型。总体TLS分数保持稳定,皮尔逊相关系数为0.93或更高,预后模式与原始结果相似(补充材料1的eFigure 22)。

总体TLS分数预测总体生存率的精度与或高于肿瘤分期(补充材料1的eFigure 13)。
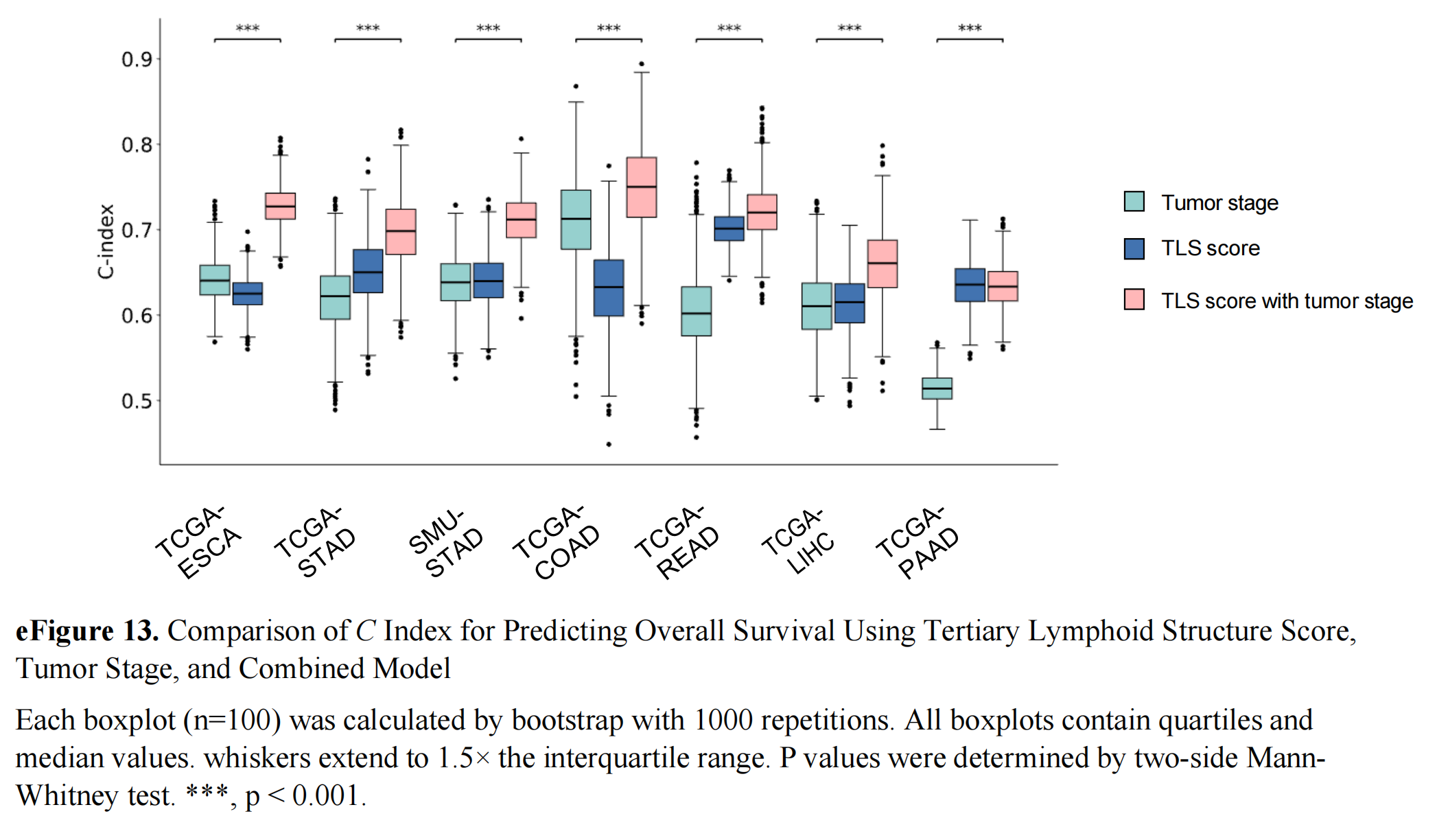
对于直肠癌和胰腺癌,TLS分数在生存预测中优于分期。为了进一步提高预后预测,作者将肿瘤分期与TLS分数相结合,分别赋予最佳权重为-0.51和1.36。与肿瘤分期相比,结合模型在生存预测中具有显著提高的C指数(补充材料1的eFigure 13)。
在包括临床病理变量和肿瘤浸润淋巴细胞密度的多变量分析中,总体TLS分数在所有7个队列中都是一个独立的预后因素(例如,对于结肠癌:HR,0.11;95% CI,0.02-0.47;P = .003)(补充材料1的eTables 4-10)。
在每个由年龄、性别、肿瘤分期和分级定义的患者亚组中,总体TLS分数与总体生存率相关(补充材料1的eFigures 14-20)。对于相同疾病分期的患者,总体TLS分数在大多数癌症类型中进一步细分患者(补充材料1的eFigure 21)。
最后,作者研究了与基于成像的TLS分数相关的分子特征,并开发了一个包含11种细胞因子的基因表达签名(补充材料1的eFigures 23-24和eTable 11)。结果进一步表明,TLS分数在1858名胃癌和结直肠癌患者中具有独立的预后价值(补充材料1的eFigures 25-27)。对于详细描述个体TLS分数的权重、分子相关性和TLS分数的基因表达签名,请参阅补充材料1的eResults。
四、讨论
在本诊断/预后研究中,作者开发了一个可解释的机器学习模型,用于基于常规H&E染色的整张切片图像自动检测、计数和分类TLSs。此外,作者提出了一个定量评分系统来评估TLSs,并确认了其在1924名国际多中心队列患者中,涵盖6种常见胃肠道癌症中的独立预后价值。最后,作者开发了一个基于成像的TLS分数的基因表达签名,进一步确认了其预后价值。
据作者所知,这是首次也是最大规模的研究,基于常规H&E染色图像开发了一个自动定量TLS评分系统,并在所有主要胃肠道癌症类型中验证了其临床相关性。最近的研究表明,TLSs的存在与多种癌症类型的良好预后和对免疫治疗的改善反应相关(参考文献4-7)。
在以往的研究中(参考文献4-7),TLSs是通过多重免疫组织化学染色或免疫荧光成像来识别的,这些方法并不常规使用。在本研究中,作者基于常规H&E切片开发了一种计算方法来评估TLSs,这种方法可能在临床环境中广泛适用。
作者发现,TLSs在6种胃肠道癌症类型中都有利于预后。
这些发现与以前关于胃肠道癌症的研究(参考文献29,30)以及其他癌症的研究(参考文献8-10,31)一致。值得注意的是,在作者的TLS评分中,具有生发中心的TLS3权重最大,这表明成熟的TLSs可能在抗肿瘤免疫反应中发挥最重要的作用,这与以前的研究一致(参考文献4-7)。
相比之下,与淋巴细胞聚集(例如TLS1)相关的肿瘤浸润淋巴细胞的密度,在调整TLS评分后并没有独立的预后关联。重要的是,作者发现TLS评分与已确立的预后因素,包括肿瘤分期和分级,是独立的,这表明其使用可能与当前分期系统的进一步细化和改善风险分层有关。
作者方法的一个重要优势是自动计数和定量描述TLSs。以往的工作依赖于病理学家对H&E染色切片上TLSs的手动和定性评估,这种方法被发现是不准确的,并且容易受到观察者间变异性的影响(参考文献12)。在本研究中,作者开发了一个自动计算流程来评估TLSs,这可能允许对TLSs进行标准化和定量评估。
虽然深度学习在数字病理学中显示出有前景的性能(参考文献13,14),但大多数研究都采用了基于补丁或瓦片的图像分析方法。由于TLSs在大小、密度和形态上高度可变,传统的基于补丁的方法面临重大挑战。
在这里,作者开发了一个基于深度学习的单细胞分析工具,这可能允许在整张切片图像上自动分割和分类肿瘤浸润淋巴细胞。通过量化淋巴细胞的时空分布,作者开发了一个准确且可解释的模型,用于根据其成熟状态对TLSs进行分类。
作者发现,平均(标准差)TLS大小不到肿瘤面积的1%,尽管在罕见情况下,这达到了5%到10%。这表明,基于图像的TLS检测在通常用于转移性癌症患者的少量活检中可能表现出更大的变异。这一局限性可以通过依赖于分泌蛋白的分子方法来克服,例如相关的细胞因子和趋化因子。
为此,作者开发了一个包含11种基因的TLS签名。与以前包含与TLSs正相关的基因的签名不同,作者的签名不仅包含与TLS形成和功能有关的已知基因(例如CXCL13、CXCL11、CXCL10),还包含与抑制TLSs发展的免疫抑制性肿瘤微环境相关的基因(例如TGFB2和VEGFB)。
五、限制
作为一个回顾性研究,它可能存在选择偏差的风险,尽管作者努力纳入具有不同特征和地理分布的所有合格患者。在胃癌中,尽管TCGA-STAD和SMU-STAD队列中TLSs的数量存在一些差异,但预后模式是相似的。
在本研究中,TLS的识别和分类是基于病理学家对H&E图像的评估进行的。理想情况下,TLS检测的标准是基于免疫组织化学。由于TCGA队列中没有可用的信息,作者无法评估某些已确立的预后因素,如淋巴血管侵犯或肝硬化,与生存结果的相关性。
此外,作者的研究没有包括接受免疫治疗的患者,因此作者无法确认TLS分数对免疫治疗反应的预测价值。
六、结论
在这项涉及1924名患者的多中心诊断/预后研究中,作者开发了一个基于机器学习的计算工具,用于自动检测和定量评估常规H&E切片上的TLSs,并确认了TLSs与胃肠道癌症生存的相关性。
提出的TLS评分系统可能补充当前的分期系统,并与风险分层的精细调整相关。可能需要进行前瞻性验证研究,以确认作者的结果是否在更广泛的病人人群中可重复和可推广。