大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
恒河猴是神经科学研究中常用的模型动物,其大脑结构和功能与人类大脑相似。大脑中复杂的遗传网络是灵长类动物行为、认知和情感的基础,一直是神经科学的核心。大脑的综合转录组图谱是更深入地了解大脑功能的关键,这将有助于神经系统疾病研究。
尽管已有人类大脑的转录组图谱,但关于恒河猴大脑的转录组信息较少。先前转录组学研究在恒河猴大脑中,要么具有有限的采样脑区,要么狭隘地集中在非编码 RNA 上。此外,开放染色质状态在基因表达调控中的作用尚未在恒河猴大脑中得到充分研究。恒河猴大脑中是否存在区域或个体相关的开放染色质模式仍然未知。
近日,北京大学第三医院于洋教授为共同通讯在Nature子刊《Nature Communications》杂志发表了题为"Transcriptomic and open chromatin atlas of high-resolution anatomical regions in the rhesus macaque brain"的科研成果。研究通过 ATAC-seq 与 RNA-seq 绘制了恒河猴大脑的解剖学综合转录组图谱和大脑皮层的染色质可及性图谱。首先对恒河猴大脑52个区域的416个样本进行了RNA测序(RNA-seq),揭示不同大脑区域之间的转录组差异,并比较恒河猴大脑与人类大脑在不同大脑区域的转录组学特征。此外利用ATAC-seq技术分析了恒河猴大脑的不同大脑皮层区域和海马CA1(cornu Ammonis 1)的开放染色质区域,揭示了特定区域的染色质可及性模式。

**题目:**Transcriptomic and open chromatin atlas of high-resolution anatomical regions in the rhesus macaque brain(恒河猴大脑高分辨率解剖区域的转录组和开放染色质图谱)
**期刊:**Nature Communications
影响因子: IF 14.7
**技术平台:**ATAC-seq、RNA-seq(易基因优势技术)
研究团队通过对8个恒河猴大脑的52个区域的416个脑样本的转录组进行了表征,鉴定出与特定大脑区域相关的基因模块,如大脑皮层、垂体和丘脑。研究发现了9703个新的基因间转录本,包括1701个编码转录本和2845个长链非编码RNA(lncRNA)。大多数新转录本仅在特定大脑区域或特定皮层区域表达。进一步使用ATAC-seq技术分析了恒河猴大脑海马CA1和不同大脑皮层区域的开放染色质区域,揭示了CA1和皮层特异性开放染色质区域。研究为关于恒河猴大脑基线转录组和开放染色质图谱的日益增长的知识体系增添了新内容。
实验方案
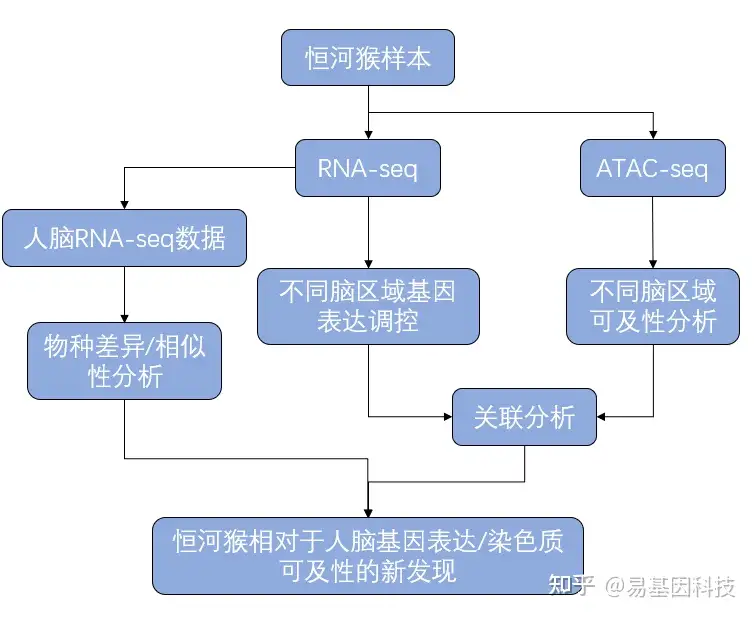
为了构建恒河猴大脑的全面转录组图谱,研究分析了 8 只恒河猴(总共 416 个大脑样本,图 1)中 52 个大脑区域的转录组。还检测了33个脑样本的6个脑区区域的ATAC-seq。
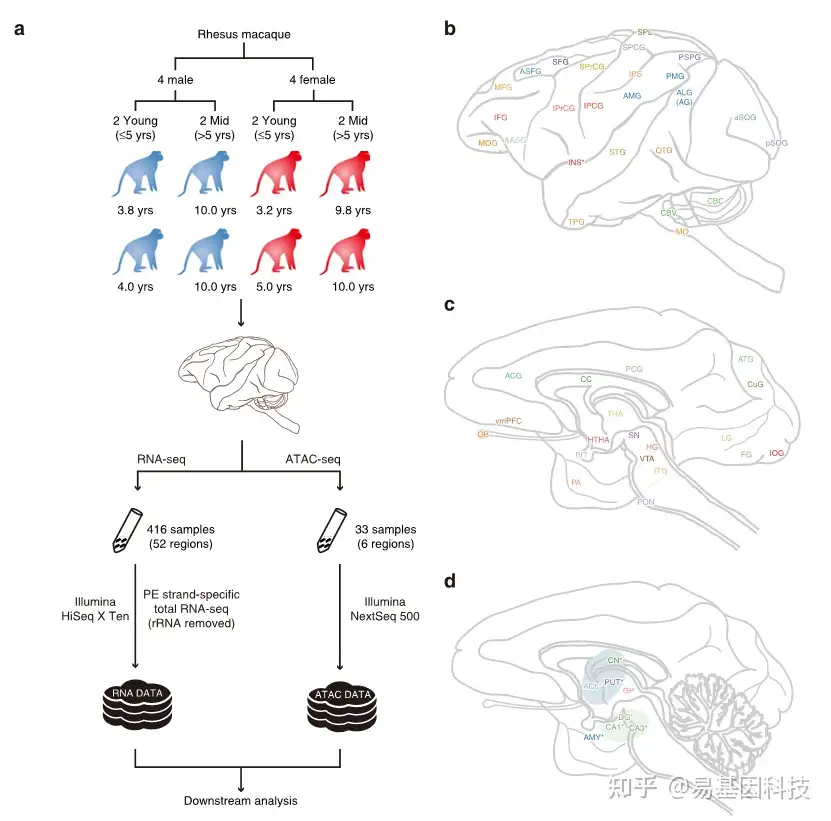
图1:实验方案,恒河猴大脑图
结果图形
(1)人类和猕猴大脑之间的相似特征
为了阐明人类和恒河猴大脑之间区域表达谱的保守性,将RNA-seq数据与从PsychEncode获得的人脑数据集进行了比较。与恒河猴类似,t-SNE分析显示,来自人脑的大脑皮层倾向于按个体聚集,而纹状体、丘脑、小脑皮层等更深的结构则按区域聚集。人和恒河猴的大脑在纹状体、丘脑和小脑皮层中表现出相似的表达模式。同源基因的相关性分析确定了人类和恒河猴之间在相同大脑区域的相似性最高,尤其是在非皮质区域。
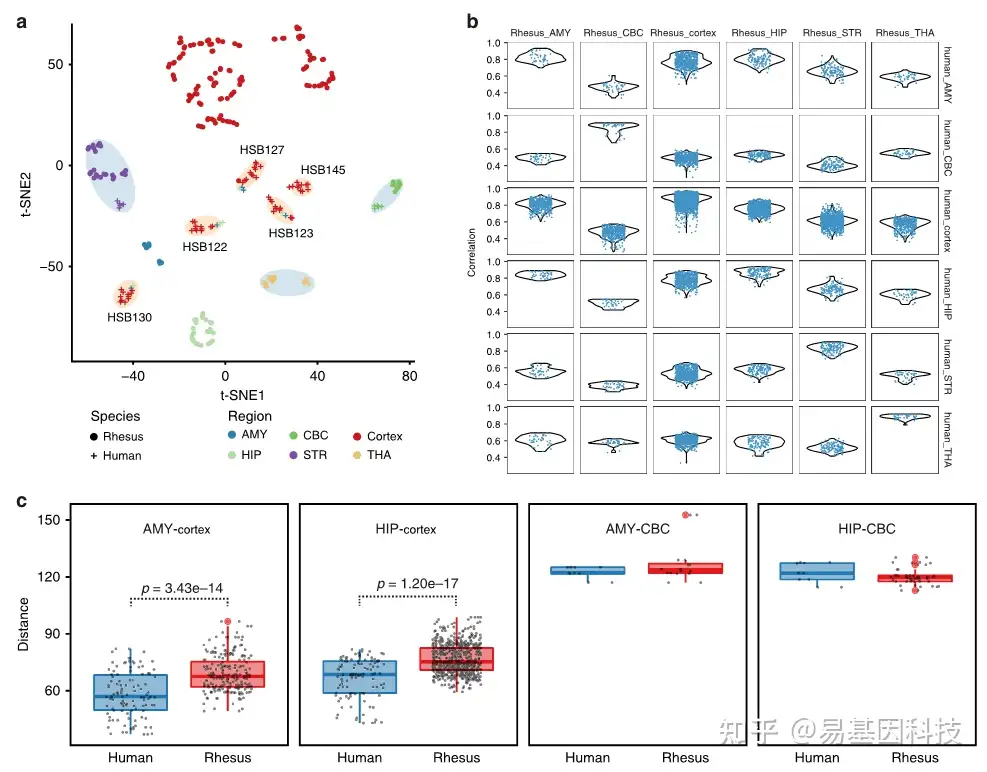
图2:人类和恒河猴大脑之间的相似表达谱。
(2)共表达分析发现区域相关模块
为了探索跨脑区的共表达和基因相互作用网络,使用WGCNA确定了59个基因共表达模块19分析。一些模块与特定的大脑区域高度相关,例如模块M2,它与垂体(PIT)区域相关(Pearson的r0.987,p值<0.001)。
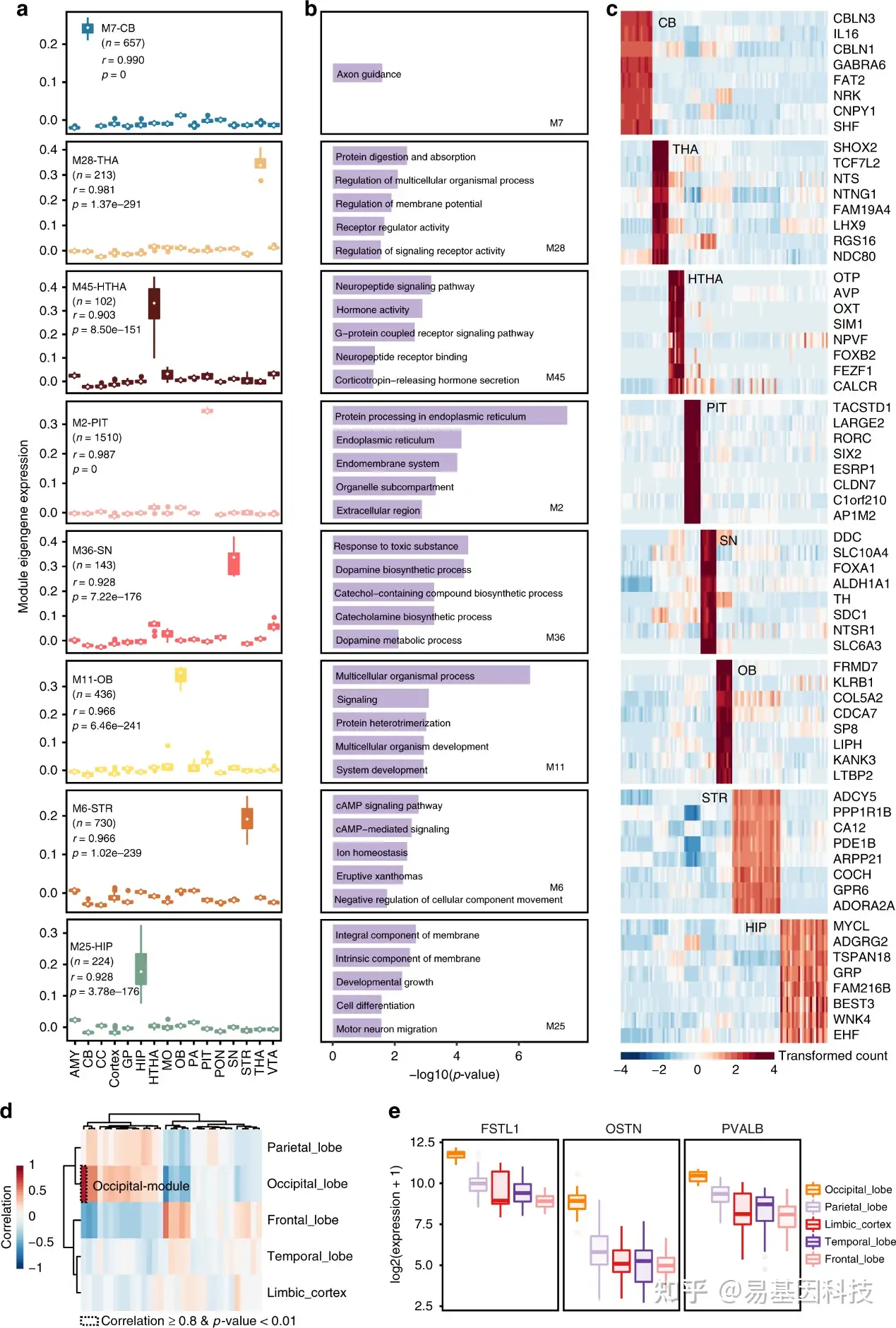
(3)与大脑区域和个体相关的新转录本
大约 30% 的 RNA-seq 读数可以映射到当前恒河猴基因组注释的外显子区域。该外显子映射率与人类和黑猩猩大脑上的 RNA-seq 数据集相当。大多数组装的转录本与已知基因的外显子重叠。
为了探索它们的潜在功能,这些新的编码转录本在计算机翻译后与来自 Swiss-Prot 的人类蛋白质序列比对。接下来比较了 Rh 脑不同区域新转录本的表达水平。
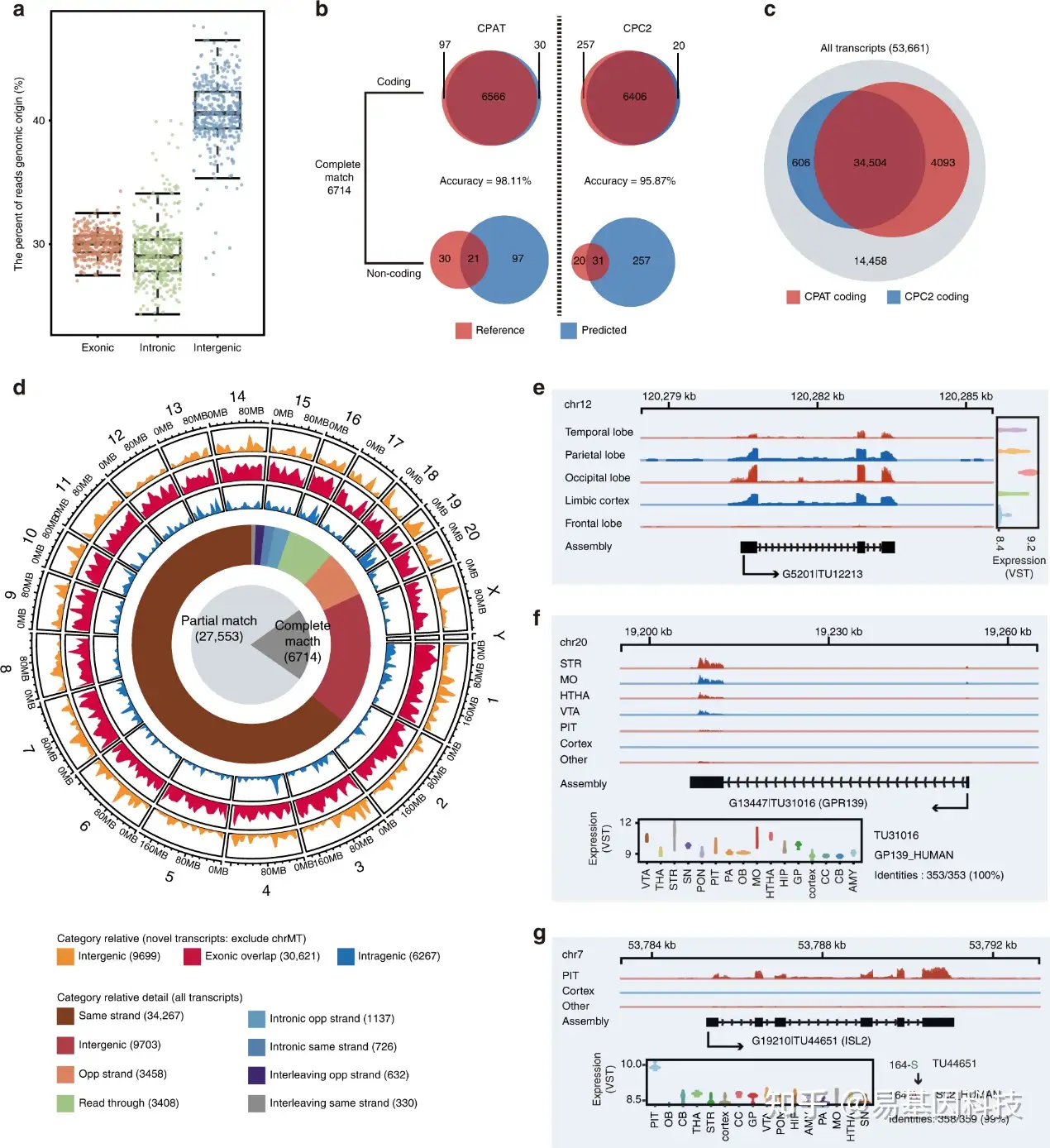
图4:与大脑区域和个体相关的新转录本。与大脑区域相关的新型lncRNA
(4)区域相关染色质可及性模式
对每个区域进行ATAC-seq peaks检测。首先比较了多个大脑区域之间共有的可重复peaks基因组特征分布。发现区域特异性peaks通常分布基因间或内含子区域。同时,更保守的ATAC peaks倾向于在启动子中富集。在所有共有peaks中,只有7.4%在所有6个区域开放。这些peaks中的大多数(58.99%)都在TSS的±1kb范围内,表明在不同大脑区域之间共有的开放ATAC peaks主要代表启动子,而CA1和PCG之间的差异peaks大多是基因间或内含子,代表远端调控元件,其与TFs和与海马和皮层相关的基因相关。
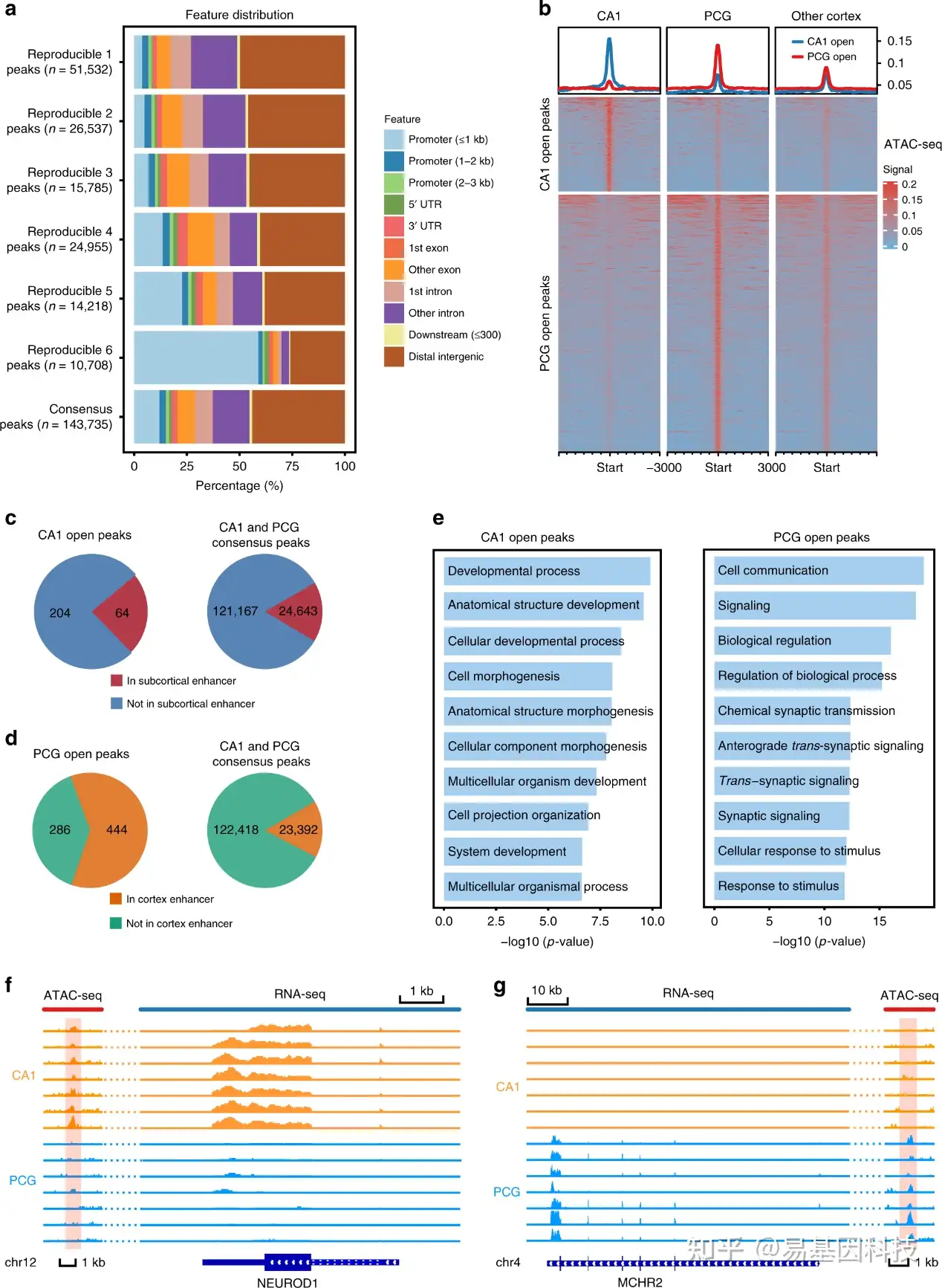
图5:染色质可及性的差异分析。
研究思路总结
易基因染色质可及性-转座酶易接近染色质测序(ATAC-seq)
ATAC-seq(Assay for Transposase-Accessible Chromatin with high throughput sequencing)是通过使用高通量测序对转座酶可接近性核染色质区域进行分析的一种创新表观遗传学研究技术。该技术通过转座酶对某种特定时空下开放的核染色质区域进行切割,进而获得在该特定时空下基因组中所有活跃转录的调控序列。
真核生物的DNA并不是裸露的,而是被包装成核小体形成串珠状结构并进一步被折叠、包装。而基因的转录,需要将这种高级结构解开,使DNA成为可以使各种转录机器与其结合的裸露状态,即形成开放染色质区域。如何鉴定开放染色质区域呢?传统的方法主要是借助和DNase-Seq、MNase-Seq及ChIP-seq。但这些方法需要的起始细胞量较大,对于少量样本和珍稀样本可行性不高。ATAC-Seq是一种新型的研究开放染色质的技术,利用Tn5转座酶进入并切割裸露的DNA,并同时连接上特异性的测序接头。因为切割和加接头一步完成,因此该技术可大大降低所需细胞起始量。
技术优势:
(1)所需细胞起始量低。
(2)应用范围广,适用于大部分物种及细胞类型。
实验策略:

信息分析:
易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化)和表观转录组学(m6A、m5C、m1A、m7G、ac4C)、染色质结构与功能组学技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900。
参考文献:
Yin S, Lu K, Tan T, Tang J, Wei J, Liu X, Hu X, Wan H, Huang W, Fan Y, Xie D, Yu Y. Transcriptomic and open chromatin atlas of high-resolution anatomical regions in the rhesus macaque brain. Nat Commun. 2020 Jan 24;11(1):474. pii: 10.1038/s41467-020-14368-z. doi: 10.1038/s41467-020-14368-z. PubMed PMID: 31980617.