在生物医学研究领域,组织病理学图像和组学数据的整合一直是重大挑战。传统计算方法往往将这两类数据分开分析,导致宝贵的空间分子信息难以被充分利用。随着空间转录组学和单细胞RNA测序技术的发展,研究人员能够获得组织切片中基因表达的空间分布信息,但如何将这些分子数据与传统的染色图像关联起来,仍缺乏系统性的解决方案。目前的计算模型要么专注于组学数据,要么专注于图像分析,缺乏将两者整合的统一多模态AI模型。
针对这一关键问题,来自休斯顿卫理公会研究所等机构的研究团队开发了名为OmiCLIP的视觉-组学基础模型,并构建了Loki分析平台。这项研究于2025年5月29日发表在《Nature Methods》上,通过创新的跨模态学习方法,成功搭建了连接组织形态学与分子特征的桥梁,为多模态组织分析提供了全新工具。
【文章核心方法】
-
OmiCLIP模型开发:这是一个视觉-转录组学基础模型,通过组织切片将H&E图像和转录组学联系起来。研究者们将转录组学数据转换为"句子",通过连接每个切片中高表达基因的符号来实现。
-
ST-bank数据集的构建:研究者们策划了一个包含220万对组织图像和转录组学数据的数据集,涵盖了32个器官,用于训练OmiCLIP模型,整合组织学和转录组学。
-
Loki平台的开发:基于OmiCLIP模型构建的一个多模态分析平台,旨在整合组织病理学图像(H&E染色图像)和转录组学数据,为生物医学研究提供强大的分析工具,它提供了五个关键功能:组织对齐、通过批量RNA测序或标记基因进行组织注释、细胞类型分解、图像-转录组学检索和从H&E染色图像预测空间转录组学基因表达。
一、OmiCLIP模型框架
-
数据集构建:研究者们构建了一个名为ST-bank的数据集,包含220万对组织图像和转录组学数据,涵盖了32种器官类型。这些数据来自113项研究,包括健康、癌症、心力衰竭和阿尔茨海默病等多种病理状态。
-
数据预处理:为了消除批次效应,研究者采用了基于排名的策略,而不是直接使用原始读数计数或标准化的基因表达值。将Ensembl基因ID转换为基因符号,并去除了管家基因。然后,将基因符号按表达水平从高到低排序,并构造成句子,用于文本编码器。
-
模型架构:OmiCLIP模型基于CoCa框架,包含一个图像编码器、一个文本编码器和一个多模态融合解码器。通过对比学习,将图像和转录组学模态对齐到一个共同的表示空间中,使得配对的图像和转录组学嵌入向量在该空间中尽可能相似。
-
模型训练:使用ST-bank数据集对OmiCLIP进行训练,通过最大化配对的H&E图像和转录组学数据之间的相似性,优化模型参数。

图1 通过对比学习使用配对图像-转录组学数据集预训练OmiCLIP模型的工作流程
二、Loki多模态平台
-
Loki Align:用于多节段组织对齐,包括ST-to-ST对齐、H&E图像-to-H&E图像对齐和H&E图像-to-ST数据对齐。
-
Loki Annotate:用于通过批量RNA-seq数据或标记基因对组织H&E图像进行注释。
-
Loki Decompose:用于从转录组学或组织学中分解细胞类型,参考单细胞RNA-seq数据。
-
Loki Retrieve:用于从H&E染色图像中检索转录组学数据。
-
Loki PredEx:用于从H&E染色图像预测空间转录组学基因表达。

图2 Loki平台的功能模块
三、验证结果与性能优势
- OmiCLIP模型鲁棒性
(1)抗图像干扰:添加高斯噪声后,图像-转录组相似度仅下降8%(PLIP下降23%)。
(2)抗测序深度差异:在低测序深度(615 UMI)下仍保持高对齐精度(相似度>0.7)。
- Loki平台性能对比
Loki平台提供多种类型功能,其组织对齐功能与现有工具如PASTE、GPSA比较,PCC提升40%(肠组织数据达0.83),解决空间扭曲问题(图3)。
在组织注释功能方面,Loki可结合基因标记与语言描述,F1分数达0.96(CRC7K数据集),与现有方法如OpenAI CLIP, PLIP相比,召回率提升至93%。
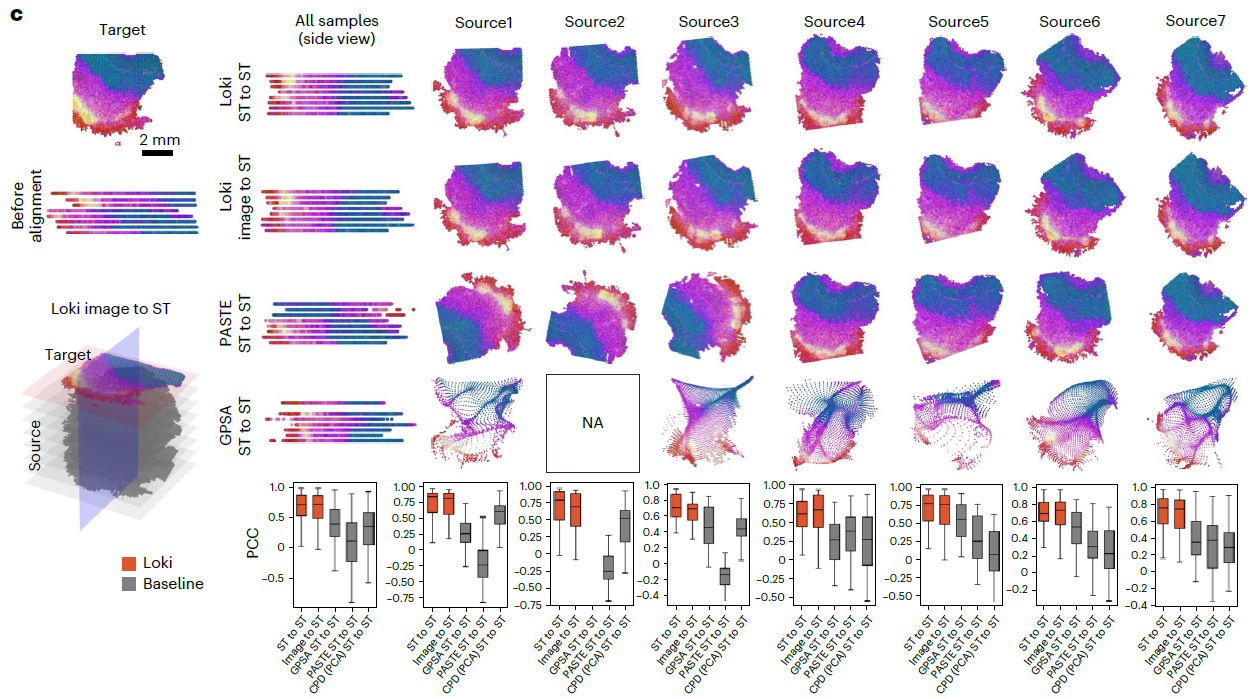
图3 Loki平台组织对齐功能的性能评估
- 模型关键性能指标评估结果
(1)跨模态检索功能:Recall@5%达0.186,比OpenAI CLIP高3.2倍(图4)

图4 使用 Loki、OpenAI CLIP 和 PLIP 对四个验证数据集和一个测试数据集进行图像到转录组学检索评估
(2)细胞分解功能:在结直肠癌全切片中准确预测6种细胞空间分布(图5)。

图5 全玻片人结直肠癌细胞型分解
四、资源可用性
代码:https://github.com/GuangyuWangLab2021/Loki
模型:https://huggingface.co/WangGuangyulab/Loki
数据:ST-bank开放访问,部分临床数据需遵循TCGA/GTEx使用协议。