-
准备优化好的POSCAR文件
-
在POSCAR目录下运行下面代码,得到KPOINTS和INCAR:
bash
vaspkit → 102 → 2 → 0.033.拖入VPKIT.in,注意第一行写1,用于前处理
bash
1 ! 1 for pre-processing; 2 for post-processing
3D ! 2D for slab, 3D for bulk
11 ! number of strain case
-0.010 -0.007 -0.005 -0.003 -0.001 0.000 0.001 0.003 0.005 0.007 0.010 ! Strain range在当前目录下运行:
bash
vaspkit → 200可以看到类似下面的输出: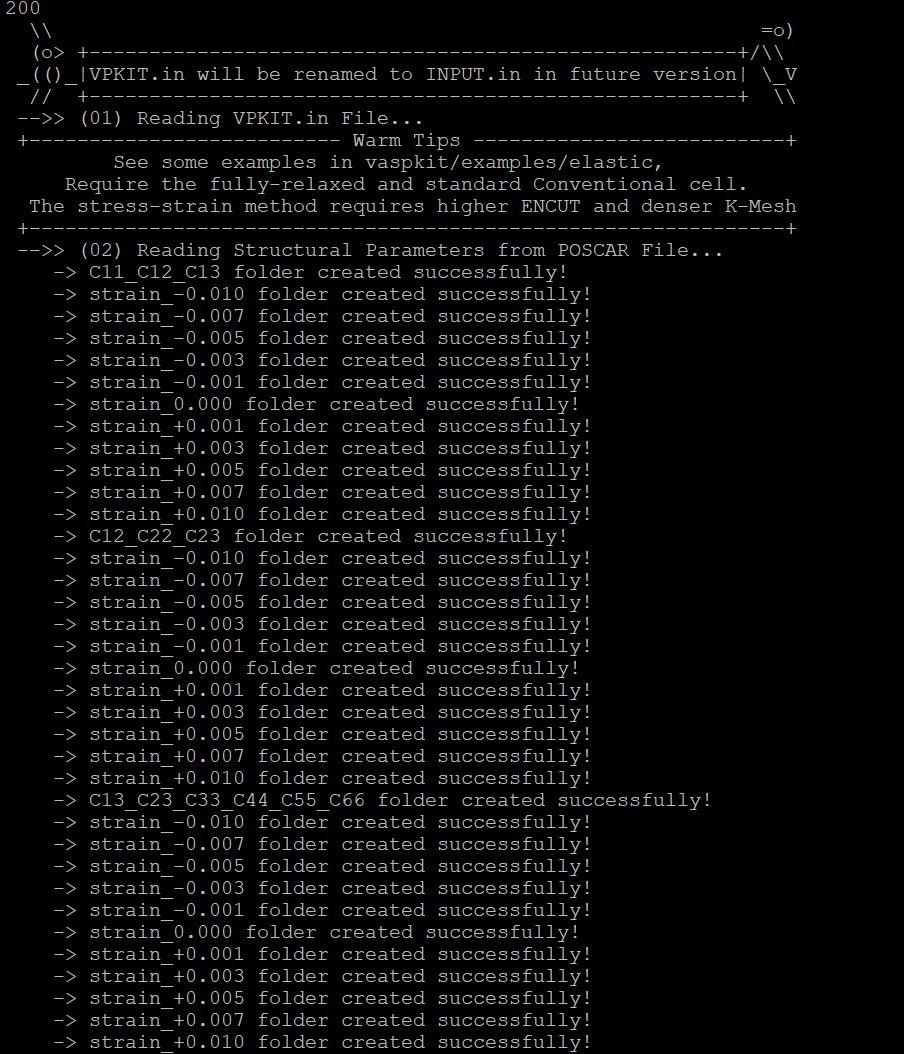
- 生成的文件夹下批量提交vasp作业即可,我的脚本如下:
python
import os
def batch_submit_strain_jobs():
for folder in sorted(os.listdir()):
if folder.startswith("strain_") and os.path.isdir(folder):
print(f"Submitting VASP job in: {folder}")
os.chdir(folder)
os.system("runvasp -q short") # 根据集群需求可改为 vasp_std > out 或 sbatch job.sh
os.chdir("..")
print("✅ All strain jobs submitted.")
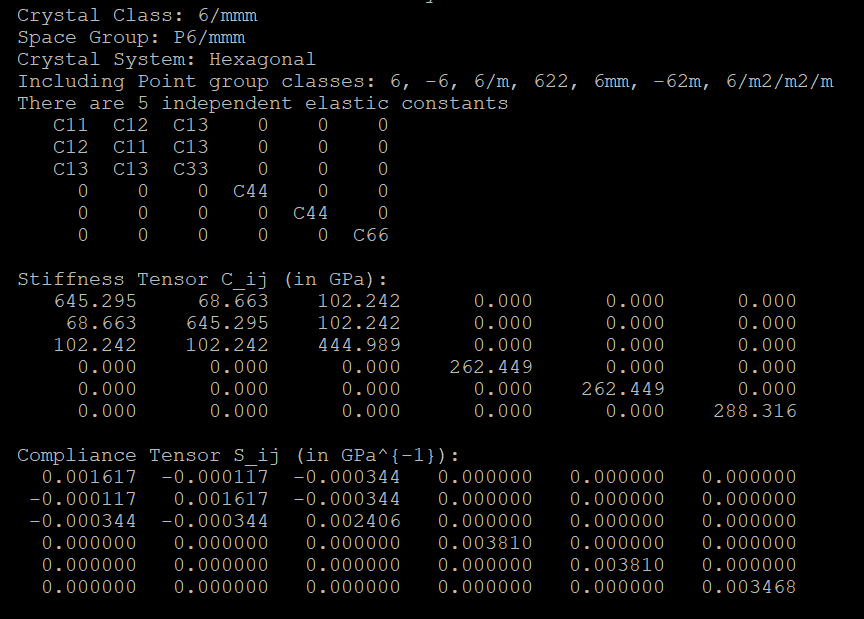
batch_submit_strain_jobs()- 等VASP全部计算完成之后,再次修改VPKIT.in文件中第一行为2 (后处理),然后再次运行VASPKIT并选择200,得到以下结果