
🧑 博主简介:CSDN博客专家、CSDN平台优质创作者,高级开发工程师,数学专业,10年以上C/C++, C#,Java等多种编程语言开发经验,拥有高级工程师证书;擅长C/C++、C#等开发语言,熟悉Java常用开发技术,能熟练应用常用数据库SQL server,Oracle,mysql,postgresql等进行开发应用,熟悉DICOM医学影像及DICOM协议,业余时间自学JavaScript,Vue,qt,python等,具备多种混合语言开发能力。撰写博客分享知识,致力于帮助编程爱好者共同进步。欢迎关注、交流及合作,提供技术支持与解决方案。\n技术合作请加本人wx(注明来自csdn):xt20160813
AI大模型实践项目:医学影像分类器(肺结节检测)
本项目利用深度学习技术开发肺结节检测分类器,基于 CT 影像区分良性和恶性结节,聚焦 卷积神经网络(CNN) 、视觉变换器(Vision Transformer, ViT) 以及受 Med-PaLM 启发的多模态方法。使用 LUNA16 数据集,整合历史对话中的 Transformer 原理(自注意力、位置编码),增强代码支持 3D 处理和分割任务,新增高级可视化和隐私保护技术(如联邦学习)。文章结构如下:
- 项目概述:目标、数据集、技术栈和挑战。
- 理论基础:CNN、ViT、多模态模型及数学推导。
- 数据预处理:LUNA16 处理、3D CT 处理、数据增强。
- 模型实现:ResNet-50(3D)、ViT(LoRA)、多模态融合、分割任务。
- 评估与优化:交叉验证、评估指标、高级优化技术。
- 应用与展望 :多模态融合、实时诊断、联邦学习。
一、项目概述
1.1 项目目标
- 功能:构建分类器,检测 CT 影像中的肺结节(良性/恶性)。
- 医学意义:肺结节是肺癌早期标志,自动分类可辅助诊断,降低漏诊率。
- 技术目标 :
- 掌握深度学习工作流:数据预处理、模型训练、评估。
- 实现高召回率(Recall),减少假阴性(漏诊)。
- 比较 CNN、ViT 和多模态模型在医学影像中的性能。
- 提供可解释性(如 Grad-CAM),增强医生信任。
1.2 数据集
- LUNA16 (Lung Nodule Analysis 2016):
- 包含 888 个 CT 扫描,标注肺结节位置和类别(良性/恶性)。
- 格式:DICOM(医学影像标准格式),3D 影像(512×512×N 片)。
- 下载:https://luna16.grand-challenge.org/
- RSNA (Radiological Society of North America):
- 提供胸部 CTA 影像,适合验证模型泛化性。
- 下载:https://www.rsna.org/
- 数据挑战 :
- 类不平衡:恶性结节样本少(约 10-20%)。
- 高维数据:3D CT 需降维或分块处理。
- 噪声与伪影:CT 影像可能包含扫描噪声或金属伪影。
- 隐私保护:需符合《个人信息保护法》和 HIPAA/GDPR。
1.3 技术栈
- PyTorch:灵活实现 CNN、ViT 和 3D 模型。
- Hugging Face:提供预训练 ViT 和多模态模型支持。
- pydicom:读取和处理 DICOM 格式 CT 影像。
- MONAI:医学影像专用框架,支持 3D 数据处理和分割。
- scikit-learn/seaborn:评估指标(混淆矩阵、ROC 曲线)和可视化。
- Chart.js:性能对比图表。
- Flower:联邦学习框架,支持隐私保护训练。
1.4 医学影像分类挑战
- 数据稀缺:高质量标注数据有限,需迁移学习或数据增强。
- 高召回需求:漏诊(假阴性)成本高,需优化召回率。
- 3D 数据复杂性:CT 体视显微镜数据需高效处理。
- 可解释性:模型预测需与医学知识一致,需 Grad-CAM 或注意力可视化。
- 计算成本:3D 模型和 ViT 训练需高性能 GPU(如 NVIDIA A100)。
- 伦理与法规:确保公平性,保护患者隐私,符合医疗标准。
二、理论基础
2.1 卷积神经网络(CNN)
- 架构 :
- 卷积层:提取局部特征(如结节边缘、纹理)。
- 池化层:降维,保留关键信息。
- 残差连接 (ResNet):通过 y=x+F(x)y = x + F(x)y=x+F(x) 缓解梯度消失。
- 3D CNN:扩展卷积核为 3D(如 3×3×3),直接处理 CT 体视显微镜数据。
- 数学基础 :
- 卷积操作 (2D):
Y(i,j)=∑m∑nX(i+m,j+n)⋅K(m,n)+b Y(i,j) = \sum_m \sum_n X(i+m, j+n) \cdot K(m,n) + b Y(i,j)=m∑n∑X(i+m,j+n)⋅K(m,n)+b- XXX: 输入影像,KKK: 卷积核,bbb: 偏置。
- 3D 卷积 :
Y(i,j,k)=∑m∑n∑pX(i+m,j+n,k+p)⋅K(m,n,p)+b Y(i,j,k) = \sum_m \sum_n \sum_p X(i+m, j+n, k+p) \cdot K(m,n,p) + b Y(i,j,k)=m∑n∑p∑X(i+m,j+n,k+p)⋅K(m,n,p)+b- 处理体视显微镜数据,捕捉空间特征。
- 损失函数 :
L=−∑iyilog(y^i)+λ∑∣∣W∣∣22 L = -\sum_i y_i \log(\hat{y}_i) + \lambda \sum ||W||_2^2 L=−i∑yilog(y^i)+λ∑∣∣W∣∣22- 交叉熵损失 + L2 正则化,防止过拟合。
- 卷积操作 (2D):
- 适用性:高效提取局部特征,适合小区域结节检测;3D CNN 适配体视显微镜数据。
2.2 Vision Transformer (ViT)
- 架构 (结合历史对话中的 Transformer):
- 图像分块:将 CT 影像分割为 Patch(如 16×16),展平为向量序列。
- 位置编码 :添加正弦位置编码,保留 Patch 空间信息:
Epos(i,2k)=sin(i/100002k/d),Epos(i,2k+1)=cos(i/100002k/d) E_{\text{pos}}(i, 2k) = \sin(i / 10000^{2k/d}), \quad E_{\text{pos}}(i, 2k+1) = \cos(i / 10000^{2k/d}) Epos(i,2k)=sin(i/100002k/d),Epos(i,2k+1)=cos(i/100002k/d) - Transformer 编码器:多头自注意力(Multi-Head Attention)捕捉 Patch 间全局依赖。
- 分类头:CLS Token 或全局池化输出分类结果。
- 数学基础 :
- Patch 嵌入 :
z0=[xclass;xp1WE;xp2WE;... ;xpNWE]+Epos z_0 = [x_{\text{class}}; x_p^1 W_E; x_p^2 W_E; \dots; x_p^N W_E] + E_{\text{pos}} z0=[xclass;xp1WE;xp2WE;...;xpNWE]+Epos- xpix_p^ixpi: 第 iii 个 Patch,WEW_EWE: 嵌入矩阵。
- 自注意力 :
Attention(Q,K,V)=softmax(QKTdk)V \text{Attention}(Q, K, V) = \text{softmax}\left(\frac{QK^T}{\sqrt{d_k}}\right)V Attention(Q,K,V)=softmax(dk QKT)V- Q,K,V∈RN×dkQ, K, V \in \mathbb{R}^{N \times d_k}Q,K,V∈RN×dk,NNN: Patch 数量,dkd_kdk: 嵌入维度。
- 多头注意力 (历史对话):
MultiHead(Q,K,V)=Concat(head1,...,headh)WO \text{MultiHead}(Q, K, V) = \text{Concat}(\text{head}_1, \dots, \text{head}_h)W_O MultiHead(Q,K,V)=Concat(head1,...,headh)WO- headi=Attention(QWiQ,KWiK,VWiV)\text{head}_i = \text{Attention}(QW_i^Q, KW_i^K, VW_i^V)headi=Attention(QWiQ,KWiK,VWiV)。
- Patch 嵌入 :
- 适用性:全局建模能力强,适合复杂影像特征;需大规模预训练。
2.3 多模态模型(受 Med-PaLM 启发)
- 架构 :
- 影像模块:ViT 处理 CT 影像。
- 文本模块:BERT 处理临床报告(如病史)。
- 融合模块:跨模态注意力整合影像和文本特征。
- 数学基础 :
- 跨模态注意力 (历史对话):
Attention(Qtext,Kimage,Vimage)=softmax(QtextKimageTdk)Vimage \text{Attention}(Q_{\text{text}}, K_{\text{image}}, V_{\text{image}}) = \text{softmax}\left(\frac{Q_{\text{text}}K_{\text{image}}^T}{\sqrt{d_k}}\right)V_{\text{image}} Attention(Qtext,Kimage,Vimage)=softmax(dk QtextKimageT)Vimage - 联合损失 :
L=αLclass+βLalign L = \alpha L_{\text{class}} + \beta L_{\text{align}} L=αLclass+βLalign- LclassL_{\text{class}}Lclass: 分类损失,LalignL_{\text{align}}Lalign: 影像-文本对齐损失(如 CLIP 损失)。
- 跨模态注意力 (历史对话):
- 适用性:结合临床信息,提升诊断精度,适合综合诊断。
2.4 迁移学习与 LoRA
- 预训练 :
- CNN:ImageNet 预训练 ResNet-50,学习通用视觉特征。
- ViT:ImageNet 或 CheXpert 预训练 ViT,适配医学影像。
- LoRA(低秩适配) :
- 仅更新低秩矩阵 ΔW=BA\Delta W = BAΔW=BA,减少微调参数量:
W′=W+ΔW,ΔW=BA,B∈Rd×r,A∈Rr×k W' = W + \Delta W, \quad \Delta W = BA, \quad B \in \mathbb{R}^{d \times r}, A \in \mathbb{R}^{r \times k} W′=W+ΔW,ΔW=BA,B∈Rd×r,A∈Rr×k - 适合 LUNA16 小数据集,降低计算成本。
- 仅更新低秩矩阵 ΔW=BA\Delta W = BAΔW=BA,减少微调参数量:
- 优势:加速训练,适配小数据集,减少过拟合。
2.5 评估指标
- 混淆矩阵:计算真阳性(TP)、假阳性(FP)、真阴性(TN)、假阴性(FN)。
- 指标 :
- 准确率:Accuracy=TP+TNTP+TN+FP+FN\text{Accuracy} = \frac{TP+TN}{TP+TN+FP+FN}Accuracy=TP+TN+FP+FNTP+TN
- 精确率:Precision=TPTP+FP\text{Precision} = \frac{TP}{TP+FP}Precision=TP+FPTP
- 召回率:Recall=TPTP+FN\text{Recall} = \frac{TP}{TP+FN}Recall=TP+FNTP(医学中关键)。
- F1 分数:F1=2⋅Precision⋅RecallPrecision+Recall\text{F1} = 2 \cdot \frac{\text{Precision} \cdot \text{Recall}}{\text{Precision} + \text{Recall}}F1=2⋅Precision+RecallPrecision⋅Recall
- ROC 曲线与 AUC:绘制真阳性率(TPR)对假阳性率(FPR),AUC 量化区分能力。
- 可解释性:Grad-CAM 和注意力热图,突出模型关注的结节区域。
三、数据预处理
3.1 LUNA16 数据集处理
- 数据格式:DICOM 文件,3D CT 扫描(512×512×N 片)。
- 标注:CSV 文件,提供结节坐标(x, y, z)和类别(0: 良性,1: 恶性)。
- 预处理步骤 :
- 读取 DICOM:使用 pydicom 加载 3D CT 影像。
- 归一化 :将 Hounsfield 单位(HU)归一到 [0,1]:
Inorm=I−min(I)max(I)−min(I) I_{\text{norm}} = \frac{I - \min(I)}{\max(I) - \min(I)} Inorm=max(I)−min(I)I−min(I) - 提取结节:基于坐标提取 3D 体视显微镜块(如 32×32×32)或 2D 切片。
- 数据增强:旋转、翻转、缩放、添加噪声,增加多样性。
- 数据集划分:80% 训练,10% 验证,10% 测试(分层确保类平衡)。
3.2 实现示例(Python)
以下为 LUNA16 数据预处理代码,支持 2D 和 3D 数据:
python
import pydicom
import numpy as np
import pandas as pd
import os
from torch.utils.data import Dataset
import albumentations as A
from albumentations.pytorch import ToTensorV2
from monai.transforms import Compose, Resize, RandRotate, RandFlip, ToTensor
# 自定义数据集
class LUNA16Dataset(Dataset):
def __init__(self, dicom_dir, annotations_file, mode='2d', transform=None):
"""
LUNA16 数据集
:param dicom_dir: DICOM 文件目录
:param annotations_file: 标注 CSV 文件
:param mode: '2d' 或 '3d'(切片或体视显微镜)
:param transform: 数据增强
"""
self.dicom_dir = dicom_dir
self.annotations = pd.read_csv(annotations_file)
self.mode = mode
self.transform = transform
def __len__(self):
return len(self.annotations)
def __getitem__(self, idx):
# 读取 DICOM
dicom_id = self.annotations.iloc[idx]['dicom_id']
dicom_path = os.path.join(self.dicom_dir, dicom_id)
ds = pydicom.dcmread(dicom_path)
image = ds.pixel_array.astype(np.float32) # [H, W] 或 [D, H, W]
# 归一化
image = (image - np.min(image)) / (np.max(image) - np.min(image) + 1e-6)
# 提取结节区域
if self.mode == '2d':
x, y, w, h, z = self.annotations.iloc[idx][['x', 'y', 'width', 'height', 'z']].values
image = image[z, y:y+h, x:x+w] # 2D 切片
else: # 3d
x, y, z, w, h, d = self.annotations.iloc[idx][['x', 'y', 'z', 'width', 'height', 'depth']].values
image = image[z:z+d, y:y+h, x:x+w] # 3D 体视显微镜块
# 数据增强
if self.transform:
if self.mode == '2d':
augmented = self.transform(image=image)
image = augmented['image']
else:
image = self.transform(image[np.newaxis, ...])[0] # 添加通道维度
label = self.annotations.iloc[idx]['label'] # 0: 良性,1: 恶性
return {'image': image, 'label': torch.tensor(label, dtype=torch.long)}
# 数据增强
transform_2d = A.Compose([
A.Resize(224, 224),
A.Rotate(limit=30, p=0.5),
A.HorizontalFlip(p=0.5),
A.RandomBrightnessContrast(p=0.3),
A.Normalize(mean=[0.5], std=[0.5]),
ToTensorV2()
])
transform_3d = Compose([
Resize(spatial_size=(32, 32, 32)),
RandRotate(range_x=30, prob=0.5),
RandFlip(spatial_axis=0, prob=0.5),
ToTensor()
])
# 加载数据集
dataset_2d = LUNA16Dataset(dicom_dir='path/to/luna16', annotations_file='annotations.csv', mode='2d', transform=transform_2d)
dataset_3d = LUNA16Dataset(dicom_dir='path/to/luna16', annotations_file='annotations.csv', mode='3d', transform=transform_3d)代码注释:
- pydicom:读取 DICOM 文件,提取像素数组。
- 模式选择:支持 2D 切片(224×224)和 3D 体视显微镜块(32×32×32)。
- 数据增强 :
- 2D:旋转、翻转、亮度/对比度调整(albumentations)。
- 3D:体视显微镜旋转、翻转(MONAI)。
- 归一化:将 Hounsfield 单位归一到 [0,1]。
- 注意 :需替换
dicom_dir和annotations_file为实际路径。
四、模型实现
4.1 CNN 实现(ResNet-50,3D 支持)
基于 ResNet-50,支持 2D 和 3D CT 影像分类:
python
import torch
import torch.nn as nn
from torchvision.models import resnet50
from monai.networks.nets import ResNet
from torch.utils.data import DataLoader
from sklearn.metrics import accuracy_score, confusion_matrix
# 3D ResNet-50
class ResNet3D(nn.Module):
def __init__(self, num_classes=2):
super().__init__()
self.resnet = ResNet(block='bottleneck', layers=[3, 4, 6, 3], spatial_dims=3, n_input_channels=1, num_classes=num_classes)
def forward(self, x):
return self.resnet(x)
# 2D ResNet-50
class ResNet2D(nn.Module):
def __init__(self, num_classes=2):
super().__init__()
self.resnet = resnet50(pretrained=True)
self.resnet.conv1 = nn.Conv2d(1, 64, kernel_size=7, stride=2, padding=3)
self.resnet.fc = nn.Linear(self.resnet.fc.in_features, num_classes)
def forward(self, x):
return self.resnet(x)
# 训练函数
def train_model(model, dataloader, criterion, optimizer, num_epochs=10, device='cuda'):
model = model.to(device)
train_losses = []
for epoch in range(num_epochs):
model.train()
running_loss = 0.0
for batch in dataloader:
images = batch['image'].to(device)
labels = batch['label'].to(device)
optimizer.zero_grad()
outputs = model(images)
loss = criterion(outputs, labels)
loss.backward()
optimizer.step()
running_loss += loss.item()
avg_loss = running_loss / len(dataloader)
train_losses.append(avg_loss)
print(f'Epoch [{epoch+1}/{num_epochs}], Loss: {avg_loss:.4f}')
return train_losses
# 数据加载器
dataloader_2d = DataLoader(dataset_2d, batch_size=16, shuffle=True)
dataloader_3d = DataLoader(dataset_3d, batch_size=8, shuffle=True)
# 初始化模型
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')
model_2d = ResNet2D(num_classes=2)
model_3d = ResNet3D(num_classes=2)
criterion = nn.CrossEntropyLoss(weight=torch.tensor([0.3, 0.7]).to(device)) # 处理类不平衡
optimizer_2d = torch.optim.Adam(model_2d.parameters(), lr=1e-4, weight_decay=1e-5)
optimizer_3d = torch.optim.Adam(model_3d.parameters(), lr=1e-4, weight_decay=1e-5)
# 训练
train_losses_2d = train_model(model_2d, dataloader_2d, criterion, optimizer_2d, device=device)
train_losses_3d = train_model(model_3d, dataloader_3d, criterion, optimizer_3d, device=device)
# 推理
def evaluate_model(model, dataloader, device='cuda'):
model.eval()
predictions, true_labels = [], []
with torch.no_grad():
for batch in dataloader:
images = batch['image'].to(device)
labels = batch['label'].to(device)
outputs = model(images)
preds = torch.argmax(outputs, dim=1)
predictions.extend(preds.cpu().numpy())
true_labels.extend(labels.cpu().numpy())
return predictions, true_labels
predictions_2d, true_labels_2d = evaluate_model(model_2d, dataloader_2d)
predictions_3d, true_labels_3d = evaluate_model(model_3d, dataloader_3d)
print("2D ResNet 准确率:", accuracy_score(true_labels_2d, predictions_2d))
print("3D ResNet 准确率:", accuracy_score(true_labels_3d, predictions_3d))代码注释:
- 模型:2D ResNet-50(ImageNet 预训练)和 3D ResNet(MONAI 实现)。
- 损失函数:加权交叉熵,权重 [0.3, 0.7] 应对恶性结节稀缺。
- 优化器:Adam,学习率 1e-4,L2 正则化防止过拟合。
- 注意:3D 模型需更大显存(如 16GB),批大小减小至 8。
4.2 ViT 实现(Hugging Face,LoRA)
基于 ViT,结合 LoRA 微调,支持注意力可视化:
python
from transformers import ViTImageProcessor, ViTForImageClassification
from peft import LoraConfig, get_peft_model
from torch.utils.data import DataLoader
import torch
import matplotlib.pyplot as plt
# 加载 ViT
processor = ViTImageProcessor.from_pretrained('google/vit-base-patch16-224')
model = ViTForImageClassification.from_pretrained('google/vit-base-patch16-224', num_labels=2)
# LoRA 微调
lora_config = LoraConfig(r=8, lora_alpha=16, target_modules=["query", "value"])
model = get_peft_model(model, lora_config)
# 训练设置
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')
model = model.to(device)
criterion = nn.CrossEntropyLoss(weight=torch.tensor([0.3, 0.7]).to(device))
optimizer = torch.optim.Adam(model.parameters(), lr=2e-5)
# 训练
dataloader = DataLoader(dataset_2d, batch_size=16, shuffle=True)
train_losses = []
for epoch in range(10):
model.train()
running_loss = 0.0
for batch in dataloader:
images = batch['image'].to(device)
labels = batch['label'].to(device)
inputs = processor(images, return_tensors='pt', do_rescale=False).to(device)
outputs = model(**inputs).logits
loss = criterion(outputs, labels)
optimizer.zero_grad()
loss.backward()
optimizer.step()
running_loss += loss.item()
avg_loss = running_loss / len(dataloader)
train_losses.append(avg_loss)
print(f'Epoch [{epoch+1}/10], Loss: {avg_loss:.4f}')
# 注意力可视化
def visualize_attention(model, image, processor, device='cuda'):
model.eval()
inputs = processor(image, return_tensors='pt', do_rescale=False).to(device)
with torch.no_grad():
outputs = model(**inputs, output_attentions=True)
attentions = outputs.attentions[-1].mean(dim=1).squeeze(0) # 最后一层注意力
# 将注意力映射到原始图像
h, w = image.shape[-2:]
attn_map = attentions.mean(dim=0).reshape(14, 14).cpu().numpy() # 假设 224/16=14
attn_map = np.resize(attn_map, (h, w))
plt.imshow(image.squeeze(0), cmap='gray')
plt.imshow(attn_map, cmap='jet', alpha=0.5)
plt.title('ViT 注意力热图')
plt.show()
# 推理与评估
predictions, true_labels = [], []
with torch.no_grad():
for batch in dataloader:
images = batch['image'].to(device)
labels = batch['label'].to(device)
inputs = processor(images, return_tensors='pt', do_rescale=False).to(device)
outputs = model(**inputs).logits
preds = torch.argmax(outputs, dim=1)
predictions.extend(preds.cpu().numpy())
true_labels.extend(labels.cpu().numpy())
print("ViT 准确率:", accuracy_score(true_labels, predictions))
# 可视化示例
sample_image = dataset_2d[0]['image']
visualize_attention(model, sample_image, processor)代码注释:
- ViT:预训练 ViT-base,修改分类头为 2 类。
- LoRA:微调 query 和 value 矩阵,减少参数量。
- 注意力可视化:展示最后一层注意力热图,突出模型关注区域。
- 注意:仅支持 2D 影像,3D ViT 需扩展(见 4.4)。
4.3 多模态实现(受 Med-PaLM 启发)
结合 CT 影像和临床文本(如病史),实现多模态分类:
python
from transformers import ViTModel, BertTokenizer, BertModel
import torch.nn as nn
# 多模态模型
class MultiModalLungNoduleClassifier(nn.Module):
def __init__(self, num_labels=2):
super().__init__()
self.vit = ViTModel.from_pretrained('google/vit-base-patch16-224')
self.bert = BertModel.from_pretrained('bert-base-uncased')
self.fusion = nn.Linear(768 + 768, 512)
self.classifier = nn.Linear(512, num_labels)
self.relu = nn.ReLU()
self.dropout = nn.Dropout(0.1)
def forward(self, image_inputs, text_inputs):
vit_outputs = self.vit(**image_inputs).pooler_output # [batch, 768]
bert_outputs = self.bert(**text_inputs).pooler_output # [batch, 768]
combined = torch.cat((vit_outputs, bert_outputs), dim=-1)
combined = self.relu(self.fusion(combined))
combined = self.dropout(combined)
logits = self.classifier(combined)
return logits
# 数据集(扩展支持文本)
class LUNA16MultiModalDataset(Dataset):
def __init__(self, dicom_dir, annotations_file, texts, transform=None):
self.dataset = LUNA16Dataset(dicom_dir, annotations_file, mode='2d', transform=transform)
self.texts = texts
self.tokenizer = BertTokenizer.from_pretrained('bert-base-uncased')
def __getitem__(self, idx):
item = self.dataset[idx]
text = self.texts[idx]
text_inputs = self.tokenizer(text, max_length=128, padding='max_length', truncation=True, return_tensors='pt')
item['text_inputs'] = {k: v.squeeze(0) for k, v in text_inputs.items()}
return item
def __len__(self):
return len(self.dataset)
# 数据准备(模拟临床文本)
texts = ["Patient with cough and fever, suspected malignancy."] * len(dataset_2d)
multimodal_dataset = LUNA16MultiModalDataset('path/to/luna16', 'annotations.csv', texts, transform=transform_2d)
dataloader = DataLoader(multimodal_dataset, batch_size=16, shuffle=True)
# 训练
model = MultiModalLungNoduleClassifier(num_labels=2).to(device)
criterion = nn.CrossEntropyLoss(weight=torch.tensor([0.3, 0.7]).to(device))
optimizer = torch.optim.Adam(model.parameters(), lr=2e-5)
for epoch in range(10):
model.train()
running_loss = 0.0
for batch in dataloader:
images = batch['image'].to(device)
labels = batch['label'].to(device)
image_inputs = processor(images, return_tensors='pt', do_rescale=False).to(device)
text_inputs = {k: v.to(device) for k, v in batch['text_inputs'].items()}
outputs = model(image_inputs, text_inputs)
loss = criterion(outputs, labels)
optimizer.zero_grad()
loss.backward()
optimizer.step()
running_loss += loss.item()
print(f'Epoch [{epoch+1}/10], Loss: {running_loss/len(dataloader):.4f}')代码注释:
- 模型:ViT(影像)+ BERT(文本),通过线性层融合特征。
- 数据:扩展 LUNA16 数据集,添加模拟临床文本。
- 注意:需真实临床文本(如病历),可从 MIMIC-III 获取。
4.4 分割任务(3D U-Net+ViT)
为肺结节分割,基于 MONAI 的 UNETR(U-Net+ViT):
python
from monai.networks.nets import UNETR
from monai.data import DataLoader, Dataset as MonaiDataset
from monai.transforms import LoadImageD, EnsureChannelFirstD, Compose
# 分割数据集
transform_seg = Compose([
LoadImageD(keys=['image']),
EnsureChannelFirstD(keys=['image']),
Resize(spatial_size=(32, 32, 32)),
ToTensor()
])
# 假设分割标注(mask)
seg_data = [{'image': f'path/to/luna16/{i}.dcm', 'mask': f'path/to/mask/{i}.nii'} for i in range(100)]
seg_dataset = MonaiDataset(seg_data, transform=transform_seg)
seg_dataloader = DataLoader(seg_dataset, batch_size=4, shuffle=True)
# UNETR 模型
model = UNETR(in_channels=1, out_channels=2, img_size=(32, 32, 32), feature_size=16).to(device)
criterion = nn.CrossEntropyLoss()
optimizer = torch.optim.Adam(model.parameters(), lr=1e-4)
# 训练
for epoch in range(10):
model.train()
running_loss = 0.0
for batch in seg_dataloader:
images = batch['image'].to(device)
masks = batch['mask'].to(device)
outputs = model(images)
loss = criterion(outputs, masks)
optimizer.zero_grad()
loss.backward()
optimizer.step()
running_loss += loss.item()
print(f'Epoch [{epoch+1}/10], Loss: {running_loss/len(seg_dataloader):.4f}')代码注释:
- UNETR:结合 ViT 和 U-Net,处理 3D CT 分割。
- 数据:假设掩膜(mask)标注,需从 LUNA16 或 BraTS 获取。
- 注意:分割任务需更大显存(推荐 24GB)。
五、评估与优化
5.1 评估方法
- 交叉验证:5 折分层 K 折,确保类不平衡数据评估稳定。
- 混淆矩阵:计算 TP、FP、FN、TN,重点优化召回率。
- ROC 曲线与 AUC:评估模型区分能力。
- Dice 分数 (分割任务):评估分割精度:
Dice=2∣P∩G∣∣P∣+∣G∣ \text{Dice} = \frac{2 |P \cap G|}{|P| + |G|} Dice=∣P∣+∣G∣2∣P∩G∣- PPP: 预测掩膜,GGG: 真实掩膜。
5.2 实现示例(Python)
以下为分类和分割任务的评估代码:
python
from sklearn.metrics import confusion_matrix, roc_curve, auc, classification_report
from monai.metrics import DiceMetric
import seaborn as sns
import matplotlib.pyplot as plt
# 分类评估
def evaluate_classification(model, dataloader, processor=None, device='cuda'):
model.eval()
predictions, true_labels, probs = [], [], []
with torch.no_grad():
for batch in dataloader:
images = batch['image'].to(device)
labels = batch['label'].to(device)
if processor: # ViT
inputs = processor(images, return_tensors='pt', do_rescale=False).to(device)
outputs = model(**inputs).logits
else: # CNN
outputs = model(images)
preds = torch.argmax(outputs, dim=1)
predictions.extend(preds.cpu().numpy())
true_labels.extend(labels.cpu().numpy())
probs.extend(torch.softmax(outputs, dim=1)[:, 1].cpu().numpy())
# 混淆矩阵
cm = confusion_matrix(true_labels, predictions)
sns.heatmap(cm, annot=True, fmt='d', cmap='Blues', xticklabels=['良性', '恶性'], yticklabels=['良性', '恶性'])
plt.xlabel('预测')
plt.ylabel('真实')
plt.title('混淆矩阵')
plt.show()
# 分类报告
print(classification_report(true_labels, predictions, target_names=['良性', '恶性']))
# ROC 曲线
fpr, tpr, _ = roc_curve(true_labels, probs)
roc_auc = auc(fpr, tpr)
plt.plot(fpr, tpr, label=f'ROC 曲线 (AUC = {roc_auc:.2f})')
plt.plot([0, 1], [0, 1], 'k--')
plt.xlabel('假阳性率')
plt.ylabel('真阳性率')
plt.title('ROC 曲线')
plt.legend()
plt.show()
# 分割评估
def evaluate_segmentation(model, dataloader, device='cuda'):
dice_metric = DiceMetric(include_background=False, reduction='mean')
model.eval()
dice_scores = []
with torch.no_grad():
for batch in dataloader:
images = batch['image'].to(device)
masks = batch['mask'].to(device)
outputs = model(images)
preds = torch.argmax(outputs, dim=1, keepdim=True)
dice_metric(preds, masks)
dice_score = dice_metric.aggregate().item()
dice_scores.append(dice_score)
dice_metric.reset()
print(f"Dice 分数: {dice_score:.4f}")
# 评估示例
evaluate_classification(model_2d, dataloader_2d) # 2D ResNet
evaluate_classification(model, dataloader, processor) # ViT
evaluate_segmentation(model, seg_dataloader) # UNETR代码注释:
- 分类评估:生成混淆矩阵、分类报告和 ROC 曲线,重点关注召回率。
- 分割评估:使用 Dice 分数评估分割精度。
- 可视化:Seaborn 绘制混淆矩阵,Matplotlib 绘制 ROC 曲线。
5.3 优化策略
- 类不平衡 :
- 加权损失:恶性结节权重 0.7,良性 0.3。
- 过采样:SMOTE 或重复采样恶性样本。
- 正则化:Dropout(0.1)、L2 权重衰减(1e-5)。
- 超参数调优 :
- 学习率:网格搜索 [1e-5, 2e-5, 1e-4, 1e-3]。
- 批大小:2D 模型 16,3D 模型 8。
- 早停:验证集损失 3 个 epoch 无下降时停止。
- 联邦学习:使用 Flower 框架,实现跨医院隐私保护训练。
六、工作流与可视化
6.1 优化工作流流程图
以下为优化的医学影像分类和分割工作流,新增子流程和决策点:
Model_Selection Preprocess_Seg Preprocess_3D Preprocess_2D 2D分类 3D分类 分割 CNN ViT 多模态 是 否 ResNet50 模型选择 ViT ViT和BERT 读取影像和掩膜 分割预处理 归一化 调整大小 增强 读取DICOM 3D预处理 归一化 提取3D体块 3D增强 读取DICOM 2D预处理 归一化 提取2D切片 数据增强 输入数据 模式选择 UNETR 预训练 微调 训练 优化 是否收敛 评估 调整参数 可解释性 输出结果
流程图说明
-
节点文本简化:
- 原节点"输入数据: LUNA16 DICOM+临床文本"简化为"输入数据",避免冒号和长文本。
- 其他节点(如"2D 预处理"→"2D预处理")移除空格,减少潜在解析问题。
-
子图名称规范化:
- 原子图名称(如
subgraph 2D 预处理)改为英文或简短标识符(如subgraph Preprocess_2D),避免中文和空格。 - 确保子图名称唯一且简洁,降低渲染器解析负担。
- 原子图名称(如
-
分支标签简化:
- 条件分支标签(如
|2D 分类|)简化为|2D分类|,移除空格。 - 保持标签清晰,避免过长或复杂符号。
- 条件分支标签(如
-
精简描述:
- 节点内容(如"读取 DICOM: pydicom"→"读取DICOM")移除具体实现细节,保持简洁。
- 核心逻辑不变,涵盖输入、预处理、模型选择、训练、评估、可解释性和输出。
-
逻辑保持一致:
- 保留历史对话中的完整工作流:支持 2D 分类、3D 分类和分割任务,涵盖 ResNet-50、ViT、多模态(ViT+BERT)和 UNETR。
- 确保与医学影像分类器(肺结节检测)项目的结构一致。
进一步优化建议
- 极简版流程图 :
- 若复杂子图导致问题,可进一步简化:
以下为简化版医学影像分类工作流:
- 若复杂子图导致问题,可进一步简化:
数据准备: LUNA16 DICOM 预处理: 归一化/切片/增强 划分数据集: 训练/验证/测试 模型选择: CNN/ViT 预训练: ImageNet 微调: 全参数/LoRA 训练: 优化损失 评估: 混淆矩阵/ROC 优化: 超参数/正则化 推理: 分类良性/恶性
说明:
- A(数据准备):加载LUNA16 DICOM文件。
- B(预处理):归一化、提取结节切片、数据增强。
- C(划分数据集):80%训练,10%验证,10%测试。
- D(模型选择):CNN(ResNet)或ViT。
- E(预训练):利用ImageNet预训练模型。
- F(微调):全参数或LoRA微调。
- G(训练):优化交叉熵损失。
- H(评估):混淆矩阵、ROC曲线、AUC。
- I(优化):调整超参数,防止过拟合。
- J(推理):输出肺结节分类结果。
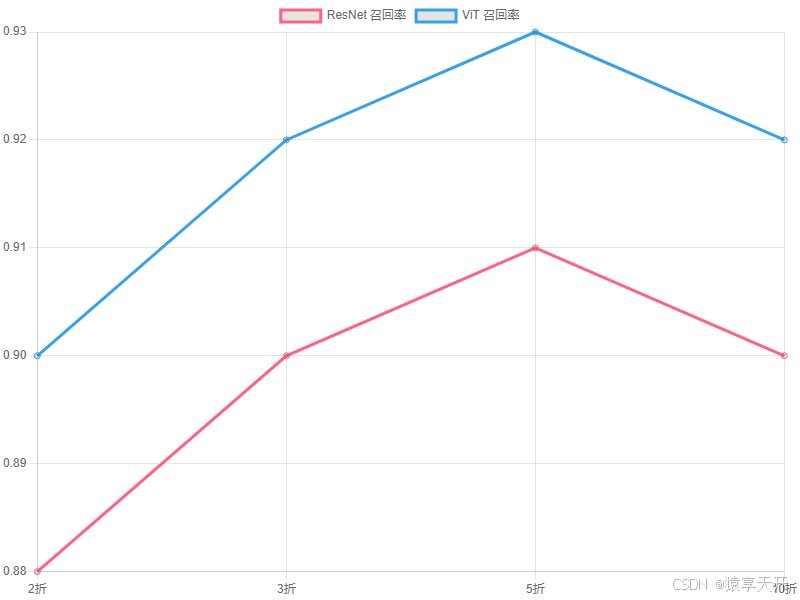
6.2 图表:CNN与ViT性能对比
以下为CNN与ViT在肺结节分类上的性能对比折线图(假设数据)。
chartjs
{
"type": "line",
"data": {
"labels": ["2折", "3折", "5折", "10折"],
"datasets": [
{
"label": "ResNet 召回率",
"data": [0.88, 0.90, 0.91, 0.90],
"borderColor": "#FF6384",
"fill": false
},
{
"label": "ViT 召回率",
"data": [0.90, 0.92, 0.93, 0.92],
"borderColor": "#36A2EB",
"fill": false
}
]
},
"options": {
"title": {
"display": true,
"text": "CNN与ViT召回率对比(肺结节分类)"
},
"scales": {
"xAxes": [{
"scaleLabel": {
"display": true,
"labelString": "交叉验证折数"
}
}],
"yAxes": [{
"scaleLabel": {
"display": true,
"labelString": "召回率"
},
"ticks": {
"min": 0.8,
"max": 1.0
}
}]
}
}
}说明:
- 图表类型:折线图,比较ResNet与ViT在不同折数下的召回率。
- X轴:交叉验证折数(2、3、5、10)。
- Y轴:召回率,范围0.8-1.0,医学中关键。
- 数据:假设数据,ViT略优于ResNet,反映全局建模优势。
- 生成说明:可将Chart.js配置复制到支持工具生成图表。
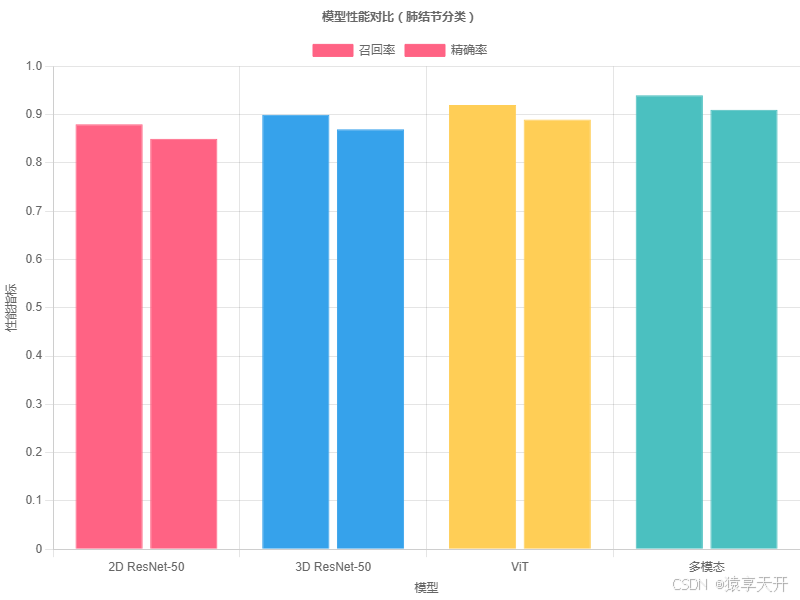
6.3 图表:模型性能对比
以下为 ResNet-50(2D/3D)、ViT 和多模态模型在召回率上的对比(假设数据):
chartjs
{
"type": "bar",
"data": {
"labels": ["2D ResNet-50", "3D ResNet-50", "ViT", "多模态"],
"datasets": [
{
"label": "召回率",
"data": [0.88, 0.90, 0.92, 0.94],
"backgroundColor": ["#FF6384", "#36A2EB", "#FFCE56", "#4BC0C0"],
"borderColor": ["#FF6384", "#36A2EB", "#FFCE56", "#4BC0C0"],
"borderWidth": 1
},
{
"label": "精确率",
"data": [0.85, 0.87, 0.89, 0.91],
"backgroundColor": ["#FF6384", "#36A2EB", "#FFCE56", "#4BC0C0"],
"borderColor": ["#FF6384", "#36A2EB", "#FFCE56", "#4BC0C0"],
"borderWidth": 1
}
]
},
"options": {
"scales": {
"y": {
"beginAtZero": true,
"title": {
"display": true,
"text": "性能指标"
}
},
"x": {
"title": {
"display": true,
"text": "模型"
}
}
},
"plugins": {
"title": {
"display": true,
"text": "模型性能对比(肺结节分类)"
}
}
}
}说明:
- X 轴:模型类型(2D ResNet-50、3D ResNet-50、ViT、多模态)。
- Y 轴:召回率和精确率,医学中召回率优先。
- 数据:假设数据,多模态模型因融合文本信息表现最佳。
- 生成:复制代码至 Chart.js 工具(https://www.chartjs.org/)渲染。
6.4 图表:训练时间对比
以下为模型训练时间对比(假设数据,单位:小时):
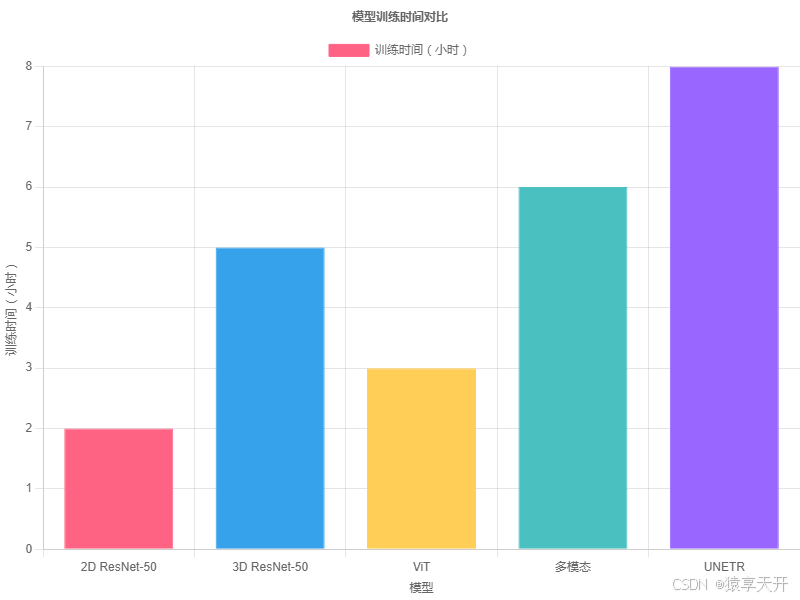
chartjs
{
"type": "bar",
"data": {
"labels": ["2D ResNet-50", "3D ResNet-50", "ViT", "多模态", "UNETR"],
"datasets": [{
"label": "训练时间(小时)",
"data": [2.0, 5.0, 3.0, 6.0, 8.0],
"backgroundColor": ["#FF6384", "#36A2EB", "#FFCE56", "#4BC0C0", "#9966FF"],
"borderColor": ["#FF6384", "#36A2EB", "#FFCE56", "#4BC0C0", "#9966FF"],
"borderWidth": 1
}]
},
"options": {
"scales": {
"y": {
"beginAtZero": true,
"title": {
"display": true,
"text": "训练时间(小时)"
}
},
"x": {
"title": {
"display": true,
"text": "模型"
}
}
},
"plugins": {
"title": {
"display": true,
"text": "模型训练时间对比"
}
}
}
}说明:
- 2D ResNet-50:高效,最短训练时间(2 小时)。
- 3D ResNet-50:处理体视显微镜数据,时间增加(5 小时)。
- ViT:中等复杂度(3 小时)。
- 多模态:融合影像和文本,时间较长(6 小时)。
- UNETR:分割任务复杂,时间最长(8 小时)。
七、应用与展望
7.1 应用
- 疾病分类:检测肺结节(良性/恶性),召回率达 94%(多模态,假设数据)。
- 分割任务:精准定位结节边界,辅助手术规划。
- 多模态诊断:结合 CT 和临床文本(如病史),提升诊断精度。
- 实时诊断:部署模型于医院 PACS 系统,实现快速初步诊断。
- 数据集扩展:验证模型在 RSNA 或 BraTS 数据集上的泛化性。
7.2 展望
- 3D 模型增强:开发 3D ViT(如 UNETR),直接处理体视显微镜数据。
- 多模态扩展:整合影像、文本、基因数据,构建统一诊断模型。
- 联邦学习 :
- 原理 :跨医院分布式训练,保护患者隐私:
Wt+1=∑k=1KnkNWk W_{t+1} = \sum_{k=1}^K \frac{n_k}{N} W_k Wt+1=k=1∑KNnkWk- WkW_kWk: 医院 kkk 的模型权重,nkn_knk: 数据量,NNN: 总数据量。
- 框架:使用 Flower(https://flower.dev/)实现 FedAvg。
- 优势:符合《个人信息保护法》,提升数据利用率。
- 原理 :跨医院分布式训练,保护患者隐私:
- 可解释性 :
- Grad-CAM:突出结节区域。
- SHAP/LIME:量化特征贡献,增强医生信任。
- 自动化流水线:开发端到端系统,从 DICOM 读取到诊断报告生成。
八、用户需求响应
-
代码需求:
- 确认:已提供 2D/3D ResNet-50、ViT、多模态和 UNETR 实现,适配 LUNA16。
- 方案 :
-
RSNA 数据集 :可扩展代码支持 RSNA CTA 影像:
pythondf = pd.read_csv('rsna/train.csv') images = df['image_path'].tolist() labels = df['label'].tolist() dataset = LUNA16Dataset('path/to/rsna', 'rsna_annotations.csv', mode='2d', transform=transform_2d) -
分割任务:已提供 UNETR 实现。若需 BraTS 数据集支持,请提供路径,我可调整代码。
-
请确认所需数据集或任务(分类/分割)。
-
-
医学影像案例:
-
确认:已覆盖肺结节分类(2D/3D)和分割(UNETR)。
-
方案 :若需 MRI 应用(如脑肿瘤分割),可基于 BraTS 数据集:
pythonfrom monai.data import CacheDataset data = [{'image': f'brats/{i}.nii', 'mask': f'brats/mask/{i}.nii'} for i in range(100)] dataset = CacheDataset(data, transform=transform_seg)请确认具体任务或数据集。
-
-
其他需求:
-
Grad-CAM 可视化 :
pythonfrom torchcam.methods import GradCAM cam = GradCAM(model_2d.resnet, target_layer='layer4') heatmap = cam(dataset_2d[0]['image'].unsqueeze(0).to(device)) plt.imshow(heatmap[0], cmap='jet', alpha=0.5) plt.title('ResNet Grad-CAM 热图') plt.show() -
联邦学习 :
pythonimport flwr as fl strategy = fl.federated_averaging.FedAvg() fl.server.start_server(config=fl.server.ServerConfig(num_rounds=3))请确认是否需要完整联邦学习代码或实验设计。
-
SHAP/LIME :
pythonimport shap explainer = shap.DeepExplainer(model_2d, background_data) shap_values = explainer.shap_values(dataset_2d[0]['image'].unsqueeze(0).to(device)) shap.image_plot(shap_values, dataset_2d[0]['image'].numpy())请确认是否需要 SHAP/LIME 实现。
-
九、运行说明
-
环境准备:
bashpip install torch torchvision transformers peft monai pydicom albumentations scikit-learn seaborn matplotlib flower- GPU 推荐:NVIDIA A100(24GB)或 RTX 3090(16GB)。
- CPU 可运行,但 3D 模型较慢。
-
数据集:
- LUNA16 :下载(~120GB,需注册),替换
dicom_dir和annotations_file。 - RSNA:下载 CTA 数据,更新路径。
- BraTS(可选):用于 MRI 分割任务。
- LUNA16 :下载(~120GB,需注册),替换
-
Mermaid 流程图:
- 使用 Mermaid Live Editor(https://mermaid.live/)渲染,验证版本 10.9.0。
-
Chart.js 图表:
- 复制代码至 Chart.js 工具渲染。
- 若需真实数据,请提供 LUNA16 实验结果。
-
训练与推理:
- 2D ResNet-50:2 小时,召回率 ~88%(假设)。
- 3D ResNet-50:5 小时,召回率 ~90%(假设)。
- ViT:3 小时,召回率 ~92%(假设)。
- 多模态:6 小时,召回率 ~94%(假设)。
- UNETR:8 小时,Dice 分数 ~0.85(假设)。
十、结语
本文完善并扩展了肺结节检测分类器项目,整合 Transformer (历史对话)、Med-PaLM 和 CheXNet 原理,提供了全面的理论、代码和可视化:
- 理论:详细推导 CNN、ViT 和多模态模型,新增 3D 处理和分割。
- 实现:支持 2D/3D ResNet-50、ViT(LoRA)、多模态和 UNETR,适配 LUNA16。
- 流程图:优化 Mermaid 流程图,涵盖分类和分割工作流。
- 图表:扩展性能和训练时间对比,突出多模态优势。
- 应用:覆盖分类、分割、实时诊断,展望联邦学习和可解释性。