几个月前有个3D开发需求,展示化学分子结构,需求学科专业化程度较高了!
对方推荐了JSmol,但是这个跟Jmol(Java分子结构库)紧密结合。最终查找资料找到纯前端解决方案,决定用3Dmol.js实现。本文简单记录一下使用方法,免得忘了!
1.什么是3Dmol.js
 官方介绍:
官方介绍:
3Dmol.js是一个基于WebGL的面向对象的JavaScript库,用于在线分子可视化 - 无需Java! 使用3Dmol.js,您可以将精美渲染的分子可视化添加到您的Web应用程序中。
特性
- 支持pdb、sdf、mol2、xyz和cube格式
- 并行分子表面计算
- 球体、棒状、线条、十字、卡通和曲面样式
- 基于原子属性的选择和样式设置
- 标签
- 与分子数据的可点击交互
- 几何形状,包括球体和箭头
Github地址 https://github.com/3dmol/3Dmol.js
官网地址 https://3dmol.org/
2.使用3Dmol.js
2.1 安装
sh
pnpm add 3dmol该库基于typescript开发,友好支持typescript!
或者使用script形式引入
html
<script src="https://3Dmol.org/build/3Dmol-min.js"></script>
<script src="https://3Dmol.org/build/3Dmol.js"></script>
<script src="https://cdnjs.cloudflare.com/ajax/libs/3Dmol/2.0.1/3Dmol-min.js"></script>
<script src="https://cdnjs.cloudflare.com/ajax/libs/3Dmol/2.0.1/3Dmol.js"></script>2.1 创建场景
html
<div id="container" style="height:800px;width:800px"></div>
ts
import * as $3Dmol from '3dmol/build/3Dmol.js';
const container=document.getElementById("container");
//创建视图
const viewer = createViewer(container, {backgroundColor: "orange"});
//添加绿色球体
viewer.addSphere({center: {x: 0, y: 0, z: 0}, radius: 10.0, color: "green"});
//缩放适配渲染内容大小
viewer.zoomTo();
//开始渲染
viewer.render();
//缩放动画 2秒缩放到0.8视角
viewer.zoom(0.8, 2000);效果如下

常用的几个配置
-
backgroundColor背景颜色 -
antialias是否开启抗锯齿 -
backgroundAlpha背景透明度,范围[0~1] -
defaultcolors3D分子模型默认颜色,如使用JS模型颜色$3Dmol.elementColors.Jmol,那么3D分子模型的样式会是:H氢原子白色O氧原子红色- 元素原子都会配置对应颜色作为区别标识
-
更多视图方法的配置可以看官网文档createViewer
常用的的方法
render()渲染场景内容resize()调整大小,用于窗口大小变化的时候spin(axis, speed)视角变化,自动绕某个轴旋转clear()清空场景所有setConfig(c)修改更新配置setStyle()修改模型全局样式配置- 更多视图对象操作请看GLViewer`
2.2 添加形状并设置样式
添加球体
ts
const sphere= viewer.addSphere({center: {x: 2.0, y: 0, z: 0}, radius: 1.0, color: "red"});
const sphere1=viewer.addSphere({center: {x: -2.0, y: 0, z: 0}, radius: 1.0, color: "red", opacity: 0.5});参数说明
center中心坐标radius半径大小color颜色opacity透明度 可以使用
可以使用updateStyle修改样式
ts
sphere.updateStyle({color: "blue"});
viewer.render();注意:形状修改样式后记得执行 viewer.render才能重新渲染生效。
添加柱体
ts
viewer.addCylinder({
start: {x: 1.0, y: 0.0, z: 0.0},
end: {x: -1.0, y: 0.0, z: 0.0},
radius: 0.2,
color: "white",
fromCap: $3Dmol.CAP.FLAT,
toCap: $3Dmol.CAP.ROUND
});参数说明
-
start开始坐标 -
end结束坐标 -
radius半径大小 -
color颜色 -
fromCap开始端是否圆角,NONE无,FLAT平角, ROUND圆角 -
toCap结束端是否圆角,NONE无,FLAT平角, ROUND圆角

可以开启虚线,多段柱体
ts
viewer.addCylinder({
start: {x: 1.0, y: 0.0, z: 0.0},
end: {x: -1.0, y: 0.0, z: 0.0},
radius: 0.2,
color: "white",
fromCap: $3Dmol.CAP.FLAT,
toCap: $3Dmol.CAP.FLAT,
dashed: true,
dashLength: 0.5,
gapLength: 0.3
});参数说明
dashed是否开启虚线dashLength段长度gapLength间隔长度

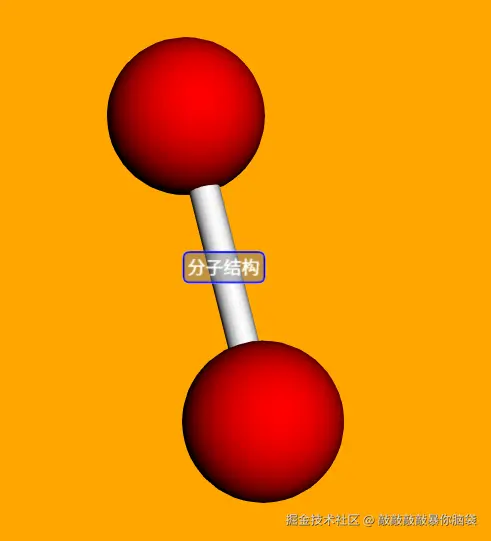
添加标签
ts
viewer.addLabel("分子结构", {
alignment: "center",
font: "sans-serif",
fontSize: 18,
fontColor: "white",
fontOpacity: 1,
borderThickness: 2.0,
borderColor: "blue",
borderOpacity: 1,
backgroundColor: "black",
backgroundOpacity: 0.5,
position: {x: 0.0, y: 0.0, z: 0.0},
inFront: true,
showBackground: true
});参数说明
alignment对齐方式,可选项"topLeft", "topCenter", "topRight", "centerLeft", "center", "centerRight", "bottomLeft", "bottomCenter", "bottomRight",也可以是xy坐标position标签位置inFront是否总在模型前面font字体系列fontSize字体大小fontColor字体颜色fontOpacity字体透明度borderThickness边框宽度borderColor边框颜色borderOpacity边框透明度showBackground是否显示背景backgroundColor背景颜色backgroundOpacity背景透明度
还有其他更多形状,如矩形,箭头,曲线等,可以到官方API文档查看怎么使用
https://3dmol.org/doc/GLViewer.html
2.3 加载3D分子结构模型
通过请求下载获取并添加D分子结构模型,推荐这种方法,3Dmol.js自动识别格式并解析加载,很方便。
ts
download("/1ycr.pdb", viewer, {multimodel: true, frames: true}, (model: any) => {
console.log("🚀 ~ model:", model);
viewer.setStyle({}, {cartoon: {color: "spectrum"}});
viewer.render();
});viewer.setStyle设置全局样式,使得3D分子结构结构以卡通模式显示
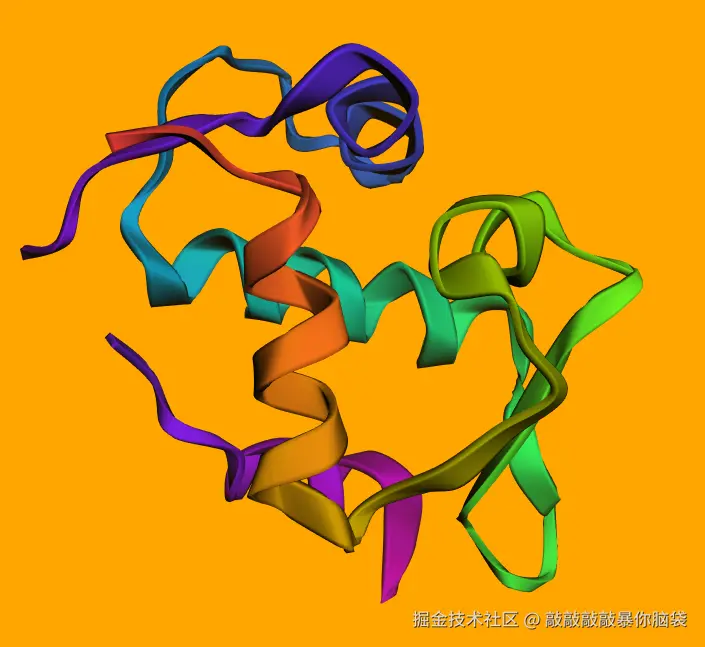
通过打印的模型信息,可以看到模型相关数据有A,B,C三条链,α,β,γ三个角的角度
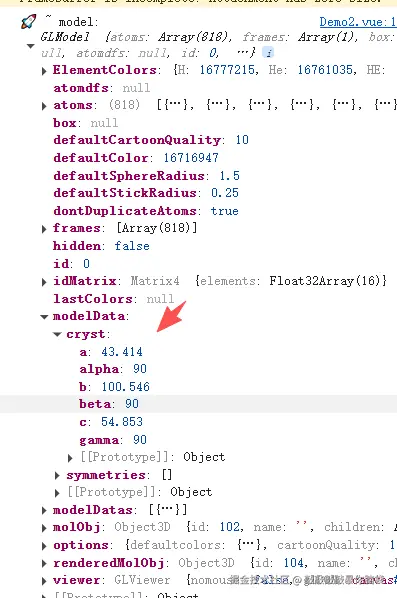
设置分子结构链A为绿色
ts
viewer.setStyle({chain: "A"}, {cartoon: {color: "green"}});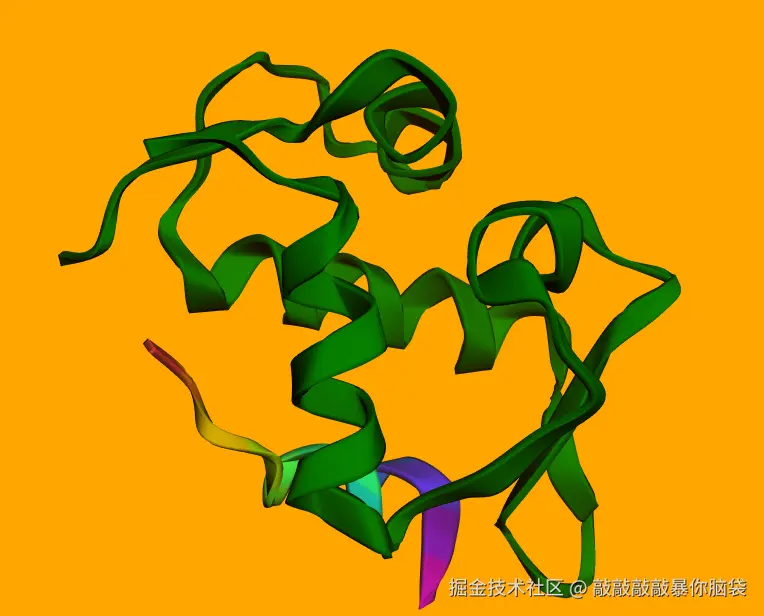
通过模型格式文本解析添加3D分子结构
这是7个水分子的3D分子结构模型文本
txt
symmetry c1
OpenBabel01312416543D
21 14 0 0 0 0 0 0 0 0999 V2000
0.59904 0.98737 -3.22086 O 0 0 0 0 0 0
-1.51456 1.20243 -0.31112 O 0 0 0 0 0 0
0.13045 0.14890 -1.25059 O 0 0 0 0 0 0
-0.20952 -0.68359 -1.35644 H 0 0 0 0 0 0
-0.69349 0.71484 -0.83445 H 0 0 0 0 0 0
-0.05336 1.70041 -3.27063 H 0 0 0 0 0 0
0.39550 0.49975 -2.28181 H 0 0 0 0 0 0
-2.04082 1.71243 -0.64258 H 0 0 0 0 0 0
-1.91286 0.87596 0.33191 H 0 0 0 0 0 0
-0.80317 -1.78194 -1.51703 O 0 0 0 0 0 0
4.83471 2.50607 2.80654 H 0 0 0 0 0 0
-2.85573 -2.53790 -1.84489 O 0 0 0 0 0 0
-0.13238 -2.61116 -1.78592 H 0 0 0 0 0 0
-1.84863 -2.10567 -1.62688 H 0 0 0 0 0 0
-3.18539 -2.82307 -2.85475 H 0 0 0 0 0 0
-3.60191 -2.86990 -1.10817 H 0 0 0 0 0 0
5.44460 1.75213 2.56206 O 0 0 0 0 0 0
6.37580 2.09935 2.45041 H 0 0 0 0 0 0
1.49202 -0.02736 0.38172 O 0 0 0 0 0 0
1.15940 0.32819 1.19843 H 0 0 0 0 0 0
0.74030 0.06841 -0.41057 H 0 0 0 0 0 0
4 3 1 0 0 0
5 3 1 0 0 0
6 1 1 0 0 0
7 1 1 0 0 0
8 2 1 0 0 0
9 2 1 0 0 0
13 10 1 0 0 0
14 10 1 0 0 0
15 12 1 0 0 0
16 12 1 0 0 0
17 11 1 0 0 0
18 17 1 0 0 0
20 19 1 0 0 0
21 19 1 0 0 0
M END
$$$$设置采用Jmol模型颜色风格,
ts
const viewer = $3Dmol.createViewer(container, {
defaultcolors: $3Dmol.elementColors.Jmol,
backgroundColor: "#f9f9f9"
});
viewer.addModel(data, 'sdf');
viewer.setStyle(
{},
{
stick: {
radius: 0.15
},
sphere: {
scale: 0.25
}
}
);- 其中模型格式可选
'pdb', 'sdf', 'xyz', 'pqr', 'mol2',这个需要人工识别格式。非专业人士真不懂,所以还是推荐download viewer.setStyle通过设置全局样式为棍棒模式
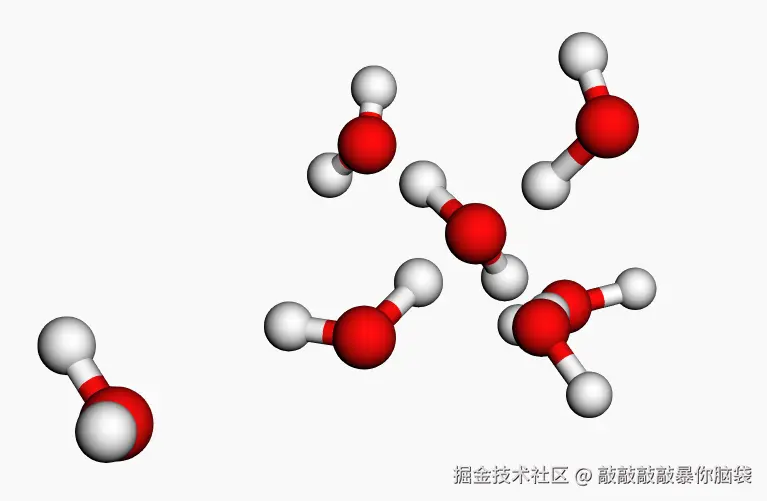
2.4 切换3D分子结构模式
有四种常见的分子展示模式
ts
const modes = [
{label: "球棍模式", value: "ball"},
{label: "线框模式", value: "line"},
{label: "球体模式", value: "spacefill"},
{label: "卡通模式", value: "cartoon"}
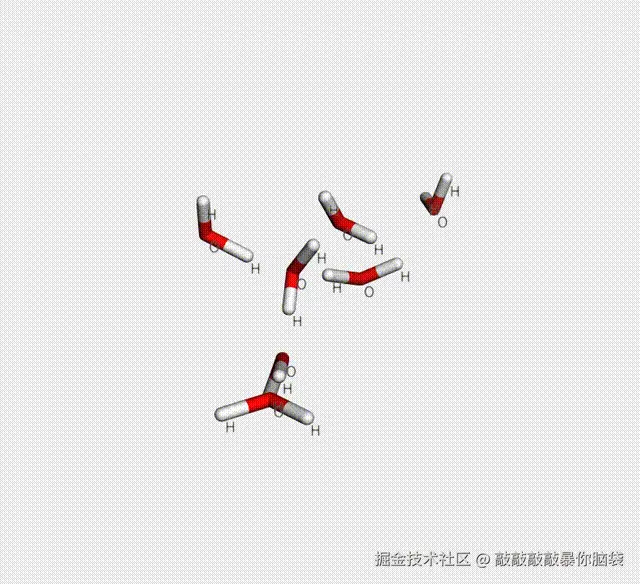
];线框模式的全局样式设置
ts
viewer.setStyle(
{},
{
stick: {
radius: 0.15
}
}
);
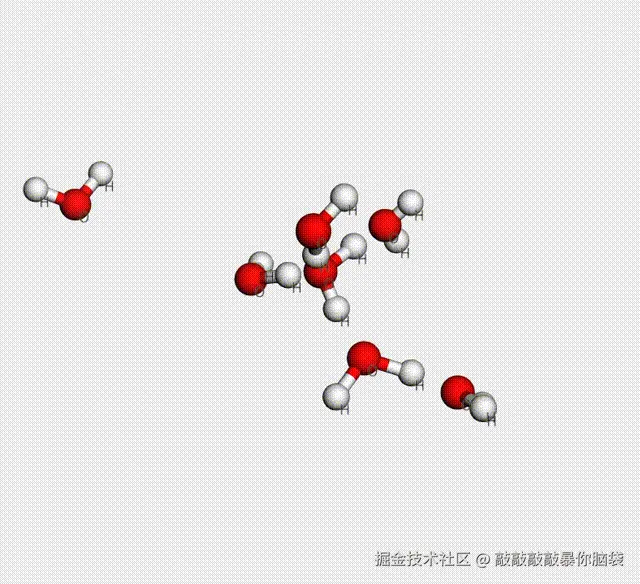
球体模式的全局样式设置
ts
viewer.setStyle(
{},
{
sphere: {
scale: 0.75
}
}
);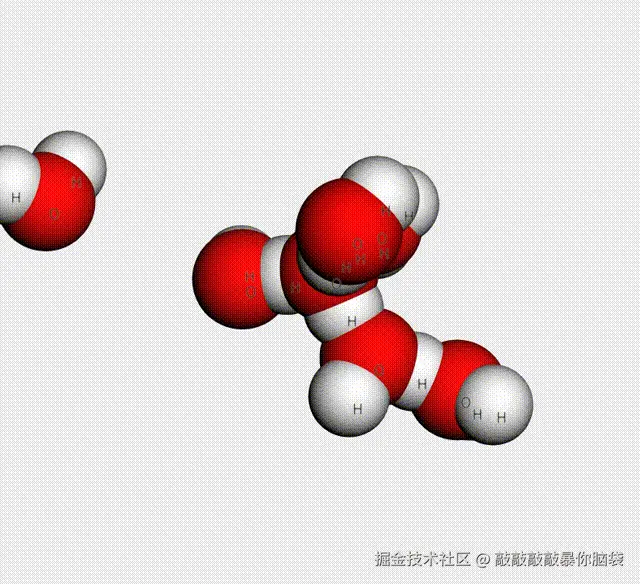 球棍模式的全局样式设置
球棍模式的全局样式设置
ts
viewer.setStyle(
{},
{
stick: {
radius: 0.15
},
sphere: {
scale: 0.25
}
}
);
卡通模式的全局样式设置,注意卡通模式是特殊的,仅适用于蛋白质或核酸二级结构的可视化,如果是水分子之类则可能空白一片或者只有线。
ts
viewer.setStyle({}, {cartoon: {color: "spectrum"}});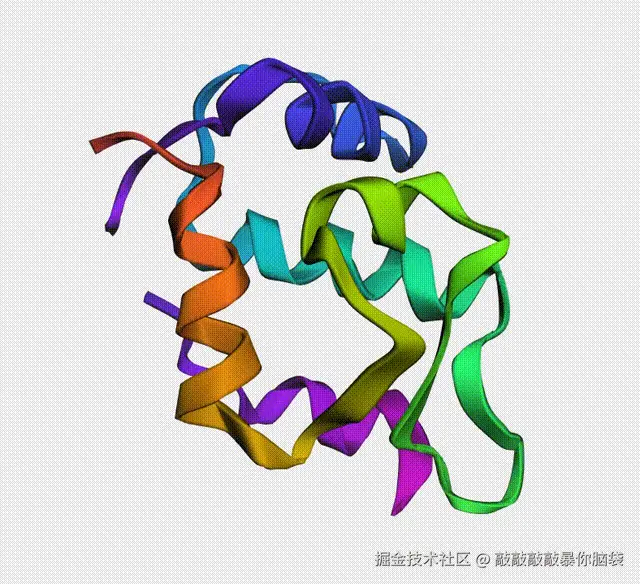
让视图内容自动沿y轴旋转
ts
viewer.spin("y");给模型原子添加标签
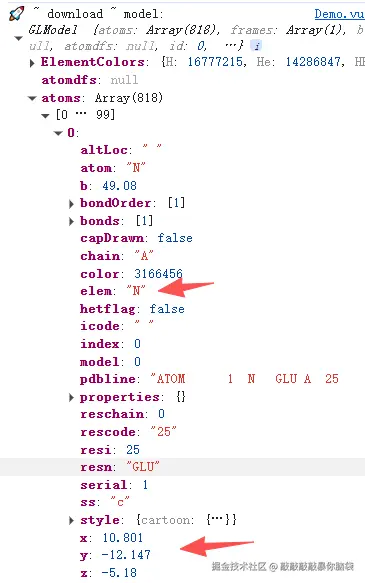
打印模型信息,可以看到在atoms属性有所有原子的信息
elem该原子的元素- xyz对应原子的位置
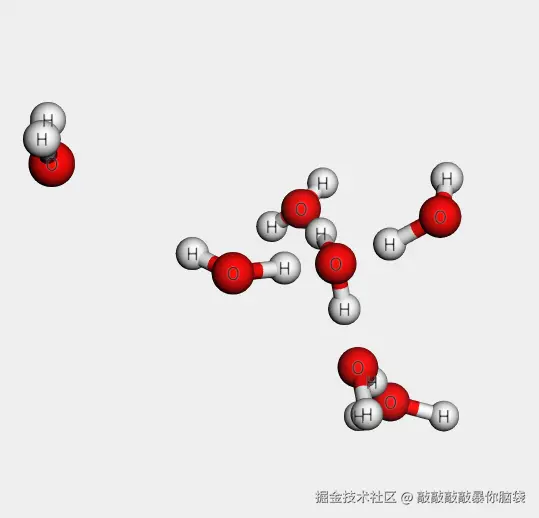
遍历所有原子即可添加所有元素标签
ts
model.atoms.forEach((item: any) => {
viewer.addLabel(item.elem, {
alignment:'center',
position: {
x: item.x,
y: item.y,
z: item.z
},
fontColor: "black",
showBackground: false
});
});
当然可以通过移除所有标签和模型,添加新的模型和标签
ts
viewer.removeAllLabels();
viewer.removeAllModels();销毁视图前记得清空
ts
viewer.clear();3. 总结
3Dmol.js的缩放操作跟Three.js的OrbitControls有些不同,滚轮缩放是相反的,向前滚是缩小,向后滚是放大,并且找了一圈配置项,没有发现修改缩放操作的配置。3Dmol.js的不能移动操作,只能旋转。不过可以通过viewer.setView()来配置视角位置。
- 3D分子结构不论大小都可以通过
viewer.zoomTo()来适配视角,可以通过viewer.zoom(zoom,time)设置缩放等级
3Dmol.js限制缩放视角大小的参数很鸡肋,是根据相机距离物体的距离根据判断条件,而不同分子结构的大小是不限的,有的总大小可能是100+,有的总大小只有0.5,那么限制值就得谨慎计算出合适值才行,否则就会导致显示的分子结构太小或太大。
ts
const viewer= $3Dmol.createViewer(container,{
lowerZoomLimit: 100,
upperZoomLimit: 200,
})
viewer.setZoomLimits(lower,upper);通过viewer.getView()返回视角参数[pos.x, pos.y, pos.z, rotationGroup.position.z, q.x, q.y, q.z, q.w ],对应平移、缩放和旋转四元数,可以获取当前视角距离z
然后我以为通过zoom(minZoom)和zoom(maxZoom)获取对应缩放值的视角距离限制,结果发现lowerZoomLimit和upperZoomLimit跟getView的z距离不是一个东西。
ts
viewer.zoom(0.5);
const v = viewer.getView();
const minZ = v[3];
viewer.zoom(3);
const v1 = viewer.getView();
const maxZ = v1[3];
viewer.setZoomLimits(minZ, maxZ);
viewer.render();于是,查看了一下文档,有个setViewChangeCallback方法可以监听视角变化
ts
viewer.setViewChangeCallback((view: number[]) => {
console.log("🚀 ~ view:", view[3]);
});
可以看到,物体离摄像机越远,即缩小,则view的z距离值越小,物体离摄像机越近,即放大,则view的z距离值越大。
3.1 实现通用视角范围限制
第一步,遍历模型元素,获取其包围框范围
ts
const box = {
minx: Number.MAX_SAFE_INTEGER,
miny: Number.MAX_SAFE_INTEGER,
minz: Number.MAX_SAFE_INTEGER,
maxx: Number.MIN_SAFE_INTEGER,
maxy: Number.MIN_SAFE_INTEGER,
maxz: Number.MIN_SAFE_INTEGER
};
model.atoms.forEach((item: any) => {
box.minx = Math.min(box.minx, item.x);
box.maxx = Math.max(box.maxx, item.x);
box.miny = Math.min(box.miny, item.y);
box.maxy = Math.max(box.maxy, item.y);
box.minz = Math.min(box.minz, item.z);
box.maxz = Math.max(box.maxz, item.z);
});
const size = {
x: box.maxx - box.minx,
y: box.maxy - box.miny,
z: box.maxz - box.minz
};
const distance = Math.max(size.x, size.y, size.z);第二步,zoomTo()后获取最佳适配视角作为基础距离
ts
const v = viewer.getView();
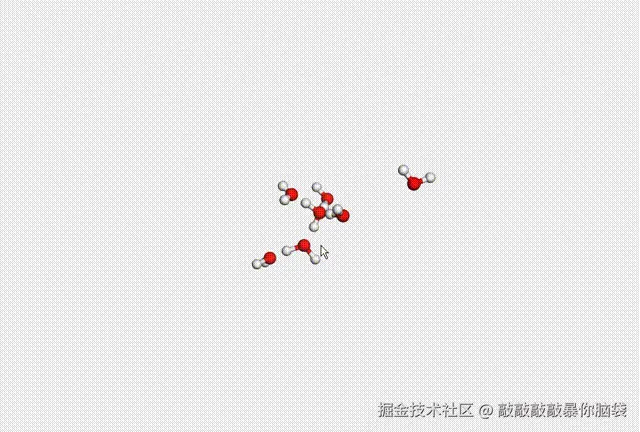
const baseDistance = v[3];第三步,根据包围框大小,设置缩放最大最小距离z,监听视角变化,基于基础距离baseDistance进行对比和限制
ts
const minDistance = -distance * 3;
const maxDistance = distance * 2;
viewer.setViewChangeCallback((view: number[]) => {
const z = view[3];
if (z < baseDistance + minDistance) {
//缩小限制
view[3] = baseDistance + minDistance;
viewer.setView(view);
} else if (z > baseDistance + maxDistance) {
//放大限制
view[3] = baseDistance + maxDistance;
viewer.setView(view);
}
});
4. Github地址
https://github.com/xiaolidan00/3dmol-project 
参考