本文基于HTML、Python与MySQL开发了一套基因程序可视化大屏系统,以科技风格的大屏展示为核心,结合人体轮廓图交互功能,让基因数据呈现更具冲击力与实用性。本文将从项目亮点、技术架构、部署流程及核心功能等方面,为大家全面解析该系统。
一、项目核心亮点:
相较于传统基因数据展示工具,该系统最大的优势在于"可视化+交互性"的深度融合,既满足科研数据的精准呈现,又兼顾了视觉体验与操作便捷性,核心亮点如下:
- 酷炫大屏:科技感拉满的视觉体验
系统采用全黑色科技风格设计,搭配动态粒子背景效果,数据加载与更新过程中伴随流畅过渡动画,打破了科研工具"单调刻板"的固有印象。无论是实验室展示还是学术汇报,都能快速吸引注意力,让数据呈现更具说服力。
- 交互式人体轮廓:基因数据的"空间定位"
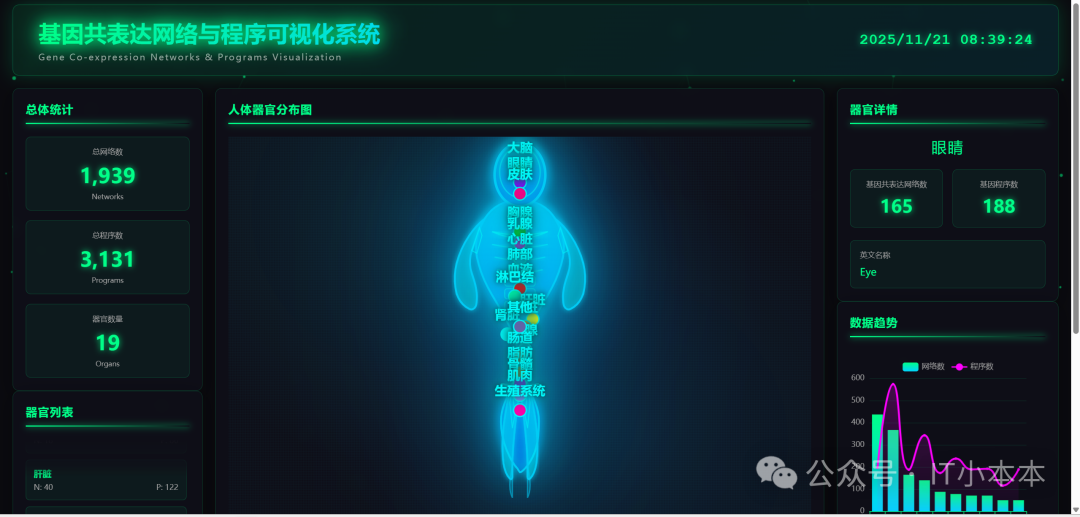
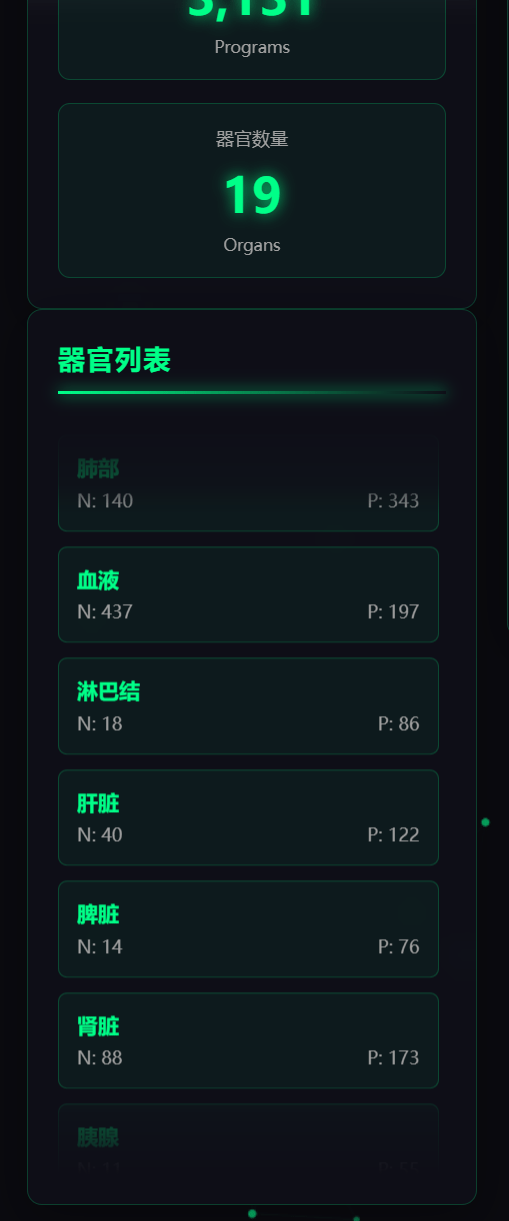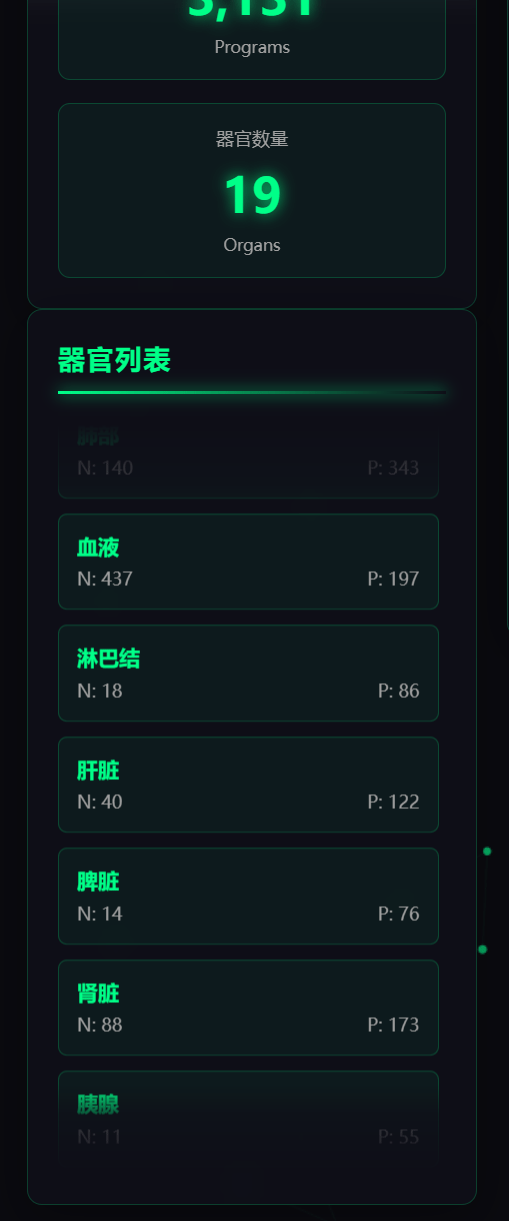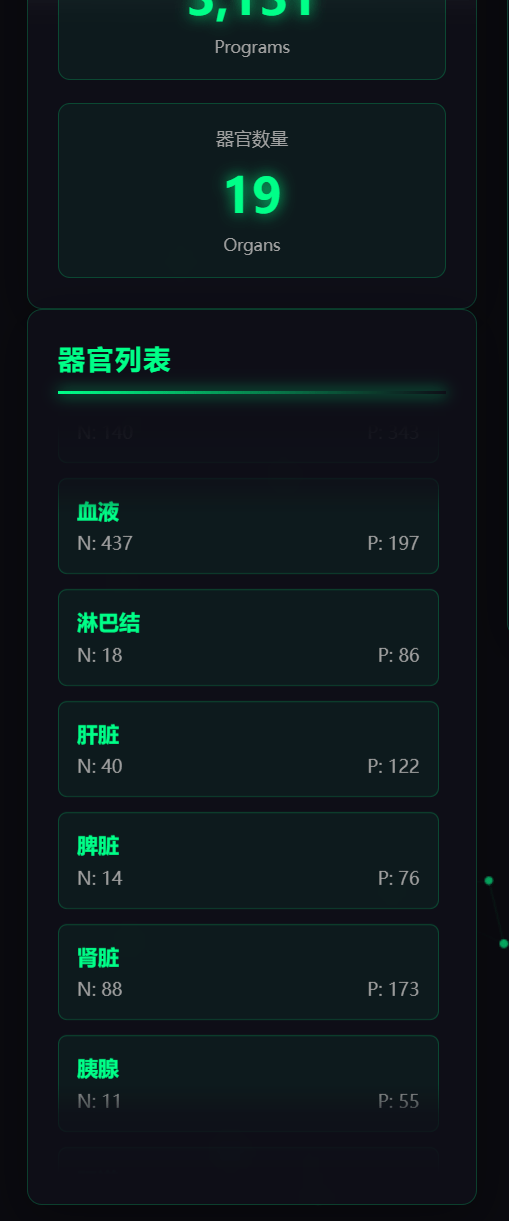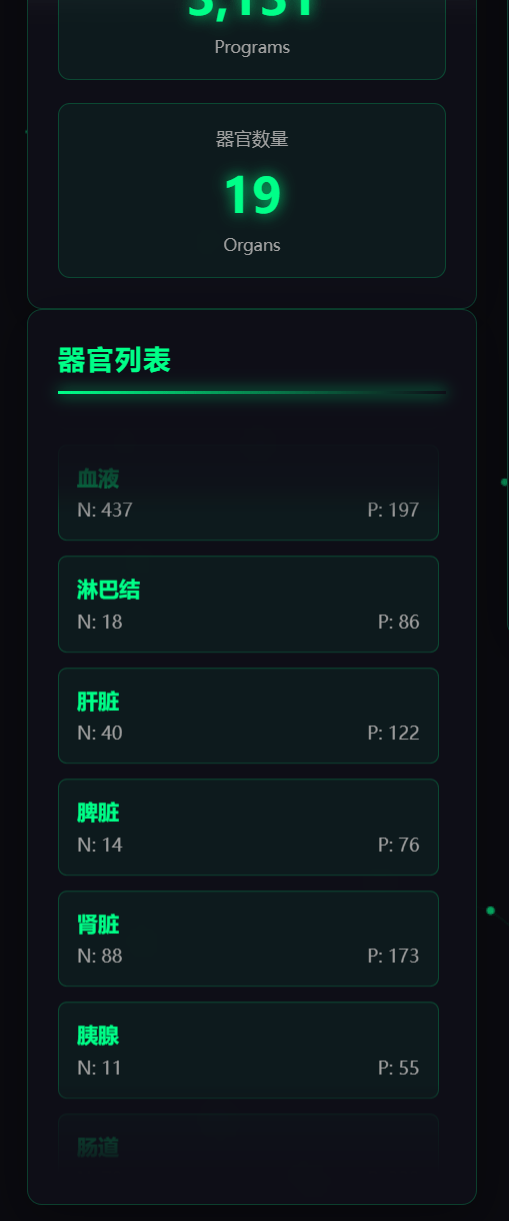
基于技术构建的交互式人体轮廓图是系统的核心创新点之一。图中精准标注了大脑、心脏、肝脏等19个关键器官的位置,点击任意器官标记点或侧边列表项,即可实时显示该器官对应的基因共表达网络数量(N值)与基因程序数量(P值),让原本抽象的"基因-器官关联数据"变得空间化、具象化。
- 多维数据可视化:从趋势到细节全掌握
集成ECharts可视化库,支持折线图、柱状图、饼图等多种图表类型,可灵活展示不同器官的基因数据对比趋势、整体数据分布等信息。顶部状态栏实时显示总网络数、总程序数及当前时间,实现"宏观统计+微观细节"的全方位数据掌控。
- 稳定可靠:数据存储与实时更新双保障
采用MySQL数据库进行完整的数据存储与管理,配合Python Flask后端构建的API接口,支持数据的批量导入、单点更新与实时查询。前端通过异步请求实现数据动态加载,无需刷新页面即可获取最新科研数据,确保科研过程中数据的时效性。
二、技术栈解析:轻量高效的全栈架构
系统采用"前端可视化+后端接口+数据库存储"的经典三层架构,技术选型聚焦"轻量、高效、易扩展",非常适合科研团队快速部署与二次开发。
- 前端:交互与视觉的双重实现
-
基础框架:HTML5+CSS3+JavaScript,利用HTML5语义化标签优化页面结构,通过CSS3实现粒子背景、渐变效果等视觉元素,借助原生JS完成DOM操作与接口请求。
-
可视化工具:ECharts,高效渲染各类统计图表,支持动态数据更新与交互事件绑定,满足科研数据的多维度展示需求。
-
交互核心:SVG技术,构建可交互的人体轮廓图,实现器官标记点的点击响应、数据弹窗等功能,兼具跨浏览器兼容性与渲染效率。
- 后端:轻量灵活的接口服务
采用Python Flask框架开发后端服务,其优势在于"轻量易上手、路由配置简洁、扩展性强"。核心功能包括:
-
数据库交互:通过MySQL Connector实现与数据库的连接、查询、更新等操作,封装统一的数据处理函数。
-
API接口开发:提供RESTful风格接口,支持器官数据的查询(GET)、更新(PUT)等核心操作,满足前端数据交互需求。
-
配置管理:支持直接修改代码配置或通过独立配置文件管理数据库参数,提升部署灵活性。
- 数据库:稳定高效的数据存储
选用MySQL作为数据存储引擎,具备以下优势:
-
结构化存储:通过数据表精准定义器官数据字段,支持数据完整性约束,确保科研数据的准确性。
-
高效查询:支持索引优化,针对器官ID、名称等常用查询条件建立索引,提升数据查询效率。
-
便捷初始化:提供数据库初始化脚本(database.sql),支持一键执行创建数据表与测试数据,简化部署流程。
三、快速部署:5分钟上手的实操指南
系统部署流程简洁明了,无需复杂的环境配置,无论是Windows还是Linux/Mac系统,都能快速完成搭建,具体步骤如下:
- 环境准备
-
安装MySQL数据库:确保服务正常启动,记录用户名与密码(后续配置需使用)。
-
安装Python环境:推荐Python 3.7及以上版本,确保pip工具可用。
- 数据库初始化
通过以下两种方式之一创建数据库与数据表:
mysql -u root -p < database.sql
或手动登录MySQL,执行database.sql文件中的SQL语句,完成数据库初始化。
- 配置与启动
- 安装Python依赖:进入项目根目录,执行命令安装所需依赖包。
pip install -r requirements.txt
- 配置数据库连接:两种配置方式可选
方式一:直接修改app.py文件第11-17行的数据库配置
DB_CONFIG = {
'host': 'localhost',
'user': 'root', # MySQL用户名
'password': 'your_password', # MySQL密码
'database': 'gene_programs_db',
'charset': 'utf8mb4'
}方式二(推荐):复制config.example.py为config.py,修改其中的数据库参数,然后在app.py中导入配置。
- 启动系统:根据操作系统选择对应命令
直接执行:python app.py
- 访问系统
打开Chrome或Edge浏览器(推荐,以获得最佳视觉效果),输入地址:http://localhost:5000,即可进入系统。
四、核心功能与API详解
系统的核心价值在于"数据交互与可视化",以下将重点解析核心功能的使用方式与API接口规范,方便开发人员二次开发。
- 核心功能演示
-
器官数据查询:点击人体轮廓图上的器官标记点,或侧边栏的器官列表项,右侧面板将实时显示该器官的N值(基因共表达网络数)与P值(基因程序数)。
-
数据统计与趋势:顶部状态栏展示总网络数、总程序数,右侧图表区域可切换不同图表类型,对比各器官数据差异与分布趋势。
-
实时数据更新:后端数据更新后,前端通过定时异步请求接口,实现数据的自动刷新,无需手动刷新页面。
- 关键API接口
系统提供RESTful风格API,支持与第三方系统或科研工具集成,核心接口如下:
(1)获取所有器官数据
-
请求方式:GET
-
请求地址:/api/organs
-
响应示例:
{
"success": true,
"data": [
{
"id": 1,
"organ_name": "Brain",
"organ_name_cn": "大脑",
"networks_count": 367,
"programs_count": 574,
"position_x": 50,
"position_y": 15,
"color": "#00ff88"
}
],
"total": 19
}
(2)获取单个器官详情
-
请求方式:GET
-
请求地址:/api/organs/(将替换为器官ID)
-
功能:返回指定ID的器官完整数据,用于详情展示。
(3)更新器官数据
-
请求方式:PUT
-
请求地址:/api/organs/
-
功能:更新指定器官的N值、P值等数据,支持科研过程中数据的实时修正。
(4)获取统计信息
-
请求方式:GET
-
请求地址:/api/statistics
-
功能:返回系统总体数据统计,包括总器官数、总网络数、总程序数等。
五、注意事项
- 部署与使用注意事项
-
确保MySQL服务在系统启动前已正常启动,否则将导致数据库连接失败。
-
数据库配置信息需准确填写,若使用远程MySQL服务,需将host参数修改为远程服务器IP,并开放对应端口权限。
-
视觉效果与交互体验在Chrome、Edge浏览器中最佳,不建议使用IE浏览器。
这款基因程序可视化大屏系统通过"前端视觉化+后端轻量化+数据精准化"的设计思路,有效解决了基因数据展示抽象、交互不便的问题。无论是实验室日常科研还是学术成果展示,都能为科研人员提供高效、直观的数据支撑。