ANNEVO 官网
ANNEVO 是一种基于深度学习的从头基因注释方法,用于解析基因组功能。该方法能够直接从基因组序列中,对远端序列信息以及不同物种间的联合进化关系进行建模。
https://github.com/xjtu-omics/ANNEVO #官网1 ANNEVO 核心特点
ANNEVO 是基于"混合专家模型(MoE)"开发的基因组语言模型,其核心优势集中于三大维度,精准破解传统基因注释工具的固有痛点,具体表现如下:
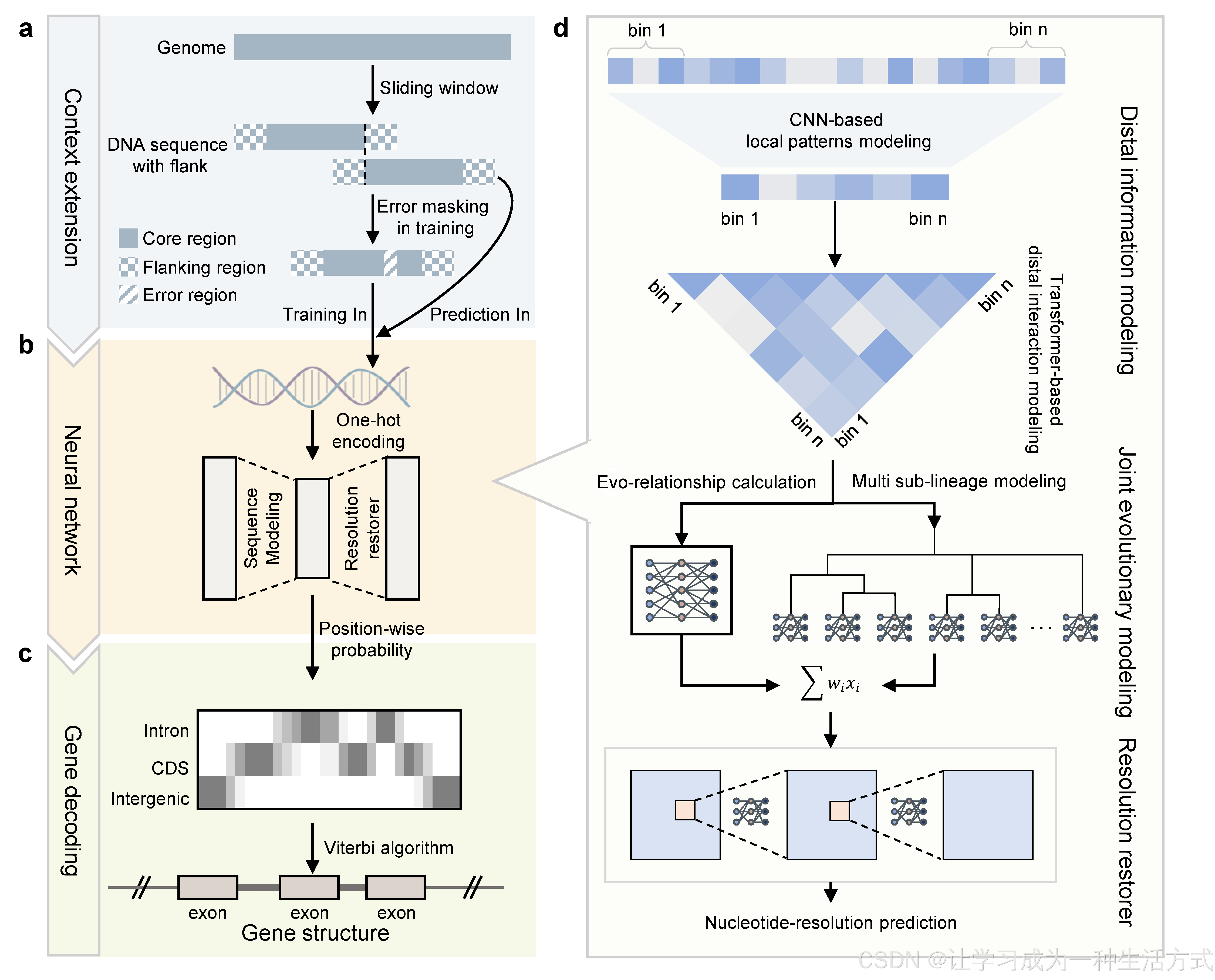
1.1 建模能力革新:兼顾进化关系与长片段依赖
传统工具(如 Augustus)多依赖单一物种训练模型,难以捕捉跨类群的进化信号。ANNEVO 创新搭载"联合进化建模模块",整合 8 个亚谱系的专家网络,可同步解析垂直遗传与水平基因转移引发的序列变异,进化视角更全面。
针对超长基因(如人类 365kb 的 TRIO 基因),传统工具常出现碎片化预测问题。ANNEVO 借鉴 Hi-C 技术思路,通过 Transformer 架构建模 40kb 范围内的远端序列相互作用,实现超长基因的完整结构重建,解决了长片段注释的核心难题。
1.2 无证据依赖:单一输入实现高精度注释
无需依赖 RNA-seq、蛋白质序列等外部实验数据,仅输入基因组 FASTA 文件即可完成高精度基因注释。这一特性对缺乏实验数据的新测序物种(占 NCBI RefSeq 物种总数的 46%)而言,提供了高效可行的注释解决方案,极大拓展了工具的适用场景。
1.3 效率双优:速度与参数效率同步提升
计算速度较经典工具 Augustus 提升 2 倍,实际应用中,拟南芥基因组注释仅需 6 分钟,人类基因组注释耗时仅 4.5 小时,大幅降低大规模基因组项目的时间成本。
参数总量仅 170 万,远低于同类深度学习工具 Helixer(300 万参数),资源占用率低,普通 GPU 集群即可支撑运行,降低了技术落地的硬件门槛。
1.4 自带纠错功能:提升现有注释质量
除从头预测基因外,还可修正现有注释中的错误。在甘蓝基因组测试中,ANNEVO 成功纠正 4% 的 BUSCO 核心基因注释问题,涵盖基因融合、断裂、缺失等典型错误,且所有修正结果均经 RNA-seq 实验验证,可靠性有保障。
2 与主流从头注释工具的对比
| 对比维度 | ANNEVO | 传统工具 |
|---|---|---|
| 准确率 | 566 个物种测试中,核苷酸 F1 值提升 7.5%-22.3%,BUSCO 值提升 9%-34.5% | 依赖近缘物种训练,跨类群预测准确率大幅下降 |
| 超长基因预测 | 对 >100kb 基因的完整预测率达 50% 以上 | 仅 25.5%(以 Augustus 为例),易出现碎片化注释 |
| 外部数据依赖 | 无依赖,仅需基因组序列 | 需 RNA-seq/蛋白质数据(如 BRAKER3)或类群特异性训练(如 Augustus) |
| 计算效率 | 拟南芥:6 分钟;人类:4.5 小时 | 拟南芥:16 小时(BRAKER3);人类:超 10 小时 |
| 注释纠错 | 可修正基因融合、断裂等注释错误 | 仅能预测新基因,无纠错功能 |
注意:ANNEVO 输入文件无需屏蔽重复序列,简化预处理流程。
当前局限性与拓展空间
-
功能覆盖:目前仅支持蛋白编码基因注释,暂未涵盖非编码 RNA(ncRNA)、转座子等基因组元件;
-
类群匹配:模型按真菌、胚胎植物、无脊椎动物、哺乳动物、其他脊椎动物五大类群预训练,需严格匹配输入物种类群,否则可能影响预测准确率;
-
参数优化:超参数未进行系统优化,进阶用户可结合具体物种特性调整参数,进一步提升注释性能。
3 安装与使用指南
ANNEVO 代码已开源,免费向学术、政府及非营利机构开放使用,操作流程简洁高效。推荐在 Linux 平台下,使用 conda 虚拟环境完成安装与部署。
3.1 安装步骤
bash
### 1.1 获取源代码
git clone https://github.com/xjtu-omics/ANNEVO.git
cd ANNEVO
### 1.2 环境安装(二选一)
# 方式一:快速安装(适用于 CUDA 版本 > 12.1 的用户)
conda env create -f ANNEVO.yml -n your_env_name
# 方式二:手动安装(适用于 CUDA 版本 ≤ 12.1 的用户,需手动调整 PyTorch 版本)
# 1. 创建并激活环境
conda create -n ANNEVO python=3.10
conda activate ANNEVO
# 2. 安装 PyTorch(需根据 GPU 配置调整,推荐参考 PyTorch 官方指引:https://pytorch.org/get-started/previous-versions/)
# 注意:ANNEVO 基于 PyTorch 1.10 开发,但完全兼容 PyTorch 2.x;H100/A100 等高端 GPU 建议使用 PyTorch 2.x 避免报错
# 示例:安装 PyTorch 2.1.0(适配 CUDA 12.1)
conda install pytorch==2.1.0 torchvision==0.16.0 torchaudio==2.1.0 pytorch-cuda=12.1 -c pytorch -c nvidia
# 3. 安装其他依赖包
pip install -r requirements.txt
### 1.3 验证 CUDA 可用性
python -c "import torch; print(torch.__version__, torch.version.cuda, torch.cuda.is_available())"2. 使用指南
ANNEVO 的核心注释逻辑:只要训练集包含目标物种所属"纲"级别的物种(即使仅1-2个),即可实现良好的注释效果。可参考 train_class.txt 查看训练集涵盖的类群信息。
### 2.1 一步式执行(推荐资源充足时使用)
# 核心指令:线程数(threads)建议拉满以提升速度,多GPU环境自动适配
python annotation.py --genome path_to_genome --model_path path_to_model --output path_to_gff --threads 48
# 资源适配:GPU内存不足时,可通过 --batch_size 调整(例:--batch_size 8 仅需约 3G 显存)
### 2.2 分步式执行(推荐GPU资源充足但CPU有限的场景)
# 阶段1:核苷酸信息预测(推荐在GPU资源丰富的环境执行)
python prediction.py --genome path_to_genome --model_path path_to_model --model_prediction_path path_to_save_predction
# 阶段2:基因结构解码(推荐在CPU资源丰富的环境执行)
python decoding.py --genome path_to_genome --model_prediction_path path_to_save_predction --output path_to_gff --threads 48
### 2.3 演示数据运行(示例:拟南芥第4号染色体注释)
# 演示数据路径:./example
# Arabidopsis_chr4_genome.fna:拟南芥第4号染色体序列
# Arabidopsis_chr4_annotation.gff:拟南芥第4号染色体参考注释
# 一步式执行演示
python annotation.py --genome example/Arabidopsis_chr4_genome.fna --model_path ANNEVO_model/ANNEVO_Embryophyta.pt --output gff_result/Arabidopsis_chr4_annotation.gff --threads 48
# 分步式执行演示
python prediction.py --genome example/Arabidopsis_chr4_genome.fna --model_path ANNEVO_model/ANNEVO_Embryophyta.pt --model_prediction_path prediction_result/Arabidopsis_chr4/model_prediction.h5
python decoding.py --genome example/Arabidopsis_chr4_genome.fna --model_prediction_path prediction_result/Arabidopsis_chr4/model_prediction.h5 --output gff_result/Arabidopsis_chr4_annotation.gff --threads 484、模型重训练与微调
4.1 模型重训练(新增物种类群或特定支系)
# 1. 配置参数
train_species_list="训练物种类表" # 用于模型训练的物种
val_species_list="验证物种类表" # 用于模型验证的物种
h5_data_path="h5文件存储路径" # 训练数据存储位置
mkdir -p tmp # 创建临时文件夹
# 2. 清空历史文件(每次运行前执行)
rm -f ${h5_data_path}/train.h5 ${h5_data_path}/train_with_intergenic.h5
rm -f ${h5_data_path}/val.h5 ${h5_data_path}/val_with_intergenic.h5
# 3. 生成训练集数据
for species_name in "${train_species_list[@]}"; do
path_to_genome="该物种基因组路径"
path_to_annotation="该物种注释文件路径"
# 过滤注释文件中重复基因ID等问题(避免Biopython解析报错)
python src/filter_wrong_record.py --input_file ${path_to_annotation} --output_file "tmp/tmp_${species_name}.gff"
# 转换基因组与注释为模型可读取的H5格式
python generate_datasets.py --genome ${path_to_genome} --annotation "tmp/tmp_${species_name}.gff" --output_file "${h5_data_path}/train" --threads 64
rm -f "tmp/tmp_${species_name}.gff" # 清理临时文件
done
# 4. 生成验证集数据
for species_name in "${val_species_list[@]}"; do
path_to_genome="该物种基因组路径"
path_to_annotation="该物种注释文件路径"
python src/filter_wrong_record.py --input_file ${path_to_annotation} --output_file "tmp/tmp_${species_name}.gff"
python generate_datasets.py --genome ${path_to_genome} --annotation "tmp/tmp_${species_name}.gff" --output_file "${h5_data_path}/val" --threads 64
rm -f "tmp/tmp_${species_name}.gff"
done
# 5. 启动模型训练
python model_train.py --h5_path ${h5_data_path} --model_save_path path_to_new_model.pt4.2 模型微调(目标物种近缘种稀缺时)
# 1. 配置参数
fine_tune_species_list="微调物种类表"
h5_data_path="h5文件存储路径"
mkdir -p tmp
# 2. 清空历史文件
rm -f ${h5_data_path}/fine_tune.h5 ${h5_data_path}/fine_tune_with_intergenic.h5
# 3. 生成微调数据集
for species_name in "${fine_tune_species_list[@]}"; do
path_to_genome="该物种基因组路径"
path_to_annotation="该物种注释文件路径"
python src/filter_wrong_record.py --input_file ${path_to_annotation} --output_file "tmp/tmp_${species_name}.gff"
python generate_datasets.py --genome ${path_to_genome} --annotation "tmp/tmp_${species_name}.gff" --output_file "${h5_data_path}/fine_tune" --threads 64
rm -f "tmp/tmp_${species_name}.gff"
done
# 4. 启动微调(基于ANNEVO预训练模型)
python fine_tune.py --model_path path_to_existing_model.pt --model_save_path path_to_new_model.pt --h5_path ${h5_data_path}5 参考文献
Kai Ye, Pengyu Zhang, Tun Xu et al. Highly accurate ab initio gene annotation with ANNEVO, 23 April 2025, PREPRINT (Version 1) available at Research Square [https://doi.org/10.21203/rs.3.rs-6402260/v1\]