目录标题
-
- 要求
- 1、根据要求配置环境
- 2、对已给文件进行分析
-
- [2.1 简单阅读论文了解背景](#2.1 简单阅读论文了解背景)
- [2.2 train.csv和vaild.csv 数据集分析](#2.2 train.csv和vaild.csv 数据集分析)
- [2.3 了解基本实现](#2.3 了解基本实现)
-
- [2.3.1 data_processing.py](#2.3.1 data_processing.py)
- [2.3.2 create_embeding.py](#2.3.2 create_embeding.py)
- [2.3.3 配置yaml文件](#2.3.3 配置yaml文件)
- 3、项目设计model
-
- 3.1、生成嵌入脚本create_embeding.py
- 3.2、加载数据和构件图脚本data_loader.py
-
- [3.2.1 加载已经生成的嵌入文件](#3.2.1 加载已经生成的嵌入文件)
- [3.2.2 根据嵌入文件从csv文件中构建图](#3.2.2 根据嵌入文件从csv文件中构建图)
- [3.2.3 加载数据](#3.2.3 加载数据)
- 3.3、配置模块config.py
- 3.4、最重要的模型model.py
- 3.4、训练脚本train.py
- 3.5、预测脚本predict.py
- 3.6、配置测试集预测脚本main.py
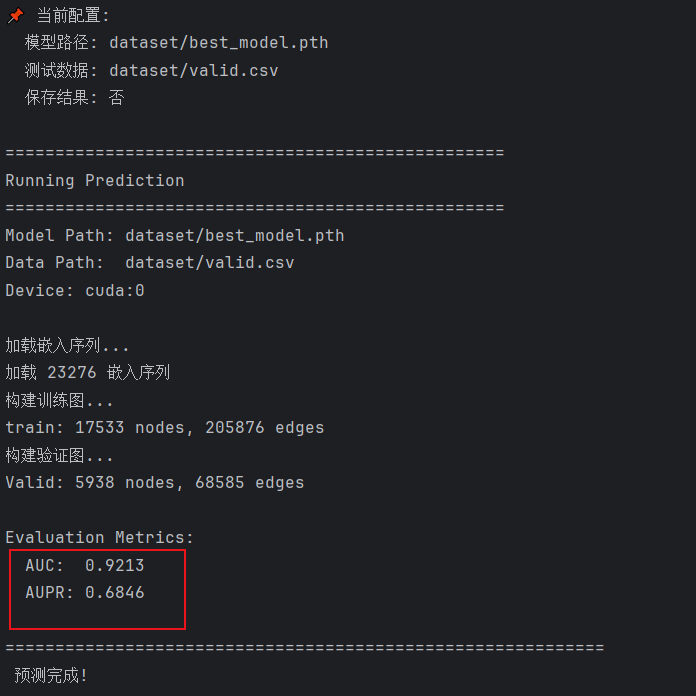
- 4、测试结果
要求
需要使用机器学习的方法,训练模型,完成生物分子相互作用的预测任务,并撰写报告。
- 模型性能判别的标准是AUC数值大小。
- 需要在代码文件中注释用的什么机器学习方法,每个函数功能需要注释(输入,输出)
- 要求将训练好的模型保存好并提交,所有的文件放在model文件夹中,最后测试时直接调用这个模型进行测试即可
- 代码文件的最下面放训练模型和预测模型的函数,命名为train、predict
要有main函数 ,直接运行main函数可以导入训练好的模型并测试测试集的性能,最后打印输出测试集的AUC值。测试集文件只提供名称,代码提交时设置好即可
数据内容 :本研究的数据集由 TCR(T细胞受体)与Epitope(抗原表位)序列配对组成。每条样本包含一个 Epitope 序列、一个 CDR3β 序列及其对应标签。
标签:标签 1 表示该配对具有真实结合关系,0 表示随机生成的非结合样本。
1、根据要求配置环境

进入conda创建环境python=3.11.10
python
conda create -n ML python=3.11.10激活环境
python
conda activate ML创建torch==2.5.1版本
python
pip install torch==2.5.1 torchvision torchaudio --extra-index-url https://download.pytorch.org/whl/cu121配置其他库,因为比较少就一个一个安装了
python
pip install numpy==2.1.3
pip install pandas==2.2.3
pip install tqdm
pip install scikit-learn==1.5.2
pip install subword-nmt查看配置结果
python
pip list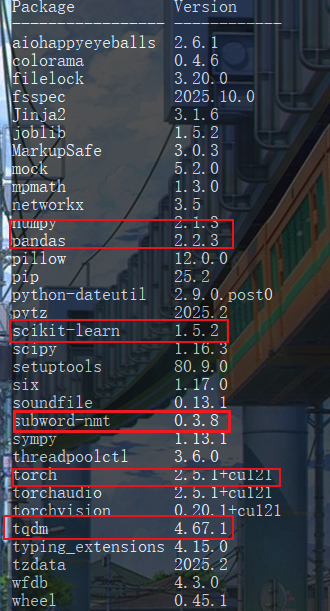
2、对已给文件进行分析
2.1 简单阅读论文了解背景
- 本课题研究了T细胞受体简称TCR 和抗原表位简称Epitope的结合关系,这种结合关系是免疫系统的基础,准确的预测谁与谁的结合,对于了解免疫系统的疾病和开发针对免疫系统的疫苗至关重要。
- 现有的传统方法只关注TCR和Epitope的序列 ,忽视了他们在人体内形成的复杂的相互作用网络及拓扑机构。
- 本文提出了GTE一种图学习框架 ,这是一个新型的图神经网络模型,他不再是孤立地看待每一对序列,而是将所有序列看做图中的节点,通过学习这个网络拓扑结构来预测关系。
- 本文创新点引入了动态边缘更新来解决负样本不足和Deep AUC最大化来解决数据不平衡优化模型性能。
2.2 train.csv和vaild.csv 数据集分析


CDR3.beta: T 细胞受体 β \beta β 链的 CDR3 序列 (TCR CDR3B sequence)。这是 T 细胞受体上用于识别抗原表位的关键区域序列
Epitope: 抗原表位序列 (Epitope sequence)。这是被 T 细胞识别的外来分子序列 。
Label: 标签 (Label)。表示该 Epitope-CDR3B 配对是否具有真实的结合关系 。
2.3 了解基本实现
2.3.1 data_processing.py
这是函数是从csv文件中加载训练数据和测试数据,从一个pickle文件加载节点的嵌入向量,转换成PyTorch Geometric 可以使用的图格式。图中节点表示 "Epitope"(抗原决定簇)和"CDR3.beta"(T 细胞受体的一个区域),边表示它们之间的相互作用,标签 y 表示这种相互作用的类型或强度。
python
import torch
import pickle
import numpy as np
from torch_geometric.data import Data
import pandas as pd
from arg_parser import parse_args
import os
import yaml
def TEINet_embeddings_5fold(config_path):
"""
加载数据、节点的嵌入向量,并为 TEINet 模型将数据转换为 PyTorch Geometric 的 Data 对象。
该函数通常用于 K-fold 交叉验证的其中一折(fold)。
Args:
config_path (str): YAML 配置文件路径,包含数据路径、嵌入路径和训练/测试文件列表。
Returns:
list: 包含两个 PyTorch Geometric Data 对象的列表:[训练集 Data, 测试集 Data]。
"""
# 假设 parse_args() 函数返回的参数对象包含 'split' 属性
# 'split' 用于指定在配置文件中加载哪个交叉验证折(例如 'fold0', 'fold1' 等)
args = parse_args()
# --- 1. 加载配置和嵌入 ---
with open(config_path) as file:
# 从 YAML 文件加载配置字典
config = yaml.safe_load(file)
with open(config["embeddings_path"], 'rb') as f:
# 从指定的路径加载预计算的节点嵌入字典
# 键为 Epitope 或 CDR3.beta 序列,值为对应的嵌入向量(如 numpy 数组)
embedding_dict = pickle.load(f)
# 从配置中获取当前折的训练和测试文件列表
# args.split 决定使用哪个折(如 config['fold0'])
train_file_list = config[args.split]['train_data']['file_list']
test_file_list = config[args.split]['test_data']['file_list']
# 获取数据文件的基本路径
file_path = config['path']
# --- 2. 加载和合并训练数据 ---
train_data = []
for file_name in train_file_list:
# 读取单个训练数据文件 (CSV)
data = pd.read_csv(os.path.join(file_path, file_name))
train_data.append(data)
# 将所有训练数据文件合并成一个 DataFrame
train_data = pd.concat(train_data)
# --- 3. 加载和合并测试数据 ---
test_data = []
for file_name in test_file_list:
# 读取单个测试数据文件 (CSV)
data = pd.read_csv(os.path.join(file_path, file_name))
test_data.append(data)
# 将所有测试数据文件合并成一个 DataFrame
test_data = pd.concat(test_data)
# --- 4. 处理数据并转换为 PyG Data 对象 ---
all_data = []
# 依次处理训练数据和测试数据
for data in [train_data, test_data]:
node_index = {} # 字典:用于将节点名称(序列)映射到图中的唯一整数索引
num_nodes = 0 # 图中节点的总数
edge_list = [] # 边的列表,存储为 (node_idx_1, node_idx_2) 元组
X = [] # 节点的特征矩阵列表(存储嵌入向量)
y_list = [] # 边的标签列表(存储 'Label')
# 遍历 DataFrame 中的每一行,每一行代表一个相互作用(即图中的一条边)
for _, row in data.iterrows():
label = float(row["Label"])
# 相互作用的两个节点
nodes = [row["Epitope"], row["CDR3.beta"]]
# 遍历两个节点,为它们分配唯一的索引并收集嵌入
for node in nodes:
if node not in node_index:
# 如果节点是新发现的,分配一个新索引
node_index[node] = num_nodes
num_nodes += 1
# 从嵌入字典中获取该节点的嵌入向量,并加入特征列表 X
X.append(embedding_dict[node])
# 收集该相互作用(边)的标签
y_list.append(label)
# 记录这条边(使用节点的整数索引)
edge_list.append((node_index[nodes[0]], node_index[nodes[1]]))
# 将特征列表 X 转换为 PyTorch tensor
X = torch.tensor(np.array(X), dtype=torch.float)
# 将边列表转换为 PyTorch Geometric 所需的格式 (2 x num_edges)
edge_index = torch.tensor(edge_list, dtype=torch.long).t().contiguous()
# 将标签列表转换为 PyTorch tensor
y = torch.tensor(y_list, dtype=torch.float)
# 创建 PyTorch Geometric 的 Data 对象,存储图的结构和特征
all_data.append(Data(x=X, edge_index=edge_index, y=y, num_nodes=num_nodes))
return all_data返回的列表包含两个data对象[Train_Graph,Test_Graph]
每个data封装了关键属性
data.x: 节点特征矩阵。图上所有不重复的 Epitope 和 CDR3.beta 序列的嵌入向量(N,f)
data.dege_index: 边连接矩阵。表示图中的所有相互作用(边)。存储了源节点 ID 到 目标节点 ID 的映射(2,E)
data.y: 边标签向量。与 edge_index 中的每条边一一对应,表示该相互作用的标签(0 或 1)(E)
data.num_nodes: 节点总数。图中不重复序列的总数量 N N N
处理步骤:
-
原始的csv文件输入,假如:

-
序列嵌入字典
每个节点都对应一个自己独有的向量,假如:
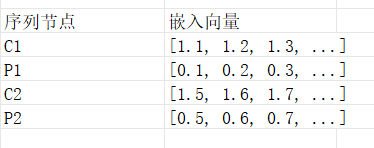
data.x=嵌入向量组合
-
首先给节点分配一个数字C1=0,P1=1,C2=2,P2=3
处理前:(C1,P1)(C2,P2) (C2,P1)
处理后:

data.edge_index=[0 2 2 / 1 3 1]
-
标签关系 1 0 1
-
Train_Graph = Data ( x = 张量 ( N × F ) , / / 所有序列的身份卡 edge_index = 张量 ( 2 × E ) , / / 相互作用的连接地图 y = 张量 ( E ) / / 相互作用的标签 ) \text{Train\_Graph} = \text{Data}( \text{x}=\text{张量}(N \times F), \quad // \text{所有序列的身份卡} \\ \text{edge\_index}=\text{张量}(2 \times E), \quad // \text{相互作用的连接地图} \\ \text{y}=\text{张量}(E) \quad // \text{相互作用的标签} \\ ) Train_Graph=Data(x=张量(N×F),//所有序列的身份卡edge_index=张量(2×E),//相互作用的连接地图y=张量(E)//相互作用的标签)
2.3.2 create_embeding.py
细读data_processing.py发现给出的文件没有embeddings.pkl嵌入文件也没有配置yaml文件,并且还没有arg_parser.py参数文件。
处理生成embeddings.pkl嵌入文件步骤:
-
首先安装esm库
pythonpip install fair-esm -
生成嵌入向量库create_embeding.py
pythonimport torch import esm import pandas as pd import numpy as np import pickle import os from tqdm import tqdm # --- 1. 配置参数 --- # 替换为您的实际文件路径 (假设文件位于 'dataset' 文件夹) TRAIN_FILE = "dataset/train.csv" VALID_FILE = "dataset/valid.csv" OUTPUT_DIR = "models" OUTPUT_PKL_PATH = os.path.join(OUTPUT_DIR, "my_project_embeddings.pkl") # --- 2. 序列提取与准备 --- def get_unique_sequences(train_path, valid_path): """提取所有不重复的 Epitope 和 CDR3.beta 序列,并返回 ESM 要求的格式。""" print("-> 正在读取并提取所有独特的序列...") train_df = pd.read_csv(train_path) valid_df = pd.read_csv(valid_path) all_sequences = set() # 提取所有独特的序列 all_sequences.update(train_df['Epitope'].astype(str).unique()) all_sequences.update(valid_df['Epitope'].astype(str).unique()) all_sequences.update(train_df['CDR3.beta'].astype(str).unique()) all_sequences.update(valid_df['CDR3.beta'].astype(str).unique()) # 清理和过滤掉无效/缺失的序列 unique_sequences = {seq for seq in all_sequences if seq and seq != 'nan'} print(f"-> 共找到 {len(unique_sequences)} 个独特的序列。") # 将序列转换为 ESM 批量编码所需的格式: [("name1", "SEQ1"), ("name2", "SEQ2"), ...] # 我们使用序列本身作为 'name' esm_data_input = [(seq, seq) for seq in unique_sequences] return esm_data_input, unique_sequences # --- 3. 嵌入生成与保存函数 --- def generate_and_save_embeddings(esm_data_input, unique_sequences): # --- A. 加载模型 --- print("-> 正在加载 ESM-2 650M 模型...") # 使用您代码中指定的强大模型 model, alphabet = esm.pretrained.esm2_t33_650M_UR50D() batch_converter = alphabet.get_batch_converter() model.eval() # 自动选择 CUDA 或 CPU device = torch.device("cuda" if torch.cuda.is_available() else "cpu") print(f"-> 正在使用设备: {device}") model = model.to(device) # --- B. 批量编码 --- # ESM 编码器一次处理所有序列可能导致内存溢出。 # 我们按批次(Batch)处理,这里设置一个合理的批次大小 BATCH_SIZE = 128 embedding_dict = {} # 启用 tqdm 进度条,并按批次进行处理 for i in tqdm(range(0, len(esm_data_input), BATCH_SIZE), desc="生成嵌入批次"): batch_data = esm_data_input[i:i + BATCH_SIZE] # 1. 转换数据为 tokens batch_labels, batch_strs, batch_tokens = batch_converter(batch_data) batch_lens = (batch_tokens != alphabet.padding_idx).sum(1) batch_tokens = batch_tokens.to(device) # 将 token 移动到 GPU # 2. 提取残基表示 (Per-residue representations) with torch.no_grad(): # repr_layers=[33] 确保我们使用第33层(即最后一层)的表示 results = model(batch_tokens, repr_layers=[33], return_contacts=False) # 获取第33层的 tokens 嵌入 token_representations = results["representations"][33].cpu() # 移回 CPU 处理 # 3. 核心步骤:生成序列级表示 (Per-sequence representations) # 通过对残基嵌入求平均,将可变长度的序列转换为固定长度的向量 for j, tokens_len in enumerate(batch_lens): # NOTE: token 0 是 BOS (序列开始) token,最后一个 token 是 EOS (序列结束) token # 我们对从 token 1 到 tokens_len - 1 的残基嵌入求平均 # (tokens_len.item() - 1) 是 EOS 的位置 sequence_embedding = token_representations[j, 1: tokens_len.item() - 1].mean(0).numpy() # 找到对应的原始序列字符串 original_seq = batch_data[j][1] # 保存到字典 embedding_dict[original_seq] = sequence_embedding # --- C. 保存文件 --- os.makedirs(OUTPUT_DIR, exist_ok=True) with open(OUTPUT_PKL_PATH, 'wb') as f: pickle.dump(embedding_dict, f) # 4. 验证保存结果 embedding_dim = embedding_dict[list(embedding_dict.keys())[0]].shape[0] print("\n==============================================") print(f"✅ 嵌入生成成功!文件已保存到: {OUTPUT_PKL_PATH}") print(f"✅ 字典中包含 {len(embedding_dict)} 个序列的嵌入。") print(f"✅ 每个嵌入向量的维度 (F) 为: {embedding_dim}") print("==============================================") # --- 4. 主程序执行 --- if __name__ == "__main__": esm_data_input, unique_sequences = get_unique_sequences(TRAIN_FILE, VALID_FILE) generate_and_save_embeddings(esm_data_input, unique_sequences) -
检查是否生成
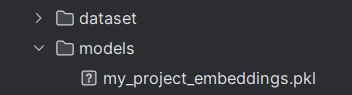
2.3.3 配置yaml文件
yaml
# ----------------------------------------------------------------------
# 全局配置 (Global Configuration)
# ----------------------------------------------------------------------
# 项目数据集名称,便于识别
dataset_name: MyDataSets
# 数据文件所在的根目录。
# 假设您的 train.csv 和 valid.csv 文件位于项目根目录下的 'dataset' 文件夹中。
path: dataset
# 模型保存路径。请根据您的需要修改文件名。
save_model: results/MyProject_FinalModel.pth
# 预计算嵌入文件的路径。
# 必须指向您上一步使用 ESM-2 生成的那个文件。
embeddings_path: models/my_project_embeddings.pkl
# ----------------------------------------------------------------------
# 默认划分配置 (Default Split Configuration)
# ----------------------------------------------------------------------
#
# 这是一个单一的、固定的划分方式,用于读取 train.csv 和 valid.csv
# 在您的程序中,您需要将 'default_split' 作为参数传递给数据加载函数。
#
default_split:
train_data:
# 训练集文件列表,只有一个 train.csv
file_list:
- train.csv
test_data:
# 测试集文件列表,使用 valid.csv 作为验证/测试集
file_list:
- valid.csv3、项目设计model
11月1日写了一点,哈哈今天是11月30日又开始了。
3.1、生成嵌入脚本create_embeding.py
使用上面的脚本生成train.csv和val.csv的嵌入pkl文件。如下图这样的。
3.2、加载数据和构件图脚本data_loader.py
我们修改上面的加载方式,修改成单纯的加载构建模块,把配置模块拆分出来。
3.2.1 加载已经生成的嵌入文件
python
def load_embeddings(embeddings_path):
"""
从pickle文件加载预先计算的序列嵌入。
Args:
embeddings_path: 嵌入pickle文件的路径
Returns:
dict: 字典将序列映射到嵌入向量
"""
with open(embeddings_path, 'rb') as f:
embeddings = pickle.load(f)
return embeddings3.2.2 根据嵌入文件从csv文件中构建图
python
def build_graph_from_csv(csv_path, embeddings_dict):
"""
从CSV文件中构建图
CSV format: columns=['Epitope', 'CDR3.beta', 'Label']
Args:
csv_path: CSV文件路径
embeddings_dict: 字典嵌入序列
Returns:
torch_geometric.data.Data: 图的数据格式
"""
# Read CSV
data = pd.read_csv(csv_path)
# 初始化图组件
node_index = {} # sequence -> node_id
num_nodes = 0
edge_list = []
X = [] # Node features
y_list = [] # Edge labels
# 构件图
for _, row in data.iterrows():
label = float(row['Label'])
sequences = [row['Epitope'], row['CDR3.beta']]
# 创建不存在的节点
for seq in sequences:
if seq not in node_index:
if seq not in embeddings_dict:
print(f"Warning: Sequence '{seq}' not found in embeddings, skipping...")
break
node_index[seq] = num_nodes
num_nodes += 1
X.append(embeddings_dict[seq])
else:
# 当两个节点创建时,创建边
y_list.append(label)
edge_list.append((node_index[sequences[0]], node_index[sequences[1]]))
# 转换张量
X = torch.tensor(np.array(X), dtype=torch.float)
edge_index = torch.tensor(edge_list, dtype=torch.long).t().contiguous()
y = torch.tensor(y_list, dtype=torch.float)
return Data(x=X, edge_index=edge_index, y=y, num_nodes=num_nodes)3.2.3 加载数据
python
def load_data(train_path, valid_path, embeddings_path):
"""
加载数据
Args:
train_path: Path to training CSV
valid_path: Path to validation CSV
embeddings_path: Path to embeddings file
Returns:
tuple: (train_data, valid_data) 作为PyG数据对象
"""
print("加载嵌入序列...")
embeddings = load_embeddings(embeddings_path)
print(f"加载 {len(embeddings)} 嵌入序列")
print("构建训练图...")
train_data = build_graph_from_csv(train_path, embeddings)
print(f"train: {train_data.num_nodes} nodes, {train_data.num_edges} edges")
print("构建验证图...")
valid_data = build_graph_from_csv(valid_path, embeddings)
print(f"Valid: {valid_data.num_nodes} nodes, {valid_data.num_edges} edges")
return train_data, valid_data3.3、配置模块config.py
需要的一些路径,模型参数单独放在一起,训练、测试时引入
python
"""
配置文件
"""
# 数据路径
TRAIN_DATA_PATH = 'dataset/train.csv'
VALID_DATA_PATH = 'dataset/valid.csv'
MODEL_SAVE_PATH = 'dataset/best_model.pth'
EMBEDDINGS_PATH = 'dataset/my_project_embeddings.pkl' # 嵌入文件
# 模型参数
EMBEDDING_DIM = 320 # 序列嵌入维度
HIDDEN_DIM = 128 # 隐藏层尺寸
MLP_HIDDEN_DIM = 256 # MLP隐藏维度
DROPOUT_RATE = 0.1 # Dropout
# 训练参数
LEARNING_RATE = 0.001
EPOCHS = 100
BATCH_SIZE = 1 # 图形级批处理
WEIGHT_DECAY = 5e-4
# 损失权重
BCE_WEIGHT = 1.0 # 二元交叉熵损失权重
AUC_WEIGHT = 0.0 # AUC损失
# 不平衡处理
POSITIVE_WEIGHT = 1.0 # 正类权重的乘数
# 设置
DEVICE = 'cuda'
GPU_ID = 0
# 随机种子
RANDOM_SEED = 42
# 评估
SAVE_BEST_MODEL = True # 是否保存3.4、最重要的模型model.py
3.4、训练脚本train.py
3.5、预测脚本predict.py
3.6、配置测试集预测脚本main.py
4、测试结果