决策曲线分析(Decision Curve Analysis, DCA)是一种用于评估、比较和优化诊断试验、预测模型或分子标志物临床实用性的统计方法。它由Andrew J. Vickers和Eugene B. Elkin于2006年提出,旨在弥补传统统计指标(如灵敏度、特异度、ROC曲线下面积)在评估模型临床价值方面的不足。
在既往文章,本人介绍了我的ggscidca包,可以用于逻辑回归,生存分析等各种决策曲线绘制,但是据我所知,目前还没有R包能支持复杂加权数据(nhanes数据)生存分析决策曲线,在上篇文章《R语言绘制复杂加权数据(nhanes数据)生存分析决策曲线》我已经介绍了R语言绘制复杂加权数据(nhanes数据)生存分析决策曲线,目前我继续升级了ggscidca包,使得它能进一步支持nhanes多模型生存分析决策曲线,
下面我来演示一下,先导入数据和R包
r
library(survey)
library(ggscidca)
library(scinhanes)
bc<-read.csv("E:/r/test/nahnesme.csv",sep=',',header=TRUE)
bc <- na.omit(bc)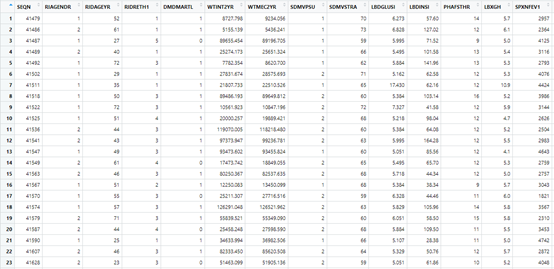
我介绍一下数据,SEQN:序列号,RIAGENDR, # 性别, RIDAGEYR, # 年龄,RIDRETH1, # 种族,DMDMARTL, # 婚姻状况,WTINT2YR,WTMEC2YR, # 权重,SDMVPSU, # psu,SDMVSTRA,# strata,LBDGLUSI, #血糖mmol表示,LBDINSI, #胰岛素( pmmol/L),PHAFSTHR #餐后血糖,LBXGH #糖化血红蛋白,SPXNFEV1, #FEV1:第一秒用力呼气量,SPXNFVC #FVC:用力肺活量,ml(估计肺容量),LBDGLTSI #餐后2小时血糖,factor.FVC是我把肺活量分为了2分类,方便用于测试。
把分类变量转成因子
r
bc$DMDMARTL<-ifelse(bc$DMDMARTL==1,1,0)
bc$RIAGENDR<-as.factor(bc$RIAGENDR)
bc$RIDRETH1<-as.factor(bc$RIDRETH1)
bc$DMDMARTL<-as.factor(bc$DMDMARTL)
bc$oGTT2<-as.factor(bc$oGTT2)如果做预测模型要拆分成建模集和验证集。我这里拆分一下
r
set.seed(123)
tr1<- sample(nrow(bc),0.7*nrow(bc))##随机无放抽取
bc_train <- bc[tr1,]#70%数据集
bc_test<- bc[-tr1,]#30%数据集建立抽样调查函数
r
bcSvy2<- svydesign(ids = ~ SDMVPSU, strata = ~ SDMVSTRA, weights = ~ WTMEC2YR,
nest=TRUE,data = bc_train)建立生存分析模型
r
svyfit <- svycoxph(Surv(time, factor.FVC) ~ RIDAGEYR+RIAGENDR+LBDINSI+RIDRETH1, x = TRUE,
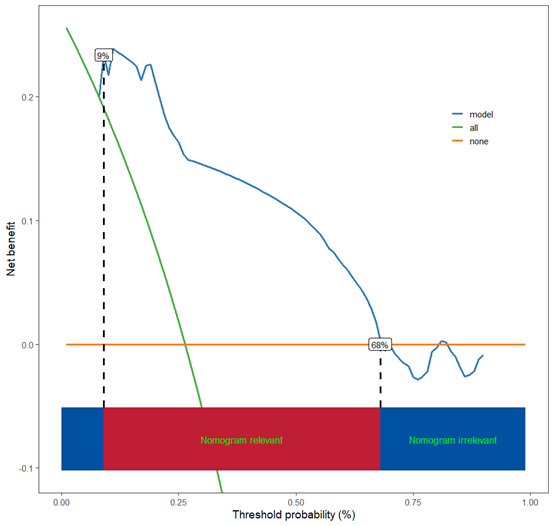
design = bcSvy2)复习一下单个决策曲线模型的内容,绘制复杂加权数据(nhanes数据)决策曲线一共需要2步:
- 第一步,把模型变成ggscidca包能够识别的类型
r
fit<-scisvycoxphmodel(svyfit,username=username,token=token)- 绘制决策曲线
r
scidca(fit)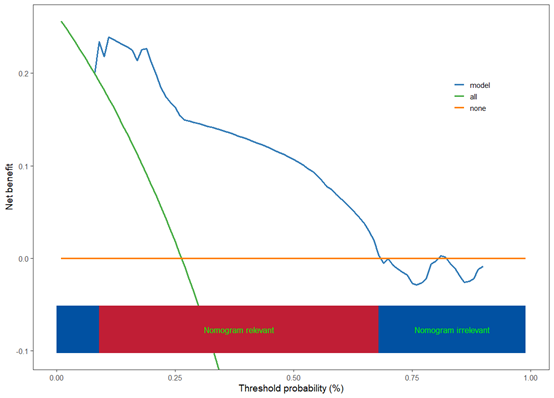
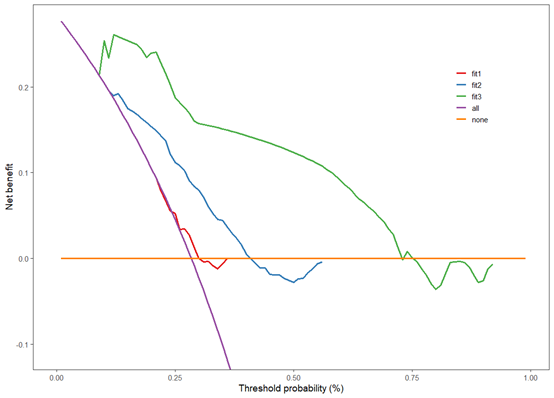
绘制多模型的决策曲线其实就是复制单个决策曲线的过程,由两种情况:
- 就是同一个模型多个时间点的
r
fit1<-scisvycoxphmodel(fit=svyfit,timepoint=300,username=username,token=token)
fit2<-scisvycoxphmodel(fit=svyfit,timepoint=500,username=username,token=token)
fit3<-scisvycoxphmodel(fit=svyfit,timepoint=700,username=username,token=token)制作数据列表的绘图,如果你既往使用过ggscidca包,是一模一样的,上手0困难
r
newdat<-list(bc_train,bc_train,bc_train)
cox.tcdca(fit1,fit2,fit3,newdata=newdat,timepoint=c(300,500,700))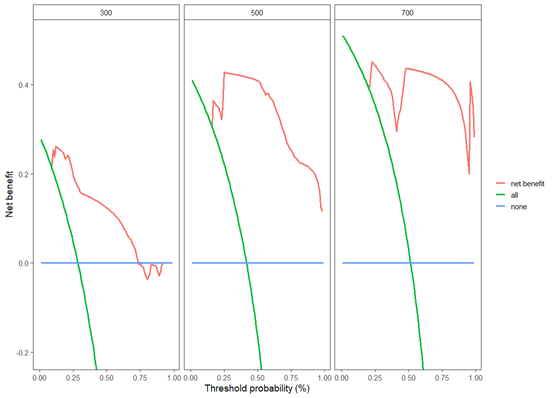
- 第二个情况就是多个模型同一个时间点的
###多个模型一个时间点
r
svyfit1 <- svycoxph(Surv(time, factor.FVC) ~ RIDAGEYR, x = TRUE,
design = bcSvy2)
svyfit2 <- svycoxph(Surv(time, factor.FVC) ~ RIDAGEYR+RIAGENDR, x = TRUE,
design = bcSvy2)
svyfit3 <- svycoxph(Surv(time, factor.FVC) ~ RIDAGEYR+RIAGENDR+LBDINSI+RIDRETH1, x = TRUE,
design = bcSvy2)因为模型不同,都要转换一遍
r
###转换模型
fit1<-scisvycoxphmodel(svyfit1,username=username,token=token)
fit2<-scisvycoxphmodel(svyfit2,username=username,token=token)
fit3<-scisvycoxphmodel(svyfit3,username=username,token=token)绘图
r
newdat<-list(bc_train,bc_train,bc_train)
cox.tcdca(fit1,fit2,fit3,newdata=newdat,timepoint=c(300))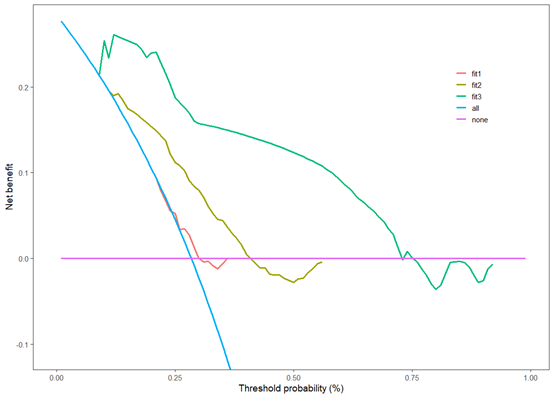
支持自定义颜色
r
cox.tcdca(fit1,fit2,fit3,newdata=newdat,timepoint=c(300),lincol =c("#E41A1C", "#377EB8", "#4DAF4A", "#984EA3", "#FF7F00"))