本章将重点介绍监督学习的实施------分类法。
分类技术或模型试图从观察值中得出某种结论。在分类问题中,我们有分类输出,如黑或白,或教学与非教学。在构建分类模型时,我们需要包含数据点和相应标签的训练数据集。例如,如果我们想检查图像是否是汽车。为了验证这一点,我们将构建一个训练数据集,包含与 car 相关的两个类别和无 car 的类别。然后我们需要利用训练样本来训练模型。分类模型主要用于人脸识别、垃圾邮件识别等领域。
在 Python 中构建分类器的步骤
在构建Python分类器时,我们将使用Python 3和Scikit-learn,这是一种机器学习工具。按照以下步骤构建 Python 分类器 −
Step 1 − 导入 Scikit-learn
这将是构建Python分类器的第一步。在这一步中,我们将安装一个名为Scikit-learn的Python包,它是Python中最好的机器学习模块之一。以下命令将帮助我们导入包 −
Import Sklearn步骤2 − 导入Scikit-learns数据集
在这一步中,我们可以开始处理机器学习模型的数据集。这里,我们将使用威斯康星乳腺癌诊断数据库。该数据集包含了关于乳腺癌肿瘤的各种信息,以及恶性 或良性的分类标签。该数据集包含569个实例或数据,涵盖569个肿瘤,包含30个属性或特征的信息,如肿瘤半径、纹理、光滑度和面积。借助以下命令,我们可以导入Scikit-learns乳腺癌数据集------
from sklearn.datasets import load_breast_cancer接下来,以下命令将加载数据集。
data = load_breast_cancer()以下是重要词典键列表 −
- 分类标签名称(target_names)
- 实际的标签(目标)
- 属性/特征名称(feature_names)
- 属性(数据)
现在,借助以下命令,我们可以为每个重要信息组创建新变量并分配数据。换句话说,我们可以通过以下命令组织数据 −
label_names = data['target_names']
labels = data['target']
feature_names = data['feature_names']
features = data['data']现在,为了更清晰,我们可以通过以下命令 − 打印类标签、第一个数据实例标签、特征名称和特征值
print(label_names)上述命令将分别打印恶性和良性类名。它以下方的输出 − 表示
['malignant' 'benign']现在,下面的命令会显示它们映射到二进制值 0 和 1。这里0代表恶性癌症,1代表良性癌症。您将获得以下输出 −
print(labels[0])
0下面给出的两个命令将生成特征名称和特征值。
print(feature_names[0])
mean radius
print(features[0])
[ 1.79900000e+01 1.03800000e+01 1.22800000e+02 1.00100000e+03
1.18400000e-01 2.77600000e-01 3.00100000e-01 1.47100000e-01
2.41900000e-01 7.87100000e-02 1.09500000e+00 9.05300000e-01
8.58900000e+00 1.53400000e+02 6.39900000e-03 4.90400000e-02
5.37300000e-02 1.58700000e-02 3.00300000e-02 6.19300000e-03
2.53800000e+01 1.73300000e+01 1.84600000e+02 2.01900000e+03
1.62200000e-01 6.65600000e-01 7.11900000e-01 2.65400000e-01
4.60100000e-01 1.18900000e-01]从上述输出中,我们可以看到第一个数据实例是一个恶性肿瘤,其半径为1.7990000e+01。
步骤3 − 将数据组织为集合
在这一步中,我们将数据分为两个部分,即训练集和测试集。将数据拆分到这些集合非常重要,因为我们必须在未见数据上测试模型。为了将数据拆分为集合,sklearn 有一个叫做 train_test_split() 函数的函数。借助以下命令,我们可以将数据拆分为以下集合 −
from sklearn.model_selection import train_test_split上述命令会导入 sklearn 的 train_test_split 函数,下面的命令将数据拆分为训练数据和测试数据。在下面的示例中,我们使用了40%的数据进行测试,剩余数据用于训练模型。
python
train, test, train_labels, test_labels = train_test_split(features,labels,test_size = 0.40, random_state = 42)步骤4 − 构建模型
在这一步中,我们将构建我们的模型。我们将使用纳夫贝叶斯算法来构建模型。以下命令可用于构建模型 −
from sklearn.naive_bayes import GaussianNB上述命令将导入GaussianNB模块。接下来的命令将帮助你初始化模型。
gnb = GaussianNB()我们将通过使用 gnb.fit() 来拟合模型来训练模型。
python
model = gnb.fit(train, train_labels)步骤5 − 评估模型及其准确性
在这一步中,我们将通过对测试数据进行预测来评估模型。然后我们也能查明它的准确性。在进行预测时,我们将使用 predict() 函数。以下命令将帮助你实现这一点------
preds = gnb.predict(test)
print(preds)
[1 0 0 1 1 0 0 0 1 1 1 0 1 0 1 0 1 1 1 0 1 1 0 1 1 1 1 1 1
0 1 1 1 1 1 1 0 1 0 1 1 0 1 1 1 1 1 1 1 1 0 0 1 1 1 1 1 0
0 1 1 0 0 1 1 1 0 0 1 1 0 0 1 0 1 1 1 1 1 1 0 1 1 0 0 0 0
0 1 1 1 1 1 1 1 1 0 0 1 0 0 1 0 0 1 1 1 0 1 1 0 1 1 0 0 0
1 1 1 0 0 1 1 0 1 0 0 1 1 0 0 0 1 1 1 0 1 1 0 0 1 0 1 1 0
1 0 0 1 1 1 1 1 1 1 0 0 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 0
1 1 0 1 1 1 1 1 1 0 0 0 1 1 0 1 0 1 1 1 1 0 1 1 0 1 1 1 0
1 0 0 1 1 1 1 1 1 1 1 0 1 1 1 1 1 0 1 0 0 1 1 0 1]上述0和1系列是肿瘤类别恶性和良性预测值。
现在,通过比较两个阵组,即test_labels 和预热, 我们可以确定模型的准确性。我们将使用**accuracy_score()**函数来确定准确率。考虑以下命令 −
from sklearn.metrics import accuracy_score
print(accuracy_score(test_labels,preds))
0.951754385965结果显示NaveBayes分类器的准确率为95.17%。
通过上述步骤,我们可以在 Python 中构建分类器。
在 Python 中构建分类器
在本节中,我们将学习如何在 Python 中构建分类器。
朴素贝叶斯分类器
Nave Bayes是一种基于贝叶斯定理构建分类器的分类技术。假设预测变量是独立的。简单来说,它假设某一类中某个特征的存在与其他特征的存在无关。为了构建Nave Bayes分类器,我们需要使用名为scikit learn的Python库。在 scikit learn 包下,Nave Bayes 模型有三种类型,分别是高斯模型、多项模型和伯努利模型。
要构建Nave Bayes机器学习分类器模型,我们需要以下减号
数据集
我们将使用名为乳腺癌威斯康星诊断数据库的数据集。该数据集包括关于乳腺癌肿瘤的各种信息,以及恶性或良性的分类标签。数据集包含569个肿瘤的569个实例或数据,并包含30个属性或特征的信息,例如肿瘤半径、纹理、平滑度和面积。我们可以从sklearn包导入此数据集。
贝叶斯模型
对于建筑Nave Bayes分类器,我们需要一个Nave Bayes模型。如前所述,在 scikit learn 包下有三种类型的 Nave Bayes 模型,分别是高斯模型、多项模型 和伯努利模型。在下面的例子中,我们将使用高斯纳夫贝叶斯模型。
利用上述方法,我们将构建一个Nave Bayes机器学习模型,利用肿瘤信息预测肿瘤是恶性还是良性。
首先,我们需要安装 sklearn 模块。可以通过以下命令 − 完成
Import Sklearn现在,我们需要导入名为"威斯康星乳腺癌诊断数据库"的数据集。
python
from sklearn.datasets import load_breast_cancer接下来,以下命令将加载数据集。
data = load_breast_cancer()数据可以组织如下 −
python
label_names = data['target_names']
labels = data['target']
feature_names = data['feature_names']
features = data['data']现在,为了更清晰,我们可以打印类标签、第一个数据实例标签、特征名称和特征值,并借助以下命令 −
print(label_names)上述命令将分别打印恶性和良性类名。它以下方的输出 − 表示
['malignant' 'benign']现在,下面的命令会显示它们映射到二进制值0和1。这里0代表恶性癌症,1代表良性癌症。它以下方的输出 − 表示
print(labels[0])
0以下两个命令将生成特征名称和特征值。
print(feature_names[0])
mean radius
print(features[0])
[ 1.79900000e+01 1.03800000e+01 1.22800000e+02 1.00100000e+03
1.18400000e-01 2.77600000e-01 3.00100000e-01 1.47100000e-01
2.41900000e-01 7.87100000e-02 1.09500000e+00 9.05300000e-01
8.58900000e+00 1.53400000e+02 6.39900000e-03 4.90400000e-02
5.37300000e-02 1.58700000e-02 3.00300000e-02 6.19300000e-03
2.53800000e+01 1.73300000e+01 1.84600000e+02 2.01900000e+03
1.62200000e-01 6.65600000e-01 7.11900000e-01 2.65400000e-01
4.60100000e-01 1.18900000e-01]从上述输出中,我们可以看到第一个数据实例是一个恶性肿瘤,其主半径为1.7990000e+01。
为了在未见数据上测试模型,我们需要将数据拆分为训练数据和测试数据。可以通过以下代码 − 来实现
from sklearn.model_selection import train_test_split上述命令会导入 sklearn 的 train_test_split 函数,下面的命令将数据拆分为训练数据和测试数据。在下面的例子中,我们使用了40%的数据进行测试,挖掘数据将用于训练模型。
python
train, test, train_labels, test_labels =
train_test_split(features,labels,test_size = 0.40, random_state = 42)现在,我们用以下命令构建模型 −
from sklearn.naive_bayes import GaussianNB上述命令将导入GaussianNB模块。现在,按照下面给出的命令,我们需要初始化模型。
python
gnb = GaussianNB()我们将通过使用 gnb.fit() 拟合模型来训练模型。
python
model = gnb.fit(train, train_labels)现在,通过对测试数据进行预测来评估模型,可以如下作 −
python
preds = gnb.predict(test)
print(preds)
[1 0 0 1 1 0 0 0 1 1 1 0 1 0 1 0 1 1 1 0 1 1 0 1 1 1 1 1 1
0 1 1 1 1 1 1 0 1 0 1 1 0 1 1 1 1 1 1 1 1 0 0 1 1 1 1 1 0
0 1 1 0 0 1 1 1 0 0 1 1 0 0 1 0 1 1 1 1 1 1 0 1 1 0 0 0 0
0 1 1 1 1 1 1 1 1 0 0 1 0 0 1 0 0 1 1 1 0 1 1 0 1 1 0 0 0
1 1 1 0 0 1 1 0 1 0 0 1 1 0 0 0 1 1 1 0 1 1 0 0 1 0 1 1 0
1 0 0 1 1 1 1 1 1 1 0 0 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 0
1 1 0 1 1 1 1 1 1 0 0 0 1 1 0 1 0 1 1 1 1 0 1 1 0 1 1 1 0
1 0 0 1 1 1 1 1 1 1 1 0 1 1 1 1 1 0 1 0 0 1 1 0 1]上述0和1系列是肿瘤类别的预测值,即恶性 而且是无害的。
现在,通过比较两个阵组------test_labels 和掠食 阵列,我们可以得出 模型的准确性。我们将使用 accuracy_score() 函数来确定 准确性。考虑以下命令 −
from sklearn.metrics import accuracy_score
print(accuracy_score(test_labels,preds))
0.951754385965结果显示NaveBayes分类器的准确率为95.17%。
那是基于Nave Bayse Gaussian模型的机器学习分类器。
支持向量机(SVM)
基本上,支持向量机(SVM)是一种监督式机器学习算法,具有 可以用于回归和分类。SVM的主要概念是绘制每一个 数据项作为 n 维空间中的一点,每个特征的值为 某个特定坐标。这里 n 就是我们会拥有的特征。以下是 理解SVM的概念的简单图形表示 −
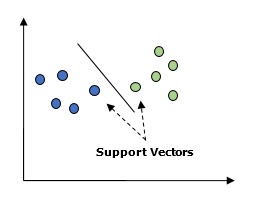
在上图中,我们有两个特征。因此,我们首先需要绘制这两个的图 二维空间中的变量,每个点有两个坐标,称为支撑 向量。该线将数据分为两个不同的分类组。这条线是 分类器。
在这里,我们将利用 scikit-learn 和 iris 数据集构建一个 SVM 分类器。Scikitlearn 库中包含 sklearn.svm 模块,并提供 sklearn.svm.svc 用于分类。 展示了基于4个特征预测鸢尾花品种的SVM分类器 如下。
数据集
我们将使用鸢尾花数据集,该数据集包含3个类别,每类50个实例,每个类别对应一种鸢尾花植物。每个实例都有四个特征,即萼片长度、萼片宽度、花瓣长度和花瓣宽度。下图展示了基于四个特征预测鸢尾花品种的SVM分类器。
内核
这是SVM使用的一种技术。基本上,这些函数将低维输入空间转换为更高维空间。它将不可分离问题转换为可分离问题。核函数可以是线性、多项式、rbf和S形函数之间的任意一个。在这个例子中,我们将使用线性核。
现在我们导入以下包 −
python
import pandas as pd
import numpy as np
from sklearn import svm, datasets
import matplotlib.pyplot as plt现在,加载输入数据−
python
iris = datasets.load_iris()我们取前两个特征−
python
X = iris.data[:, :2]
y = iris.target我们将用原始数据绘制支撑向量机器边界。我们正在创建一个网格来绘制图。
python
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
h = (x_max / x_min)/100
xx, yy = np.meshgrid(np.arange(x_min, x_max, h),
np.arange(y_min, y_max, h))
X_plot = np.c_[xx.ravel(), yy.ravel()]我们需要给出正则化参数的值。
python
C = 1.0我们需要创建SVM分类器对象。
python
Svc_classifier = svm_classifier.SVC(kernel='linear',
C=C, decision_function_shape = 'ovr').fit(X, y)
Z = svc_classifier.predict(X_plot)
Z = Z.reshape(xx.shape)
plt.figure(figsize = (15, 5))
plt.subplot(121)
plt.contourf(xx, yy, Z, cmap = plt.cm.tab10, alpha = 0.3)
plt.scatter(X[:, 0], X[:, 1], c = y, cmap = plt.cm.Set1)
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('SVC with linear kernel')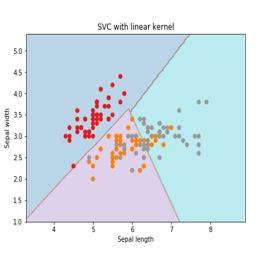
逻辑回归
基本上,逻辑回归模型是监督分类算法家族中的一个成员。逻辑回归通过使用逻辑函数估计概率来衡量因变量与自变量之间的关系。
这里,如果我们谈论因变量和自变量,那么因变量是我们要预测的目标类变量,另一端则是我们用来预测目标类的特征。
在逻辑回归中,估计概率意味着预测事件发生的可能性。例如,店主想预测进入店铺的顾客是否会购买PlayStation(例如)。顾客的性别、年龄等多种特征会被店主观察,以预测发生的可能性,比如是否购买PlayStation。逻辑函数是用来构建具有不同参数函数的S形曲线。
前提条件
在使用逻辑回归构建分类器之前,我们需要在系统上安装Tkinter包。它可以从 https://docs.python.org/2/library/tkinter.html 安装。
现在,借助下面给出的代码,我们可以利用逻辑回归 − 创建一个分类器
首先,我们将导入一些包------
python
import numpy as np
from sklearn import linear_model
import matplotlib.pyplot as plt现在,我们需要定义样本数据,具体如下 −
python
X = np.array([[2, 4.8], [2.9, 4.7], [2.5, 5], [3.2, 5.5], [6, 5], [7.6, 4],
[3.2, 0.9], [2.9, 1.9],[2.4, 3.5], [0.5, 3.4], [1, 4], [0.9, 5.9]])
y = np.array([0, 0, 0, 1, 1, 1, 2, 2, 2, 3, 3, 3])接下来,我们需要创建逻辑回归分类器,具体作如下 −
python
Classifier_LR = linear_model.LogisticRegression(solver = 'liblinear', C = 75)最后但同样重要的是,我们需要训练这个分类器------
python
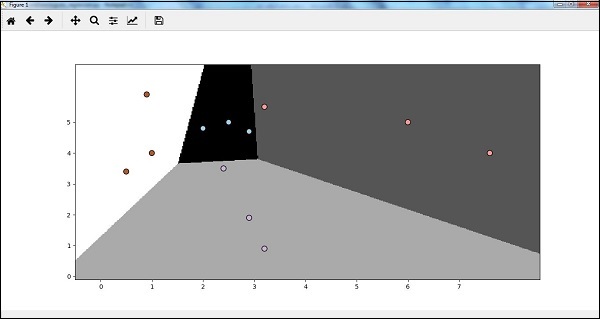
Classifier_LR.fit(X, y)那么,我们如何可视化输出呢?这可以通过创建一个名为 Logistic_visualize() − 的函数来实现
Def Logistic_visualize(Classifier_LR, X, y):
min_x, max_x = X[:, 0].min() - 1.0, X[:, 0].max() + 1.0
min_y, max_y = X[:, 1].min() - 1.0, X[:, 1].max() + 1.0在上面的那一行中,我们定义了网格中用于网格的最小值和最大值 X 和 Y。此外,我们还会定义网格绘制的步长。
python
mesh_step_size = 0.02让我们定义X和Y值的网格网格如下 −
python
x_vals, y_vals = np.meshgrid(np.arange(min_x, max_x, mesh_step_size),
np.arange(min_y, max_y, mesh_step_size))借助以下代码,我们可以在网格 − 上运行分类器
python
output = classifier.predict(np.c_[x_vals.ravel(), y_vals.ravel()])
output = output.reshape(x_vals.shape)
plt.figure()
plt.pcolormesh(x_vals, y_vals, output, cmap = plt.cm.gray)
plt.scatter(X[:, 0], X[:, 1], c = y, s = 75, edgecolors = 'black',
linewidth=1, cmap = plt.cm.Paired)以下代码行将指定该图的边界
python
plt.xlim(x_vals.min(), x_vals.max())
plt.ylim(y_vals.min(), y_vals.max())
plt.xticks((np.arange(int(X[:, 0].min() - 1), int(X[:, 0].max() + 1), 1.0)))
plt.yticks((np.arange(int(X[:, 1].min() - 1), int(X[:, 1].max() + 1), 1.0)))
plt.show()现在,运行代码后,我们将得到如下输出,逻辑回归分类器 −
决策树分类器
决策树基本上是一个二叉树流程图,每个节点根据某个特征变量划分一组观测值。
在这里,我们正在构建一个决策树分类器,用于预测男性或女性。我们将采用一个非常小的数据集,共19个样本。这些样本包括两个特征、身高和头发长度。
前提条件
为了构建以下分类器,我们需要安装pydotplus 和graphviz 。基本上,graphviz 是一个用点文件绘制图形的工具,pydotplus 是 Graphviz Dot 语言的一个模块。它可以通过包管理器或 PIP 安装。
现在,我们可以借助以下 Python 代码构建决策树分类器 −
首先,让我们导入一些重要的库,如下所示------
python
import pydotplus
from sklearn import tree
from sklearn.datasets import load_iris
from sklearn.metrics import classification_report
from sklearn import cross_validation
import collections现在,我们需要提供以下数据集 −
python
X = [[165,19],[175,32],[136,35],[174,65],[141,28],[176,15],[131,32],
[166,6],[128,32],[179,10],[136,34],[186,2],[126,25],[176,28],[112,38],
[169,9],[171,36],[116,25],[196,25]]
Y = ['Man','Woman','Woman','Man','Woman','Man','Woman','Man','Woman',
'Man','Woman','Man','Woman','Woman','Woman','Man','Woman','Woman','Man']
data_feature_names = ['height','length of hair']
X_train, X_test, Y_train, Y_test = cross_validation.train_test_split
(X, Y, test_size=0.40, random_state=5)提供数据集后,我们需要拟合模型,具体步骤如下 −
python
clf = tree.DecisionTreeClassifier()
clf = clf.fit(X,Y)预测可以通过以下 Python 代码进行 −
python
prediction = clf.predict([[133,37]])
print(prediction)我们可以借助以下 Python 代码可视化决策树 −
python
dot_data = tree.export_graphviz(clf,feature_names = data_feature_names,
out_file = None,filled = True,rounded = True)
graph = pydotplus.graph_from_dot_data(dot_data)
colors = ('orange', 'yellow')
edges = collections.defaultdict(list)
for edge in graph.get_edge_list():
edges[edge.get_source()].append(int(edge.get_destination()))
for edge in edges: edges[edge].sort()
for i in range(2):dest = graph.get_node(str(edges[edge][i]))[0]
dest.set_fillcolor(colors[i])
graph.write_png('Decisiontree16.png')它会给出上述代码的预测为**[Woman]**,并生成以下决策树−
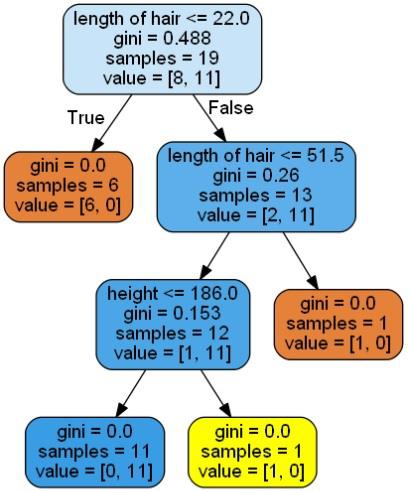
我们可以调整预测中特征的值来检验。
随机森林分类器
集成方法是将机器学习模型结合成一个更强大的机器学习模型的方法。随机森林是决策树的集合,是其中之一。它优于单一决策树,因为在保持预测能力的同时,它可以通过平均结果来减少过拟合。这里,我们将在scikit学习癌症数据集上实现随机森林模型。
导入必要的包 −
python
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
import matplotlib.pyplot as plt
import numpy as np现在,我们需要提供数据集,具体作如下和减号
python
cancer = load_breast_cancer()
X_train, X_test, y_train,
y_test = train_test_split(cancer.data, cancer.target, random_state = 0)提供数据集后,我们需要拟合模型,具体步骤如下 −
python
forest = RandomForestClassifier(n_estimators = 50, random_state = 0)
forest.fit(X_train,y_train)现在,获取训练和测试子集的准确性:如果我们增加估计量的数量,测试子集的准确率也会提高。
python
print('Accuracy on the training subset:(:.3f)',format(forest.score(X_train,y_train)))
print('Accuracy on the training subset:(:.3f)',format(forest.score(X_test,y_test)))输出
Accuracy on the training subset:(:.3f) 1.0
Accuracy on the training subset:(:.3f) 0.965034965034965现在,和决策树一样,随机森林有 feature_importance 模块,它能比决策树更好地展示特征权重。它可以绘制并可视化如下 −
python
n_features = cancer.data.shape[1]
plt.barh(range(n_features),forest.feature_importances_, align='center')
plt.yticks(np.arange(n_features),cancer.feature_names)
plt.xlabel('Feature Importance')
plt.ylabel('Feature')
plt.show()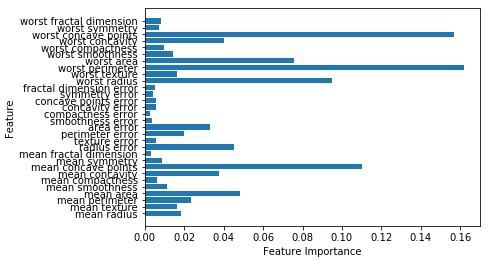
分类器的性能
在实现机器学习算法之后,我们需要了解模型的有效性,衡量有效性的标准可以基于数据集和度量。为了评估不同的机器学习算法,我们可以使用不同的性能指标。例如,假设使用一个分类器来区分不同对象的图像,我们可以使用分类性能指标,如平均准确度、AUC等。从某种意义上说,我们选择用来评估机器学习模型的度量是非常重要的,因为度量的选择会影响机器学习算法性能的度量和比较。以下是一些指标------------
混淆矩阵
基本上,它用于分类问题,其中输出可以是两种或更多类型的类。这是衡量分类器性能最简单的方法。混淆矩阵基本上是一个具有两个维度的表格,即实际和预测。这两个维度都有真阳性(TP)、真阴性(TN)、假阳性(FP)、假阴性(FN)。
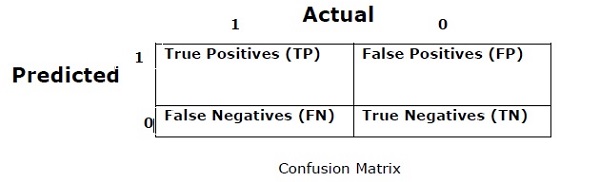
在上述混淆矩阵中,1代表正类,0代表负类。
以下是与混淆矩阵 − 相关的术语
-
**真阳性 −**TP是指实际数据点类别为1且预测值也是1的情况。
-
**真负 −**TN是指数据点的实际类别为0,而预测值也是0的情况。
-
**假阳性 −**FP是指实际数据点类别为0且预测值也是1的情况。
-
**假阴性 −**FNs是指数据点的实际类别为1,而预测值也是0的情况。
准确性
混淆矩阵本身并不是性能指标,但几乎所有性能矩阵都基于混淆矩阵。其中之一是准确性。在分类问题中,它可以定义为模型在所有类型预测中做出的正确预测数量。计算准确率的公式如下 −
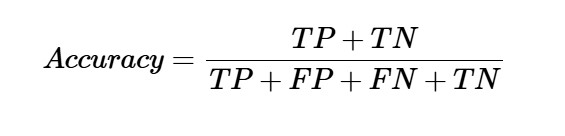
精度
它主要用于文档检索。它可以定义为有多少份返回的文件是正确的。以下是计算精度−的公式
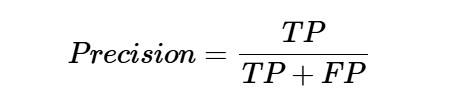
召回还是敏感
它可以定义为模型返回了多少正值。以下是计算模型回忆/灵敏度的公式 −
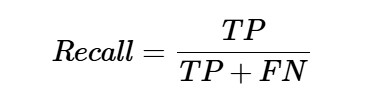
特异性
它可以定义为模型返回多少负值。回忆完全相反。以下是计算模型特异性的公式 −
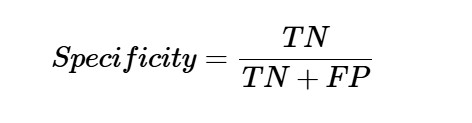
类别不平衡问题
类别不平衡是指某一类别的观测数量明显少于其他类别的情形。例如,在需要识别罕见病、银行欺诈交易等情景中,这个问题尤为突出。
不平衡类的例子
让我们以欺诈检测数据集为例,以理解不平衡类别 − 的概念
python
Total observations = 5000
Fraudulent Observations = 50
Non-Fraudulent Observations = 4950
Event Rate = 1%解决方案
平衡类可以解决不平衡类的问题。平衡阶级的主要目标是提高少数群体的频率,或降低多数阶级的频率。以下是解决不平衡类问题的方法 −
重新采样
重采样是一系列用于重建样本数据集的方法------包括训练集和测试集。重新采样是为了提高模型的准确性。以下是一些重采样技术------
- 随机欠采样 − 该技术旨在通过随机消除多数类例子来平衡类别分布。此过程持续进行,直到多数和少数阶级实例平衡。
python
Total observations = 5000
Fraudulent Observations = 50
Non-Fraudulent Observations = 4950
Event Rate = 1%在这种情况下,我们从非欺诈实例中提取10%且不替换的样本,然后将它们与欺诈实例合并------
随机样后非欺诈性观察 = 4950 = 495 的10%
将这些观测与虚假观测合并后的总观测值 = 50+495 = 545
因此,现在新数据集在采样不足后事件率 = 9%
该技术的主要优点是可以缩短运行时间并改善存储。但另一方面,它可能会丢弃有用信息,同时减少训练数据样本的数量。
- 随机过采样 − 该技术旨在通过复制少数类实例数量来平衡类别分布。
python
Total observations = 5000
Fraudulent Observations = 50
Non-Fraudulent Observations = 4950
Event Rate = 1%如果我们重复50个虚假观察30次,那么复制少数群体观察后,欺诈观察值将达到1500个。然后新数据中经过过采样后的总观测值将是4950+1500=6450。因此,新数据集的事件率将为1500/6450 = 23%。
这种方法的主要优点是不会丢失有用的信息。但另一方面,由于它复制了少数民族阶级的事件,它也增加了过度拟合的可能性。
集成技术
该方法主要用于修改现有的分类算法,使其适用于不平衡数据集。在这种方法中,我们从原始数据中构造几个两阶段分类器,然后对它们的预测进行聚合。随机森林分类器是集成分类器的一个例子。