👋 如果本文对你有帮助,欢迎点赞、收藏
这段时间在处理/对接 空间转录组(ST) 数据,也顺手把相关技术路线做了一次系统梳理。空间转录组看似"一个赛道",实际包含两条完全不同的技术范式:测序型 与 成像型。而不同平台在"分辨率---通量---样本类型---成本---下游分析复杂度"之间的取舍差异,往往决定了你最后能回答什么问题。
本文以"笔记型"的方式,把主流技术做一个全景对照学习,并重点对 Stereo-seq 的数据结构做拆解学习。
说明一下:这篇是"边学边记"并且和LLM对话的整理稿,不敢说面面俱到。若你发现哪里有疏漏,或者有更成熟的经验,欢迎随时指出,我们一起把这份笔记完善起来。
引言:空间转录组学的兴起与重要性
单细胞RNA测序(scRNA-seq)的出现让我们能够解析组织中各细胞类型的基因表达异质性,但其核心局限在于需要将组织解离成单细胞悬液,导致空间信息丢失[1]。也就是说,我们无法知道这些细胞在组织中的原始位置关系,这对于理解组织结构、细胞间相互作用和疾病微环境至关重要。空间转录组(Spatial Transcriptomics, ST)技术正是在这一背景下兴起:它旨在保留组织空间结构的同时,获取高通量基因表达信息,从而在二维(甚至三维)空间中描绘转录组图谱[2]。
空间转录组的重要性在近年来迅速凸显。Nature Methods将空间分辨转录组学评为"2020年度方法"之一,强调了这一领域的突破性意义[2]。通过在组织中测量基因表达的位置,研究者可以更深入地研究肿瘤微环境(如免疫细胞与肿瘤细胞的空间关系)、发育过程(胚胎细胞命运的空间轨迹)以及神经科学(神经元回路结构与基因表达)等重大问题[3][4]。
空间转录组技术的总体框架:测序型 vs 成像型
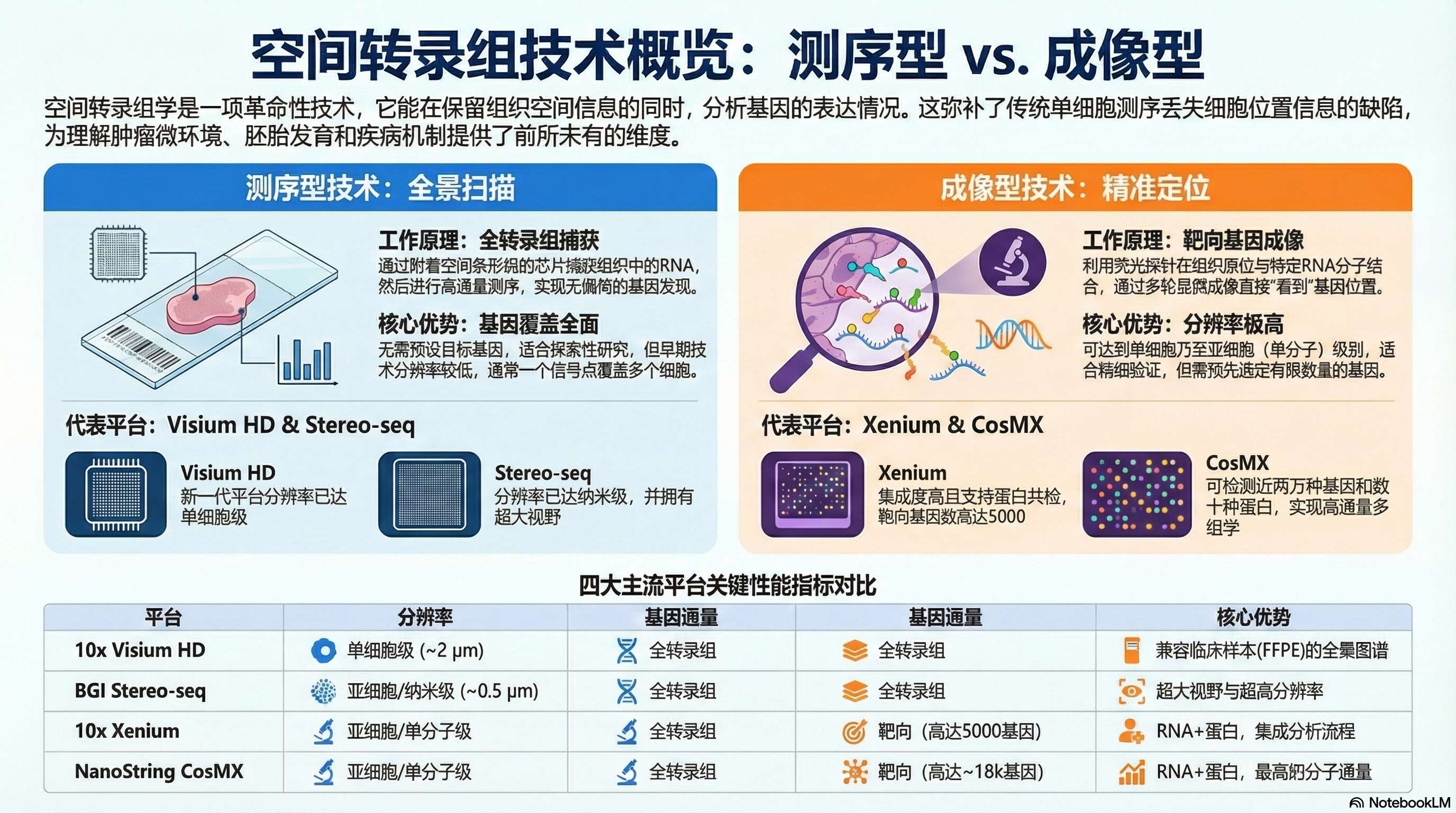
目前主流的空间转录组技术主要分为两大类:测序型(sequencing-based)和成像型(imaging-based)[5]。两类方法在原理和特征上各有区别:
- **测序型方法:**这类方法通过空间条形码标记RNAs的位置,然后将RNA提取后用高通量测序读出序列[5]。典型流程是将组织切片铺在带有位置编码寡核苷酸探针的载片上,使mRNA在原位与带有空间坐标条码的探针结合,然后进行反转录和文库构建,最后测序并将每条转录对应回其空间位置。
- **成像型方法:**这类方法基于原位核酸杂交和成像,通过荧光探针直接在组织切片中检测RNA分子的位置[9]。通常采用多轮次荧光原位杂交(FISH)策略:设计互补探针与目标mRNA结合,使用不同组合的荧光报告或者顺序编码,在显微镜下进行多轮成像,从而实现单分子、亚细胞分辨率的RNA定位。
值得注意的是,随着技术发展,两类方法正逐渐融合一些特性:例如新一代测序型平台提高了空间分辨率接近单细胞水平;成像型平台的可检测基因数量也在增加。不过总体来说,"测序型=全转录组、低到中等分辨率","成像型=高分辨率、靶向基因集合"这一框架仍是理解ST技术格局的基础。
测序型技术详解:主流平台与特点
10x Genomics Visium 系列
10x Genomics公司的Visium Spatial Gene Expression是目前应用最广泛的商业测序型空间转录组平台之一。Visium采用预制的玻片,上面印有约5000个位置固定的寡核苷酸斑点,每个斑点带有唯一空间条形码,用于捕获对应位置的mRNA[8]。在实验中,将新鲜冷冻的组织切片铺在Visium载片上,mRNA会从细胞扩散并被邻近的捕获探针(带poly(dT))结合,然后经过原位逆转录、文库制备和高通量测序,最后将测序得到的转录本通过条码定位回组织位置[5]。Visium每个捕获斑点直径约55µm,中心间距约100µm,大约覆盖1--10个细胞[7]。因此其空间分辨率在单细胞和组织结构层面之间:能够解析大型组织区域的基因表达模式,但单个斑点内可能混合了多个细胞的信号[7]。
值得一提的是,针对临床常用的FFPE(福尔马林固定石蜡包埋)样本,10x推出了Visium的探针捕获方案(Visium Spatial Gene Expression for FFPE)。该方案不依赖polyA捕获,而是采用探针panel直接杂交捕获目标转录本,设计涵盖约18,000个人类转录本的探针池,Visium可以在FFPE切片上进行空间转录分析,实现接近全转录组范围的检测[13]。
最新的发展是Visium HD(高分辨率版)的问世[14][15]。Visium HD采用了高密度寡核苷酸阵列,将捕获特征缩小到约2µm大小,并在原先6.5×6.5 mm2的区域内集成了约1100万个捕获点,相比原先的5000点密度提高数千倍。这使Visium HD能够达到单细胞甚至亚细胞级别的空间解析能力,同时支持FFPE切片,从而在临床转化中具有重要潜力[15][16]。
- **应用场景:**Visium标准版常用于组织结构清晰的样本(如肿瘤切片、大脑组织等)以获得全局基因表达概览。通过将空间表达数据与H&E染色图像对齐,能够发现在组织结构层面的空间差异基因表达区。适合在探索性研究中用于高通量筛查:先识别不同区域的基因表达差异,再辅以单细胞测序进行深入解析[18]。Visium HD由于具备单细胞精度和FFPE兼容性,适合需要精细空间分辨和临床样本分析的场景,例如肿瘤免疫微环境微区解析、病理结构相关的细胞互作研究等[15]。
BGI Stereo-seq(华大时空组学)
Stereo-seq(SpaTial Enhanced REsolution Omics-sequencing)是华大基因推出的新一代高分辨率空间转录组测序技术,代表了测序型空间组学在分辨率和覆盖面积上的重要突破[20][21]。Stereo-seq的核心是DNB(DNA nanoball)阵列芯片:在载片上规则铺设大量DNB纳米球,每个DNB携带一个唯一空间条形码(CID)。与Visium的spot捕获不同,Stereo-seq的DNB间距可达0.5µm,理论上实现亚细胞级空间分辨率[21]。实验中组织切片被贴附在DNB阵列上,通过原位反转录使mRNA转录成cDNA并捕获到邻近DNB上,形成带有空间条码的cDNA文库,后续测序后即可将表达信息映射回芯片坐标。
Stereo-seq的另一个优势是高覆盖面积:华大推出的芯片尺寸可达厘米级甚至更大(如1cm×1cm或更大区域),能够一次性测量大尺度组织甚至整个器官切片的空间转录图谱[21][22]。相较于许多高分辨率技术只能覆盖小范围,Stereo-seq在"高分辨率+大面积"这一组合上具有独特优势。
Stereo-seq V2版本进一步增强了对FFPE样本的支持[24],并在细胞分割、图像配准、数据处理流程等方面提供了配套软件(如SAW pipeline)[37]。此外,Stereo-seq也常结合ssDNA核染(类似DAPI/核酸染色)作为组织形态参考图像,以完成空间坐标配准和组织边界识别[24]。
- **应用场景:**Stereo-seq非常适用于需要高分辨率和大范围覆盖的研究,例如构建胚胎发育图谱、全器官空间表达地图、肿瘤微环境的全景解析等[21][23]。在肿瘤研究中,Stereo-seq可用于识别肿瘤边缘/核心区域不同细胞的空间分布、免疫细胞浸润模式、空间异质性等关键问题。其厘米级覆盖能力也使其适合整块组织切片分析,减少取样偏差。
Slide-seq 系列与HDST等研究型平台
除商业化平台外,学术界也发展了多种测序型空间转录组技术。Slide-seq(2019)提出利用随机铺设在玻片上的带条码微珠(直径约10µm)捕获RNA并测序,从而达到接近单细胞分辨率的空间转录组测量[38][39]。Slide-seq V2进一步提升捕获效率和灵敏度,使其能够在组织切片上得到更高质量的全转录组表达数据[40][41]。相比Visium,Slide-seq分辨率更高,但需要复杂的微珠定位和实验流程,尚未形成成熟商业产品。
HDST(High-definition spatial transcriptomics)则通过更小的微孔阵列(约2µm)或微珠实现更高分辨率,甚至达到亚细胞级别[42]。但由于捕获效率低、实验复杂度高,HDST更多用于方法学验证和极限分辨探索,目前应用相对较少。
成像型技术详解:Xenium、CosMX、MERSCOPE等
成像型空间转录组技术(也称原位空间转录组)依赖多轮FISH和高分辨显微成像,能够在单细胞/亚细胞级别定位RNA分子,并常结合细胞分割实现真正的"单细胞表达矩阵"。主流商业平台包括10x Xenium、NanoString CosMX和Vizgen MERSCOPE等[45][64]。
10x Genomics Xenium
Xenium In Situ平台是10x推出的原位成像型空间转录组平台,兼容新鲜和FFPE样本。Xenium使用探针panel(可定制50--5000基因)对目标RNA进行原位杂交,并通过多轮荧光成像实现每个RNA分子的空间定位[46][47]。Xenium还支持蛋白检测面板,使其具备"转录+蛋白"多组学能力[51]。平台配套Xenium Explorer软件用于细胞分割、表达矩阵生成和可视化分析[52]。
- **优势与应用:**Xenium具有高灵敏度和较高的基因通量,适合在肿瘤免疫微环境研究中分析细胞间相互作用、空间微区的细胞组成等[46]。特别是FFPE兼容性使其适合临床样本的大规模机制验证。
NanoString CosMX
CosMX平台同样基于原位杂交成像,兼容FFPE和新鲜样本。其最大特点是基因通量较高:通过Whole Transcriptome(WTA)模式可检测高达约18,000基因,同时支持蛋白检测(如19k RNA + 72蛋白)[54][55]。CosMX通过细胞分割获得单细胞表达矩阵,并可实现"same-cell multiomics"(同一细胞RNA+蛋白)分析[62]。
- **优势与应用:**CosMX适合需要高基因覆盖和多蛋白检测的研究,如复杂肿瘤微环境中多细胞类型的机制解析、临床转化验证等[55][57]。
Vizgen MERSCOPE(MERFISH)
MERSCOPE基于MERFISH(Multiplexed Error-Robust FISH)技术,通过编码探针和多轮成像实现高精度RNA定位。其panel通常为靶向约1000基因左右[67]。MERSCOPE在神经科学领域应用广泛,因其成像精度高、数据可靠性强[63]。MERSCOPE Ultra等升级版本进一步提升性能[63]。
- **优势与应用:**适合在需要亚细胞精度的细胞图谱研究中使用,如神经元亚区、细胞突起中RNA定位等精细结构解析。
华大Stereo-seq数据文件结构解析:普遍情况
在实际项目中,Stereo-seq交付数据通常包含表达矩阵(GEF或h5ad)、图像文件(核酸/核染参考图)、以及配套报告与可视化结果。大致文件含义与用途如下。
目录结构概览
以某个样本为例,交付目录通常包括:
analysis/:分析结果文件,常见为.h5ad(AnnData格式)以及marker特征表等。feature_expression/:表达矩阵文件(.gef等),包括原始、组织区域、cellbin等不同版本。bam/annotated_bam/:比对后的bam文件(dedup、target等),供进一步验证或自定义分析。image/:核酸染色图像(ssDNA)及其mask、配准、组织切割等图像文件。report.html:QC与分析流程报告。visualization.tar.gz:可视化交互文件或网页资源包。
analysis 文件夹:h5ad与marker_features
Stereo-seq交付的.h5ad文件通常包含不同分辨率或不同处理方式的表达矩阵与空间坐标信息:
*_bin20_1.0.h5ad/*_bin50_1.0.h5ad:binning后的空间表达矩阵。bin20表示将0.5µm DNB按20×20聚合成一个bin,形成约10µm的空间分辨率;bin50则约为25µm分辨率。binning提高信噪比、适合宏观区域分析和快速聚类。*_cellbin_1.0.h5ad:基于细胞分割的表达矩阵。cellbin通过核染图像进行细胞分割,为每个细胞生成表达矩阵(更接近"单细胞空间转录组")。该矩阵适合细胞层面的聚类、注释和细胞互作分析。*_cellbin_1.0.adjusted.h5ad:调整后的cellbin矩阵,可能包含背景校正、表达归一化或细胞边界修正后的结果(不同版本定义需参考交付说明或SAW pipeline输出)。
对应的*.marker_features.csv文件通常为每个聚类/区域计算的marker基因特征表,便于快速解读空间聚类结果。
feature_expression 文件夹:GEF表达矩阵
GEF(Gene Expression Format)是华大时空组学常用的表达数据格式,包含空间坐标、基因表达计数等信息。常见文件包括:
*_raw.gef:原始全片表达数据,未做组织区域筛选。*_tissue.gef:组织区域表达数据,通常已根据mask去除背景区域。*_cellbin.gef:细胞分割后的表达数据。*_adjusted.cellbin.gef:调整后的细胞分割表达数据。*_raw_barcode_gene_exp.txt:可读的barcode-基因表达文本格式,适合快速查看或导入其他分析软件(但通常体积较大)。
image 文件夹:ssDNA核酸/核染参考图像
Stereo-seq常使用ssDNA染色(单链DNA核酸染色)作为组织形态参考图,而不是传统H&E。原因在于ssDNA染色可以更突出细胞核结构,便于细胞分割和空间配准,同时与转录捕获芯片坐标对齐更直接。
常见图像包括:
*_ssDNA_regist.tif:配准后的核酸/核染参考图,已与芯片空间坐标或表达矩阵对齐,用于空间可视化叠加(如在stereopy/scanpy中将表达点映射回组织)。*_ssDNA_mask.tif:组织区域mask图像,标记组织与背景区域。*_ssDNA_mask_edm_dis_10.tif:mask的距离变换或扩展结果(用于更精细的组织边界处理或质量控制)。*_ssDNA_tissue_cut.tif:组织切割图像,可能为裁剪后的组织区域图像,用于可视化与报告。
配准后的核酸/核染参考图如何使用?
配准后的ssDNA_regist.tif通常用于:
- 表达矩阵叠加可视化 :将
.h5ad中的空间坐标与图像坐标对应,在Seurat、Scanpy、stereopy等工具中叠加表达点和组织形态。 - 细胞分割验证:结合cellbin输出的细胞边界,检查细胞分割是否合理。
- 跨模态融合:如果后续补做H&E或IHC,可将这些图像与ssDNA图进行配准,实现空间多模态分析。
在实际操作中,一般使用h5ad中提供的空间坐标和image链接信息(如uns/spatial字段)即可自动加载图像。若需要手动加载,可使用Python读取tif并按坐标映射。
主流平台性能横向对比
下面我们汇总目前主流的空间转录组平台,在若干关键指标上的差异:
| 平台 | 技术类别 | 空间分辨率 | 基因通量 | 多组学能力 | FFPE兼容 | 适用样本 / 优势场景 |
|---|---|---|---|---|---|---|
| 10x Visium (v1) | 测序型 | ~55 µm斑点直径,1--10细胞/点[7] | 全转录组(polyA捕获)[6];FFPE版探针Panel ~18k基因 | RNA为主(可结合组织影像,暂无蛋白检测) | 部分兼容(需探针panel) | 新鲜/固定冰冻组织;组织结构宏观概览,高通量筛查 |
| 10x Visium HD | 测序型 | ~2 µm特征尺寸,单细胞级分辨[66] | 全转录组(高密度捕获阵列) | RNA为主 | 是[15] | 新鲜/固定/FFPE;单细胞精度全景,适合异质性精细研究 |
| BGI Stereo-seq | 测序型 | 0.5 µm中心距,亚细胞分辨[21] | 全转录组(随机引物,无偏倚)[36] | RNA为主(可扩展微生物RNA) | 是(V2版支持)[24] | 新鲜/固定/FFPE;大尺度样本(cm级)单细胞图谱,如胚胎、器官、肿瘤微环境 |
| Slide-seq V2 | 测序型 | ~10 µm微珠直径,单细胞邻近[38] | 全转录组(敏感度较v1提高) | RNA为主 | 否 | 新鲜冷冻组织;小范围高分辨研究,需自建方案,适合方法开发 |
| HDST | 测序型 | ~2 µm微孔/珠,亚细胞分辨 | 全转录组(低捕获效率) | RNA为主 | 否 | 新鲜组织;原理验证性质,极限分辨率探索,目前应用少 |
| 10x Xenium | 成像型 | 单分子原位定位,亚细胞级[46] | 靶向Panel,可定制50--5000基因[47] | RNA+蛋白(支持蛋白Panel)[51] | 是[45] | 新鲜/FFPE切片;高通量细胞相互作用分析,集成度高,适合肿瘤等 |
| NanoString CosMX | 成像型 | 单分子成像,亚细胞级 | 靶向Panel,最高~18,000基因[54] | RNA+蛋白(如19k RNA + 72蛋白)[55] | 是[45] | FFPE/新鲜切片;全转录组+多蛋白分析,适合机制验证、临床研究 |
| Vizgen MERSCOPE | 成像型 | 单分子成像,亚细胞级 | 靶向Panel,最高~1000基因[67] | RNA为主(通常不测蛋白) | 是(新鲜及FFPE均可)[45] | 新鲜/FFPE切片;神经科学等精细图谱,成熟MERFISH方案,结果可靠 |
**表:**主流空间转录组平台比较[7][21][55]等。分辨率为厂家或文献报道的标称值;基因通量指单次实验可检测基因数范围;多组学指是否能同时检测其他分子(如蛋白);FFPE兼容性指是否适用于石蜡包埋样本。适用场景为根据平台特性总结的推荐应用。
**以上横向比较可以看出:**测序型平台在基因覆盖面上占优,适合发现未知分子特征,但其空间分辨往往有限,需要后续融合单细胞数据以提升细胞类型解析;成像型平台具有更高分辨率和更准确的单细胞表达矩阵,但一次实验能观察的基因数量受限,需要有针对性的panel设计。研究者应充分利用两类方法的互补优势,扬长避短地服务于各自的科研问题。
平台选型建议:不同研究场景的最佳选择
不同研究目的和样本条件下,适合采用的空间转录组策略也有所不同。以下结合基础研究、机制验证和临床转化三类场景,提供平台选型的建议:
- **基础研究(探索性课题):**在基础科研中,如构建组织/胚胎图谱、新现象的发现阶段,优先考虑测序型全景平台。例如,Stereo-seq和Visium可提供全转录组范围的数据,能够不偏倚地发现差异表达基因和新的空间表达热点区域[6]。若研究需要更高分辨率(如细胞分层结构或小型组织),并且具备相应资源,可以考虑Visium HD或Slide-seq等。
- **机制验证(假说驱动研究):**当有了明确的分子机制假说或候选标志物后,适合采用成像型定向验证的策略。成像平台如Xenium、CosMX、MERSCOPE可在单细胞/亚细胞层面验证目标基因的空间定位,并结合细胞分割分析细胞互作。如果已知需要验证的基因集合较明确,成像型平台的靶向panel模式更具性价比与解释性。
- **临床转化(生物标志物和诊断应用):**临床样本多为FFPE,平台兼容性与流程稳定性是关键。Visium FFPE、Visium HD、Xenium和CosMX均提供FFPE支持[15][45]。如果目标是发现新的疾病分子标志物,建议先用Visium在多个病例样本上进行筛选,因为其可同时覆盖全基因组表达,能统计出与临床表型相关的空间表达特征;若目标是构建可落地的诊断panel或验证特定通路,则可转向Xenium/CosMX进行更精确的单细胞定位与多组学评估。
结语:实践指引与延伸阅读
空间转录组学作为一项新锐技术,正以惊人的速度演进。从早期的方法验证到如今多种商业化平台的百花齐放,研究者面临的不仅是机遇,还有选择的挑战[70]。在实际开展研究时,我们建议科研人员把握以下要点:
首先,明确科学问题和样本限制。根据是探索未知还是验证已知、样本是动物模型还是临床组织、新鲜还是FFPE等,选择匹配的平台与实验设计。
其次,统筹实验设计与数据分析。空间转录组实验往往成本高、数据解析复杂,建议在方案设计阶段就考虑下游分析流程,包括QC、聚类、细胞类型注释、空间互作、空间差异基因等关键步骤。充分利用厂家软件(如Stereo-seq的SAW、10x的Space Ranger和Xenium Explorer,NanoString的CosMX分析管线等)以及开源工具(Seurat, Giotto, Scanpy, Squidpy等)。
再者,善用多模态融合。空间转录组并非孤立存在,它常常与组织学、单细胞测序等结合产生更大价值。例如,在获得Visium或Stereo-seq数据后,可用scRNA-seq作为reference进行细胞类型去卷积,提高对空间斑点(如Visium spot)或未标记细胞的注释准确性[71]。有条件的话,甚至可以进行空间多组学(转录+蛋白+空间代谢等),形成更完整的空间生态位解析框架。
最后,空间转录组学仍处于飞速发展阶段,新技术新方法层出不穷。应保持关注最新进展,持续更新自己的技术知识体系。阅读Nature Methods的测序型平台系统比较[1]和Nature Communications刊登的成像平台性能评估[64]等。这些研究提供了宝贵的客观数据,帮助我们理解各平台真实的性能边界。
👋 如果本文对你有帮助,欢迎点赞、收藏
相关深入阅读
- Nature Methods (2024) -- 对11种测序型空间转录组方法的系统比较分析[1][70]:提供不同平台在有效分辨率、捕获效率等方面的客观评估标准。
- Nature Communications (2023) -- 三大成像型平台(Xenium, CosMX, MERSCOPE)在FFPE组织中的基准测试[64][45]:对成像数据的检测灵敏度、特异性、细胞分割准确率等进行了详尽比较。
- Cheng et al., J. Genetics and Genomics (2023) -- 《空间分辨转录组学技术的进展、应用与挑战》综述[43]:由华大团队撰写,涵盖了测序型与成像型技术的发展脉络和代表性应用案例。
- 10x Genomics & NanoString 官方资源 -- 包括10x的技术白皮书、客户案例和NanoString的CosMX实践指南等[72][55]:获取一手参数指标和操作建议,辅助方案设计。
总之,空间转录组学正将我们对生命体系的认识提升到"空间+多维度"的新高度。从宏观组织结构到单细胞空间生态位,从基础科学发现到临床转化应用,空间转录组正不断拓展生物学研究的边界。
-
1\] \[68\] \[70\] Systematic comparison of sequencing-based spatial transcriptomic methods \| Nature Methods https://www.nature.com/articles/s41592-024-02325-3?..=cookies_not_supported\&code=54edfeb4-d8c9-493c-b0b5-0cd0e6491f95
https://www.completegenomics.com/methods/spatial-transcriptomics/
-
3\] \[5\] \[6\] \[9\] \[11\] \[45\] \[48\] \[60\] \[61\] \[64\] \[73\] ...imaging spatial transcriptomics platforms in FFPE tissues - PMC https://pmc.ncbi.nlm.nih.gov/articles/PMC10723440/
-
8\] What is the spatial resolution and configuration of the capture area ... https://kb.10xgenomics.com/s/article/360035487572-W...ation-of-the-capture-area-of-the-Visium-v1-Gene-Expression-Slide
-
12\] Visium Spatial Gene Expression - OSTR - National Cancer Institute https://ostr.ccr.cancer.gov/emerging-technologies/spatial-biology/visium/
https://nanostring.com/products/cosmx-spatial-molecular-imager/
-
14\] \[15\] \[16\] \[17\] \[18\] \[19\] \[44\] \[66\] High-definition spatial transcriptomic profiling of immune cell populations in colorectal cancer \| Nature Genetics https://www.nature.com/articles/s41588-025-02193-3?..=cookies_not_supported\&code=36b37d96-6886-4e4d-9fd3-e882799164fa
-
37\] SAW: an efficient and accurate data analysis workflow for Stereo ... https://gigabytejournal.com/articles/111
https://macoskolab.com/wp-content/uploads/2021/08/ddf8b586-248a-05ea-96b7-d3cfcd78ae0f-1.pdf
-
43\] Spatially resolved transcriptomics: a comprehensive review of their ... https://www.sciencedirect.com/science/article/pii/S1673852723000759
-
57\] Moving Beyond scRNA-seq: The Power of CosMx Whole ... https://nanostring.com/blog/moving-beyond-scrna-seq-the-power-of-cosmx-whole-transcriptome/
https://brukerspatialbiology.com/spatial-biology/cosmx-service/cosmx-spatial-transcriptomics-panels/
-
62\] CosMx® Same-Cell Multiomics - NanoString https://nanostring.com/products/cosmx-spatial-molecular-imager/cosmx-same-cell-multiomics/
-
65\] Optimizing Xenium In Situ data utility by quality assessment and best ... https://www.nature.com/articles/s41592-025-02617-2