Python 轻量级 UI:EEG 与 fNIRS 预处理图形界面
- [NeuroSignal Preprocessor 使用说明](#NeuroSignal Preprocessor 使用说明)
-
- [1. 软件简介](#1. 软件简介)
- [2. 系统要求与运行环境](#2. 系统要求与运行环境)
-
- [2.1 操作系统](#2.1 操作系统)
- [2.2 硬件建议](#2.2 硬件建议)
- [2.3 必要运行库](#2.3 必要运行库)
- [3. 安装与启动方式](#3. 安装与启动方式)
-
- [3.1 文件结构说明](#3.1 文件结构说明)
- [3.2 启动方式](#3.2 启动方式)
- [4. 软件界面说明](#4. 软件界面说明)
- [5. EEG模块使用说明](#5. EEG模块使用说明)
-
- [5.1. 模块设计理念](#5.1. 模块设计理念)
- [5.2. 数据载入(Load Data)](#5.2. 数据载入(Load Data))
-
- [5.2.1 数据载入](#5.2.1 数据载入)
- [5.2.2 支持格式](#5.2.2 支持格式)
- [5.2.3 数据状态指示](#5.2.3 数据状态指示)
- [5.3. EEG预处理](#5.3. EEG预处理)
-
- [5.3.1 坏道标记(Bad Channels)](#5.3.1 坏道标记(Bad Channels))
- [5.3.2 EOG 通道标记 (Mark EOG)](#5.3.2 EOG 通道标记 (Mark EOG))
- [5.3.3 滤波(FILTER)](#5.3.3 滤波(FILTER))
- [5.3.4 ICA(独立成分分析)](#5.3.4 ICA(独立成分分析))
-
- 1)目的
- [2)ICA 参数说明](#2)ICA 参数说明)
-
- n_components(成分数量)
- [method(ICA 算法)](#method(ICA 算法))
- random_state(随机种子)
- 参数设置建议
- [3) 执行 ICA 分解(Fit ICA)](#3) 执行 ICA 分解(Fit ICA))
- [4)ICA 成分判别与选择](#4)ICA 成分判别与选择)
- [5)编辑已选择的成分(Edit excluded comps)](#5)编辑已选择的成分(Edit excluded comps))
- [6)应用 ICA 去伪迹(Apply ICA)](#6)应用 ICA 去伪迹(Apply ICA))
- 7)科研使用建议与注意事项
- [5.3.5 重参考(Re-reference)](#5.3.5 重参考(Re-reference))
- [5.3.6 坏道插值(Interpolate)](#5.3.6 坏道插值(Interpolate))
-
-
- [5.3.7 保存结果](#5.3.7 保存结果)
- [5.4 特征提取](#5.4 特征提取)
-
- [5.4.1 Epoch / 事件标记(Epoch / Mark)](#5.4.1 Epoch / 事件标记(Epoch / Mark))
-
- 1) 支持模式 支持模式)
- 2) 按 Mark 切分 Epoch 按 Mark 切分 Epoch)
- 3) 构建 Epochs 构建 Epochs)
- [5.4.2 时域特征:熵特征](#5.4.2 时域特征:熵特征)
- [5.4.3 时频特征(Time--Frequency / ERSP):时频谱](#5.4.3 时频特征(Time–Frequency / ERSP):时频谱)
- [5.4.4 功能连接](#5.4.4 功能连接)
-
- 1) 连接指标 连接指标)
- 2) 连接矩阵与可视化 连接矩阵与可视化)
- 3) 网络分析 网络分析)
- 4)日志(Log)
-
- [6. fNIRS模块使用说明](#6. fNIRS模块使用说明)
-
- [6.1 载入数据(Load Data)](#6.1 载入数据(Load Data))
- [6.2 预处理(Preprocessing)](#6.2 预处理(Preprocessing))
-
- [6.2.1 转换、质量控制与血红蛋白计算](#6.2.1 转换、质量控制与血红蛋白计算)
- [6.2.2 Filter + Epoch 可视化(Epoch Visualization)](#6.2.2 Filter + Epoch 可视化(Epoch Visualization))
- [6.3 特征提取(Feature Extraction)](#6.3 特征提取(Feature Extraction))
-
- [6.3.1 Epoch 可视化(Epoch Visualization)](#6.3.1 Epoch 可视化(Epoch Visualization))
- [6.3.2 特征提取:Feature Extraction](#6.3.2 特征提取:Feature Extraction)
- [6.4 数据保存(Save)](#6.4 数据保存(Save))
- [7. sEMG模块使用说明](#7. sEMG模块使用说明)
- [8. 常见问题(FAQ)](#8. 常见问题(FAQ))
-
- [Q1:双击软件没有反应 / 一闪而过](#Q1:双击软件没有反应 / 一闪而过)
- Q2:软件提示"某某库未安装"
- Q3:处理大文件时运行较慢
- [9. 注意事项与已知限制](#9. 注意事项与已知限制)
- [10. 版本信息与联系方式](#10. 版本信息与联系方式)
NeuroSignal Preprocessor 使用说明
1. 软件简介
NeuroSignal Preprocessor 是一款用于 EEG / fNIRS / EMG 神经信号数据的桌面预处理与特征提取软件。
本软件面向科研人员与工程用户设计,提供图形化界面,支持从数据加载、预处理到特征提取的完整流程,无需安装 Python 环境,双击即可运行。
- 本软件由作者在科研实践中自主开发并持续维护,主要用于神经信号数据预处理与特征分析研究。
- 软件功能仍在不断完善中,由于实验条件与数据差异,在特定场景下可能存在未覆盖的边界情况。
- 软件输出结果仅供科研与方法探索参考,不构成任何临床或商业用途的保证。
- 相关分析结果应结合具体实验设计与数据特性进行合理解释。
- 如在使用过程中发现问题或有改进建议,欢迎反馈以协助改进。
软件下载链接:https://weisihong9.github.io/2025/12/11/NeuroSignalPreprocessor/
2. 系统要求与运行环境
2.1 操作系统
- Windows 10 / Windows 11(64 位)
2.2 硬件建议
- 内存:8 GB(推荐 16 GB 及以上)
- 磁盘空间:至少 2 GB 可用空间
2.3 必要运行库
本软件依赖以下系统运行库:
- Microsoft Visual C++ Redistributable 2015--2022(x64)
若软件无法启动或启动时报错,请优先确认已安装该运行库。
3. 安装与启动方式
3.1 文件结构说明
解压后目录结构如下:
javascript
NeuroSignalPreprocessor/
├─ NeuroSignalPreprocessor.exe
├─ _internal/
├─ Example_data (示例数据) ⚠ 请勿单独移动或复制 exe 文件,必须保留整个文件夹结构,否则软件将无法运行。
3.2 启动方式
- 进入
NeuroSignalPreprocessor文件夹 - 双击
NeuroSignalPreprocessor.exe - 软件启动后将显示主界面
4. 软件界面说明
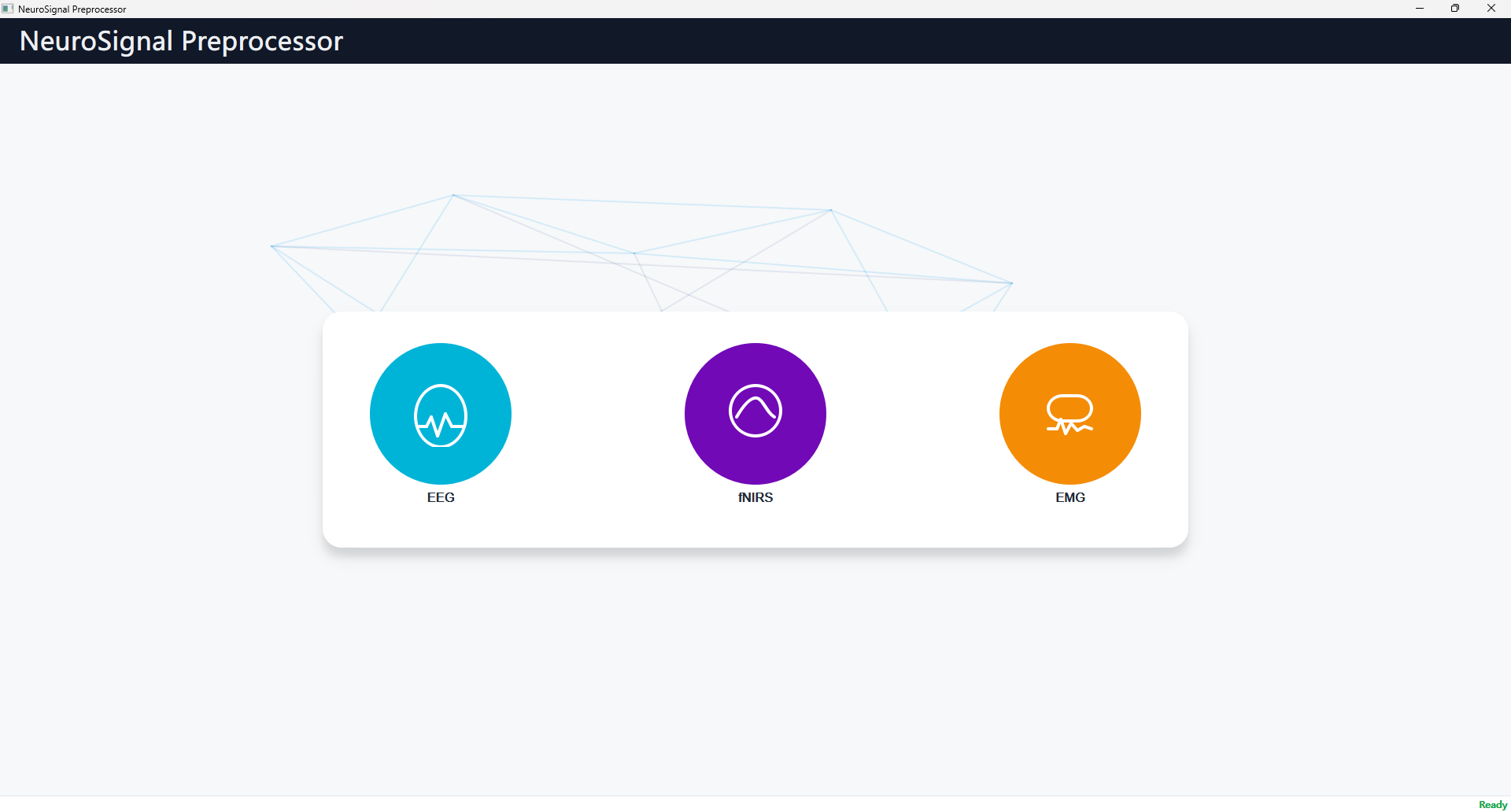
启动后进入主界面,包含以下三个模块:
- EEG:脑电信号预处理与特征提取
- fNIRS:近红外脑功能成像信号处理
- EMG:肌电信号处理
点击对应模块即可进入功能页面。
5. EEG模块使用说明
5.1. 模块设计理念
EEG 模块基于标准脑电信号处理流程设计,整体思路与 MNE-Python / EEGLAB 等主流科研工具保持一致,强调:
- 流程可控(step-by-step)
- 参数可解释
- 人工干预与自动处理结合
- 结果可复现
推荐使用顺序如下:
javascript
Load Data
→ Mark Bad Channels
→ Filter
→ ICA
→ Re-reference
→ Interpolate
→ Epoch / Mark
→ Feature Extraction
→ Save5.2. 数据载入(Load Data)
5.2.1 数据载入
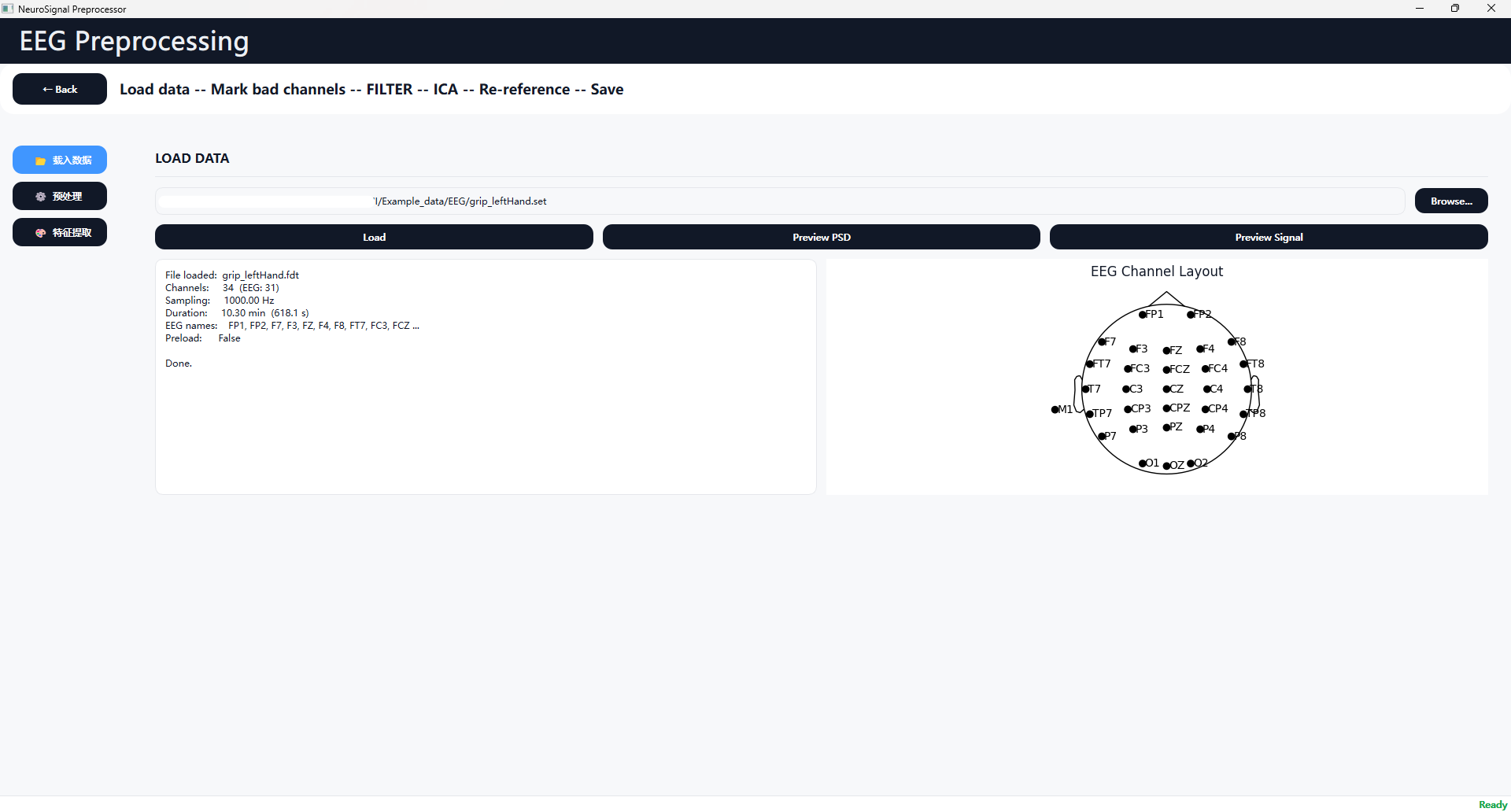
在 Load Data 页面中,用户可加载原始 EEG 数据文件。
系统基于 MNE 框架读取数据,并保持数据为 raw 状态 。
通过点击 Browse 按钮打开文件选择对话框,选择目标 EEG 数据文件并确认,即可完成数据载入。
5.2.2 支持格式
.set(EEGLAB).fif(MNE).edf / .bdf.vhdr(BrainVision)
5.2.3 数据状态指示
加载成功后,界面左侧将显示:
- 当前数据状态(raw)
- EEG / EOG 通道数量
- 当前 bad channel 数量
- 通道位置
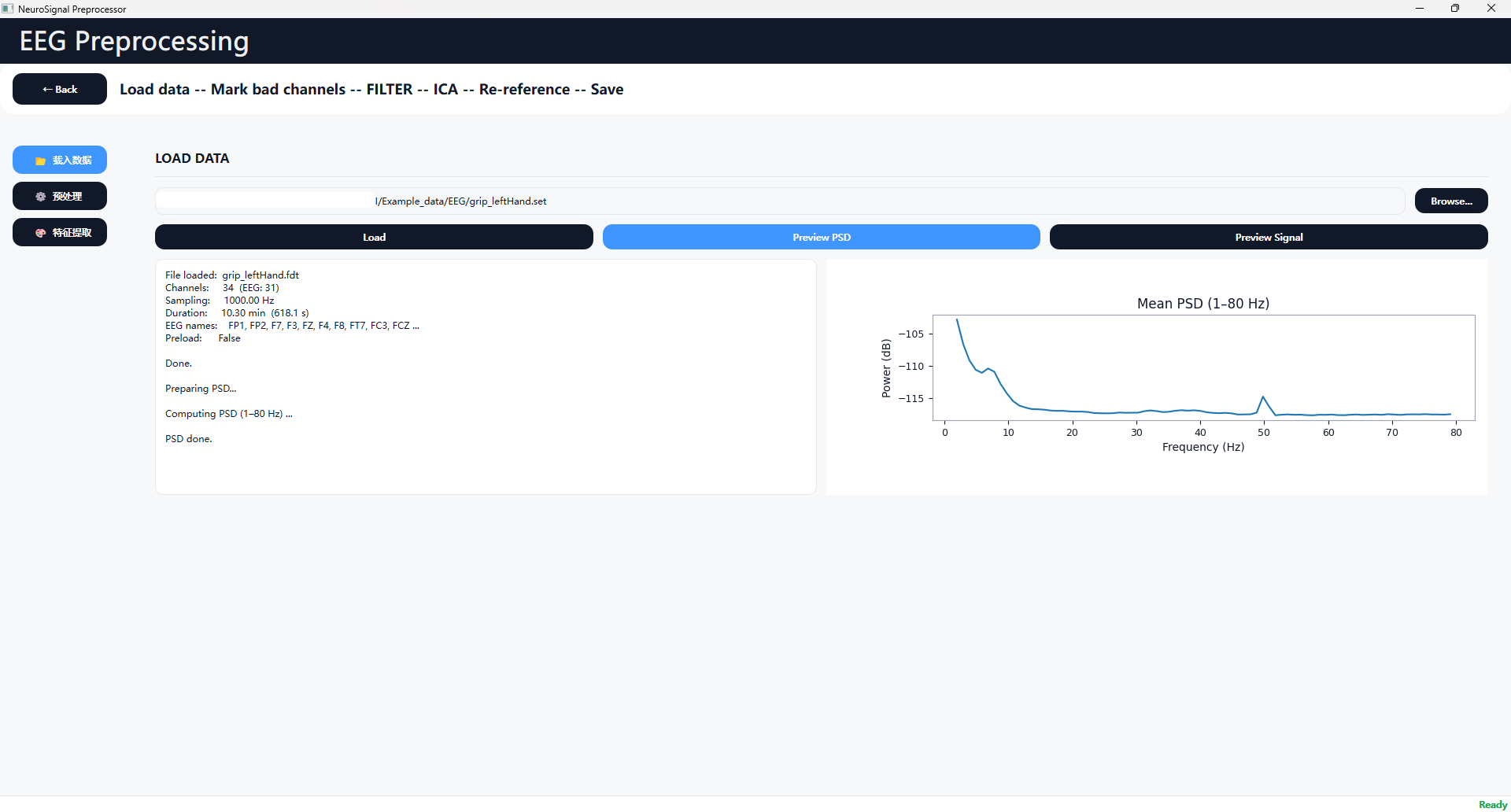
通过点击Preview PSD按钮可查看信号的功率谱密度
5.3. EEG预处理
5.3.1 坏道标记(Bad Channels)
坏道(Bad Channels)是 EEG 预处理中必须处理的问题,常见原因包括:
- 电极脱落
- 高阻抗
- 持续高噪声
- 运动伪迹
在后续滤波、ICA 和重参考前,明确坏道列表至关重要。
在 Mark Bad Channels 页面中,用户可:
- 预览原始信号(Preview Raw)
- 手动选择通道并标记为 bad
- 撤销(Undo)操作
选择坏道(Bad Channels)的方式主要有以下两种:
-
基于原始信号可视化标记
- 点击
Preview Raw按钮,系统将弹出原始 EEG 信号浏览窗口 - 在信号窗口中,用户可通过点击通道名称选择需要标记为坏道的通道
- 被选中的通道将以灰色显示以示区分
- 确认选择后,关闭信号窗口,所选通道将被自动记录为坏道
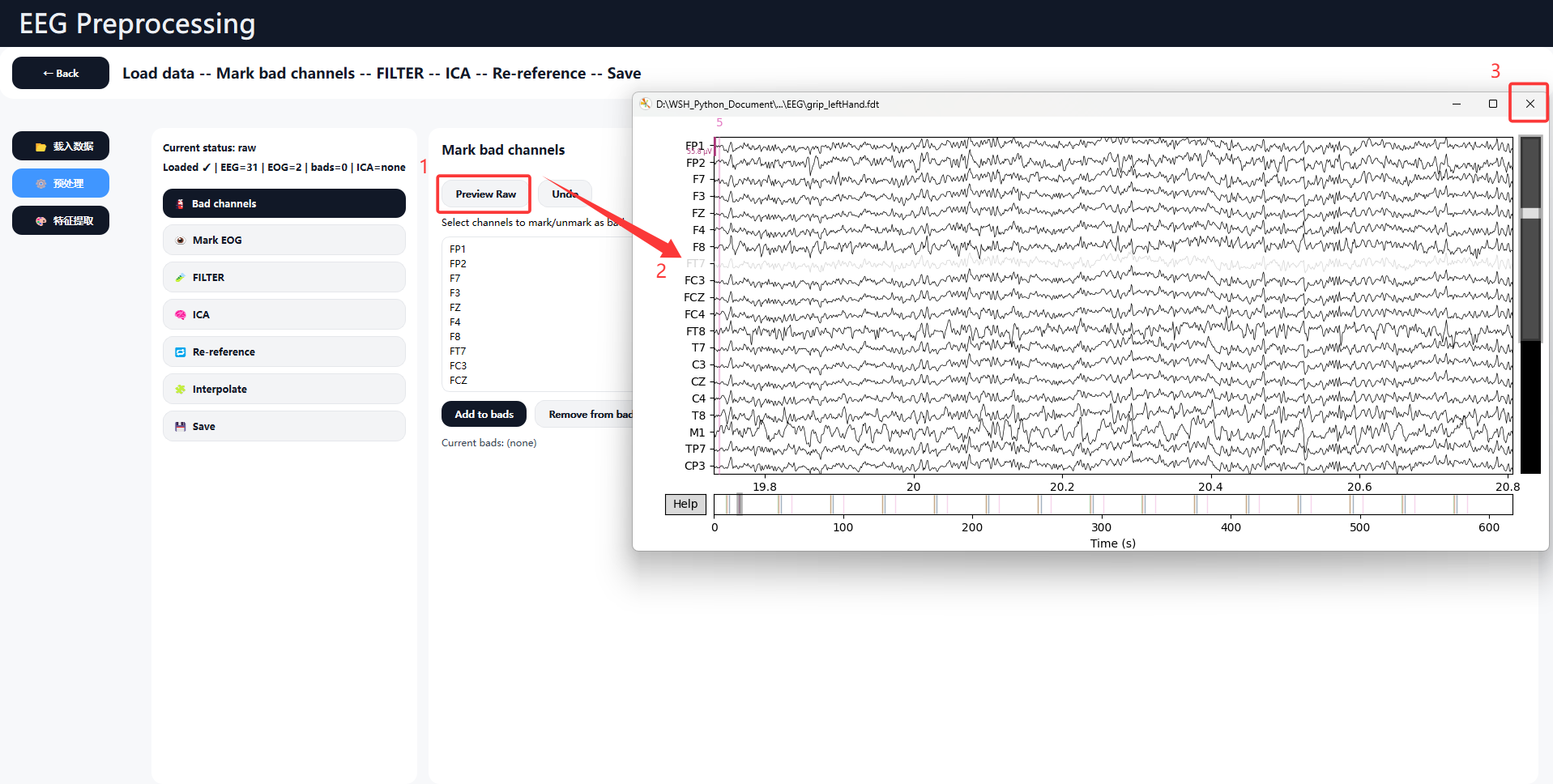
- 点击
-
基于通道列表的手动标记
- 用户亦可在主界面的通道列表中直接选择需要标记的通道,
- 点击
Add to bads按钮后,所选通道将被加入坏道列表, - 并在界面下方的
Current bads区域中实时显示。 - 如需撤销已标记的坏道,可点击
Undo按钮以恢复至上一步状态。
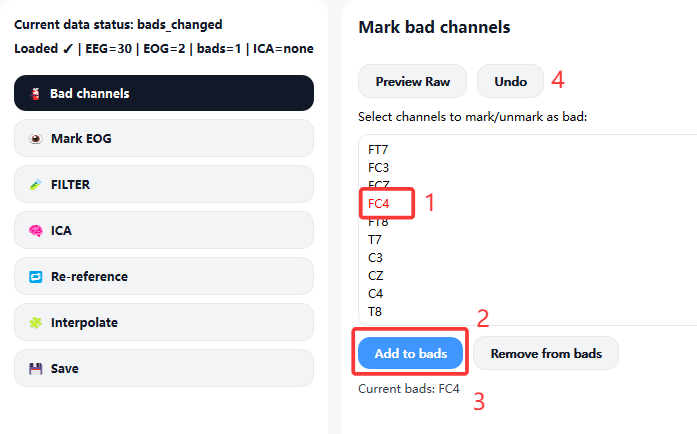
系统会将坏道信息写入raw.info['bads'],,并在后续滤波、ICA、重参考及插值过程中自动继承。
5.3.2 EOG 通道标记 (Mark EOG)
通过 Mark EOG 功能,可指定眼电通道,用于:
- ICA 中自动识别眼动伪迹成分
- 提高伪迹去除准确性
在数据加载阶段,系统将对通道类型进行自动检测,并完成以下操作:
-
自动识别眼电通道
系统将在数据加载完成后自动识别可能的眼电通道,并将其加入
Current EOG列表中,供后续处理流程使用。 -
自动识别结果的人工修正
若自动识别的眼电通道与实际通道类型不一致,用户可在通道列表中选中对应通道;
点击
Set selected back to EEG按钮,将该通道恢复为普通 EEG 通道,从而撤销眼电标记。 -
手动补充眼电通道
对于未被系统自动识别的眼电通道,用户可在通道列表中手动选中相应通道;
点击
Set selected to EOG将其标记为 EOG 通道,使其加入Current EOG列表。
所有已确认的 EOG 通道信息将被用于 ICA 阶段的伪迹成分识别与分类,以辅助眼动伪迹的自动检测与去除。
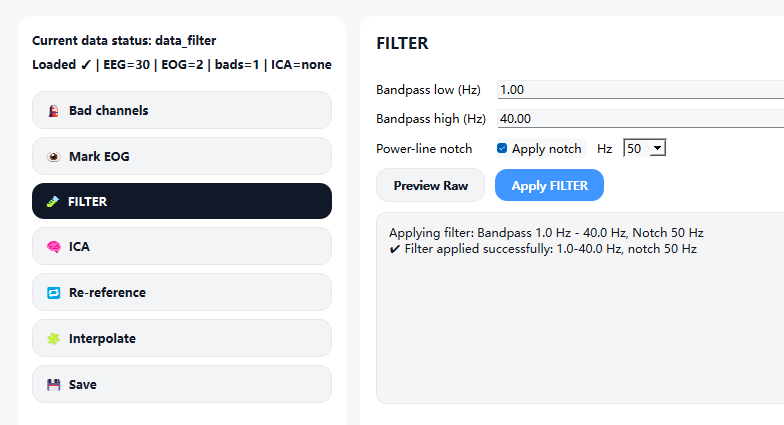
5.3.3 滤波(FILTER)
1)支持类型
- 带通滤波(Band-pass)
- 陷波滤波(Notch)
2)常用参数建议(经验值)
| 场景 | 低频 (Hz) | 高频 (Hz) |
|---|---|---|
| ERP | 0.1 | 30 |
| 频谱分析 | 1 | 40 |
| ICA 前处理 | 1 | 40 |
陷波滤波(Notch Filter)通常用于去除电源工频干扰,其常用设置如下:
- 50 Hz:适用于中国、欧洲等使用 50 Hz 市电频率的地区
- 60 Hz:适用于北美等使用 60 Hz 市电频率的地区
在完成滤波参数设置后,点击 Apply FILTER 按钮以执行滤波操作。
滤波成功后,界面下方将显示本次滤波所使用的参数信息,并提示 Filter applied successfully,以确认滤波过程已顺利完成。
5.3.4 ICA(独立成分分析)
1)目的
独立成分分析(Independent Component Analysis, ICA)是一种常用的盲源分离方法,
用于将多通道 EEG 信号分解为若干统计独立的成分,从而实现对生理与非生理伪迹的识别与剔除。
在本软件中,ICA 主要用于分离和处理以下常见伪迹来源:
- 眼动伪迹(EOG):眨眼、眼球转动引起的低频大幅度信号
- 心电伪迹(ECG):心跳相关的周期性成分
- 肌电伪迹(EMG):高频、局部化的肌肉活动信号
- 高频噪声:与真实脑活动无关的随机或系统性噪声
推荐流程
-
- 在完成滤波与坏道标记后执行 ICA
-
- 结合 EOG 通道自动辅助识别伪迹成分
-
- 人工确认并剔除无效成分
2)ICA 参数说明
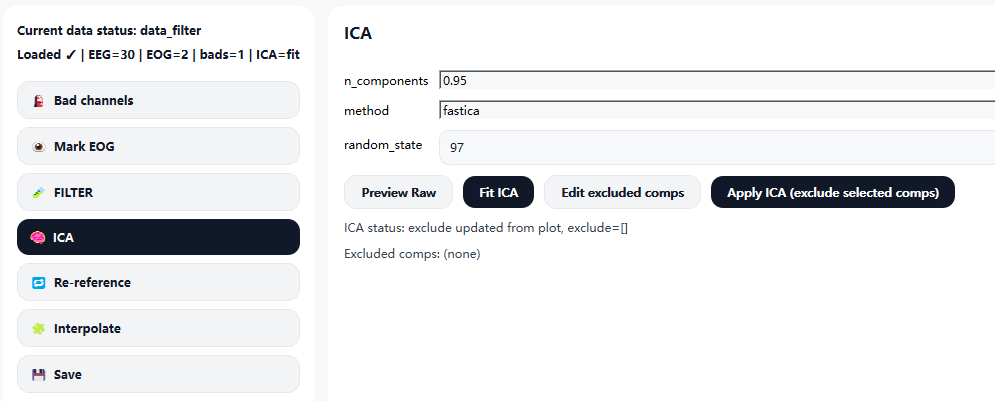
n_components(成分数量)
用于指定 ICA 分解时保留的成分数量或数据方差比例。
常见设置方式包括:
-
0.95 / 0.99(推荐)
表示保留 95% / 99% 的数据方差 ,系统将根据数据自动确定对应的 ICA 成分数量。
该方式在保证主要信号信息的同时,有助于减少噪声与过拟合,适用于大多数 EEG 数据分析场景。
-
整数值(如 20、30、50)
表示固定提取指定数量的 ICA 成分。
适用于通道数量固定、对 ICA 成分数量有明确经验或实验要求的情况。
使用建议 :
若不确定应提取多少个成分,建议优先使用 0.95 或 0.99 作为经验设置。
method(ICA 算法)
用于指定执行 ICA 分解所采用的算法类型。
- fastica(默认)
一种计算效率高、在 EEG 数据分析中应用广泛的 ICA 算法,
在大多数常规预处理任务中能够提供稳定可靠的分解结果。
random_state(随机种子)
用于控制 ICA 算法中的随机初始化过程。
- 设置固定的
random_state(如 97),
可保证在相同数据与参数条件下多次运行 ICA 得到一致的结果,
有助于提高分析流程的可复现性 与科研一致性。
参数设置建议
-
常规预处理 / 初次使用
n_components = 0.95method = fasticarandom_state保持默认值
-
需要严格复现实验结果
- 固定
random_state数值
- 固定
-
对 ICA 成分数量有明确经验
- 使用整数形式的
n_components
- 使用整数形式的
3) 执行 ICA 分解(Fit ICA)
- 点击
Fit ICA按钮后,系统将在当前已完成滤波与坏道标记的数据基础上执行 ICA 分解。 - ICA 拟合完成后,系统将自动弹出 ICA 成分可视化窗口,包括:
- 成分空间分布(头皮地形图, topography)
- 成分时间序列(ICA sources)
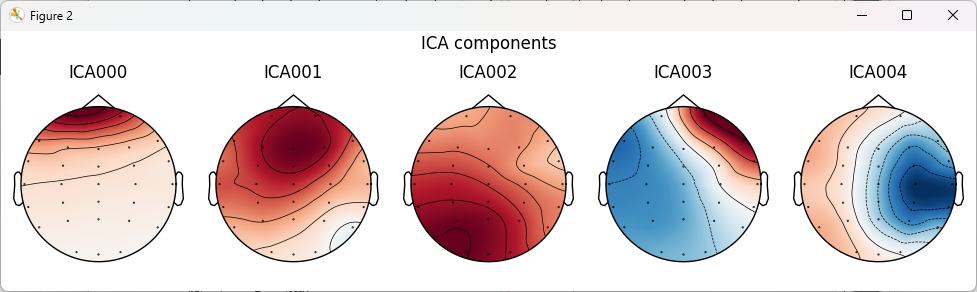
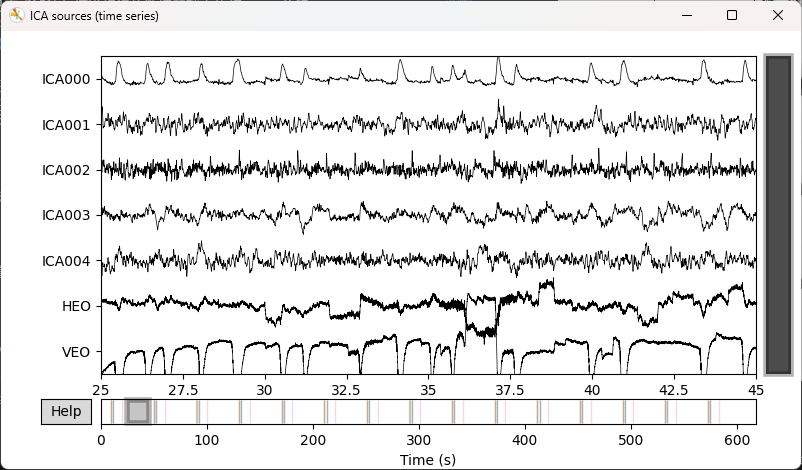
这些可视化结果用于辅助用户判断各独立成分的物理与生理意义。
4)ICA 成分判别与选择
在 ICA 成分窗口中,用户可基于以下信息对成分进行判别:
- 成分时间序列的波形特征(如眨眼相关的尖峰或缓慢漂移)
- 成分头皮分布特征(如额区主导的眼动模式)
- 与 EOG 通道的相关性(若已正确设置 EOG 通道)
成分选择方式:
- 点击需要剔除的 ICA 成分,对应成分将被高亮/置灰标记
- 确认选择后,关闭成分窗口
- 被选中的成分编号将显示在主界面的
Excluded comps信息区域中
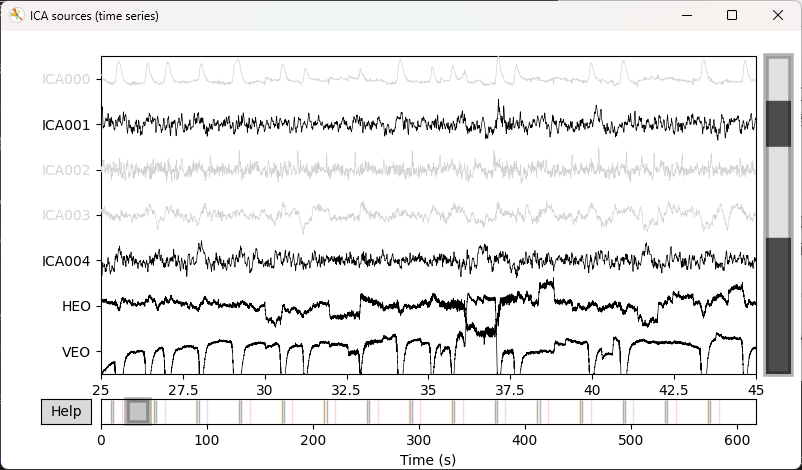
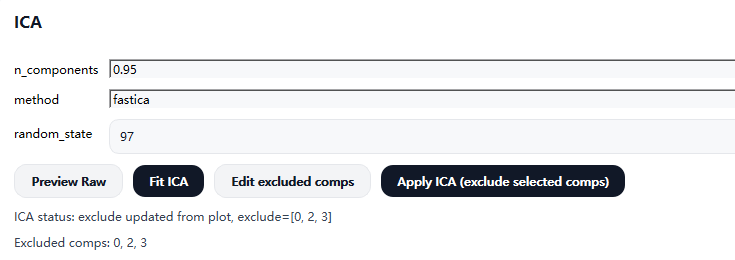
5)编辑已选择的成分(Edit excluded comps)
- 若需对已选择的剔除成分进行修改,可点击
Edit excluded comps按钮 - 系统将弹出成分列表窗口,用户可:
- 勾选或取消勾选需要剔除的 ICA 成分
- 保存更新后的剔除列表
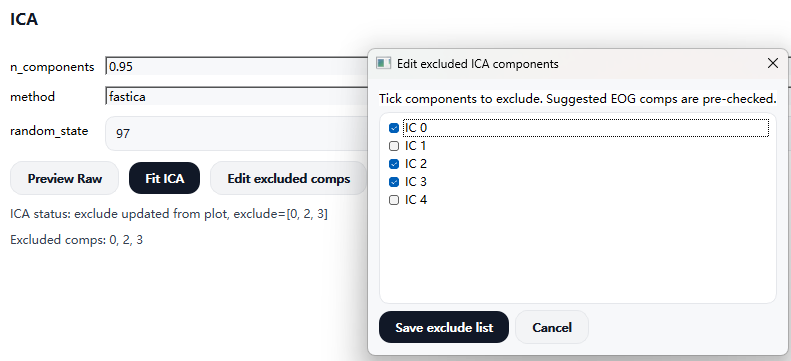
6)应用 ICA 去伪迹(Apply ICA)
- 在确认需要剔除的成分后,点击
Apply ICA (exclude selected comps)按钮 - 系统将基于所选成分,对原始数据进行重构并移除对应伪迹成分
- ICA 应用完成后,系统将自动弹出结果可视化窗口,用于对去伪迹效果进行直观评估。
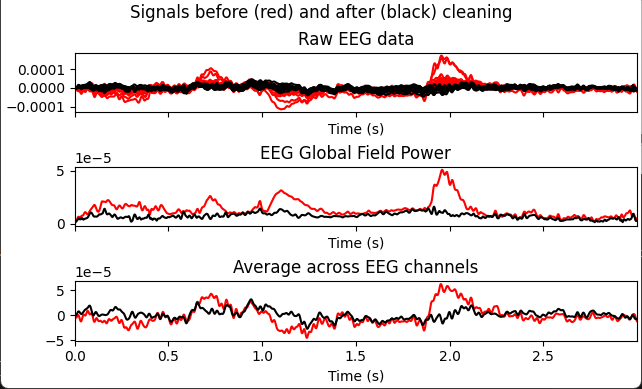
该可视化窗口通常包括以下内容:
-
原始信号与清理后信号对比
以不同颜色显示 ICA 应用前(如红色)与应用后(如黑色)的 EEG 信号,用于直观观察伪迹去除效果。
-
全局场功率(Global Field Power, GFP)变化
对比清理前后 EEG 的整体能量分布变化,辅助评估大幅度伪迹是否得到有效抑制。
-
跨通道平均信号对比
展示所有 EEG 通道平均信号在 ICA 应用前后的变化趋势,以判断整体信号稳定性是否得到改善。
通过上述可视化结果,用户可综合判断 ICA 成分剔除是否合理,在确认结果满意后,可继续执行后续的重参考、坏道插值或 epoch 切分等处理流程。
7)科研使用建议与注意事项
- 建议在 完成滤波与坏道标记之后 再执行 ICA
- ICA 成分剔除应结合自动辅助结果与人工判断,避免过度去除真实脑信号
- 推荐记录每次 ICA 使用的参数与被剔除成分编号,以保证分析过程的可复现性
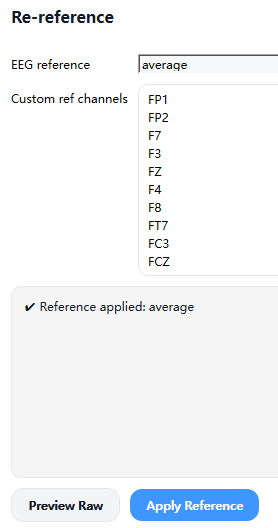
5.3.5 重参考(Re-reference)
重参考用于重新定义 EEG 信号的参考基准,对信号幅值分布与空间拓扑具有重要影响。
本软件支持以下两种常用重参考方式:
-
平均参考(Average Reference)
将所有有效 EEG 通道的平均信号作为参考基准,
有助于减少单一参考电极带来的偏置,适用于通道分布较为均匀的高密度 EEG 数据。
-
指定参考通道(Custom Reference)
选择一个或多个特定通道作为参考基准,
适用于实验范式中已明确参考电极位置或需与既往研究保持一致的情况。
确认参考方式后,点击 Apply Reference 按钮以应用重参考操作。
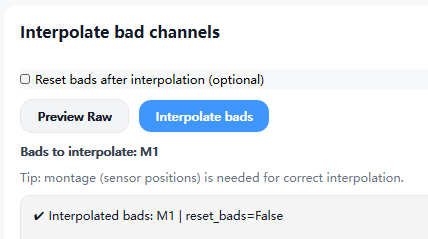
5.3.6 坏道插值(Interpolate)
坏道插值用于对已标记的坏道进行空间重建,以减少通道缺失对数据完整性和空间分析的影响。
- 插值基于电极空间位置与头模型信息,利用邻近通道信号对坏道进行估计重建
- 该操作有助于在保留整体空间拓扑结构的同时,降低因坏道剔除带来的数据损失
Reset bads after interpolation(可选)
该选项用于控制插值完成后,原先标记为坏道(bad channels)的通道状态是否重置。
- 勾选该选项 :
插值完成后,已插值重建的通道将被从坏道列表中移除,并重新作为有效 EEG 通道参与后续分析。 - 未勾选该选项 :
插值完成后,通道信号已被重建,但其坏道标记将被保留,
在后续分析中仍会被视为原始坏道,仅用于保持处理记录的可追溯性。
该设置不会影响插值本身的计算结果,仅影响通道在后续流程中的标记状态。
确认无误后点击 Interpolate bads 按钮以执行插值操作。
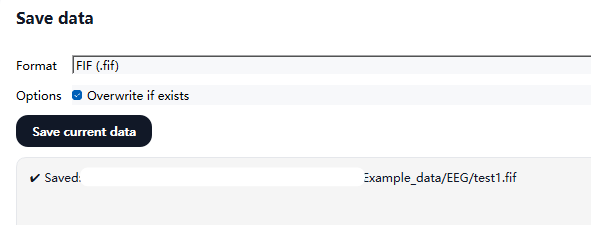
5.3.7 保存结果
预处理完成后,可将当前处理状态下的数据保存到本地文件。
- 支持保存为 FIF(
.fif) 、EDF(.edf) 或 CSV(channels × time) 等格式,具体格式可在界面中选择。 - 保存操作仅作用于当前处理后的数据副本,不会修改原始数据文件。
确认保存格式后,点击 Save current data 按钮即可完成数据导出。
5.4 特征提取
当前支持特征类型
- 时域特征
- 频域特征
- 功能连接特征
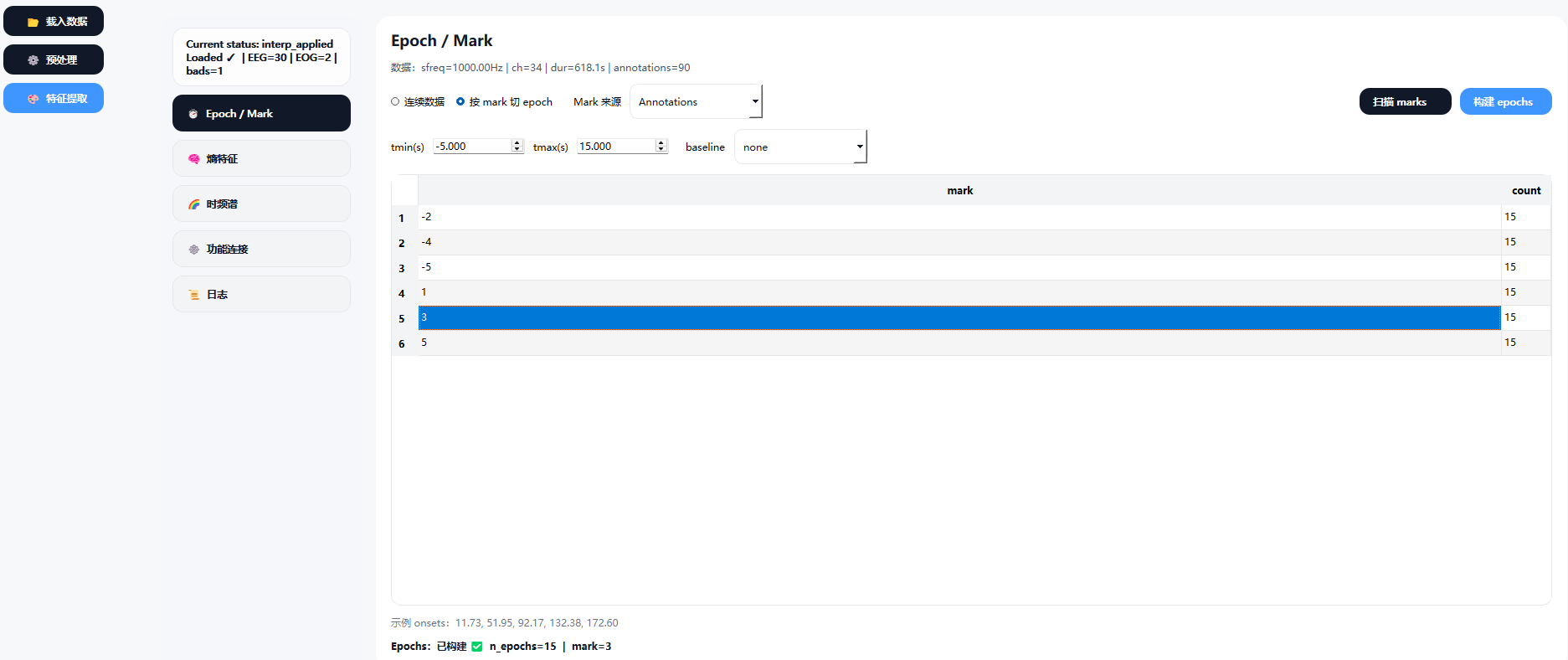
5.4.1 Epoch / 事件标记(Epoch / Mark)
该模块用于将连续 EEG 信号切分为与事件(刺激/标记)相关的时间片段(epochs),
以支持后续的事件相关分析。
1) 支持模式
- 连续数据模式:不进行事件切分,保留连续信号结构。
- 按标记切分 Epoch:基于数据采集过程中记录的事件标记(marks / annotations)构建 epochs。
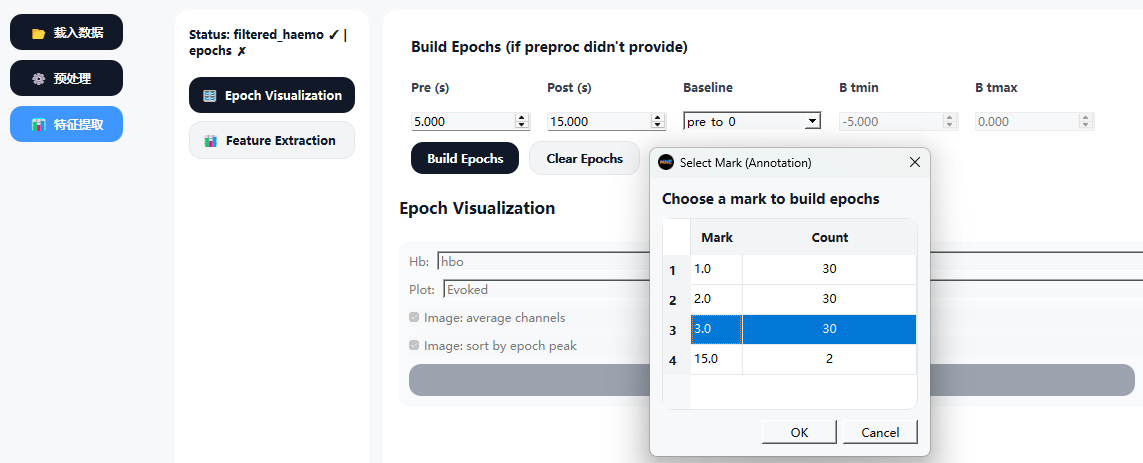
2) 按 Mark 切分 Epoch
在选择 "按 mark 切 epoch" 模式后,系统将以表格形式列出当前数据中检测到的事件标记,包括各标记的编号及出现次数。
用户可进行以下设置:
- Mark 来源:选择事件标记来源(如 Annotations)
- 时间窗设置 :
tmin:事件前时间(秒)tmax:事件后时间(秒)
- 基线校正(baseline):设置基线区间或选择不进行基线校正
3) 构建 Epochs
- 单击选中表格中需要分析的事件标记(mark)
- 点击
构建 epochs按钮执行切分操作 - 构建成功后,界面下方将显示 epoch 状态信息,包括:
- 是否已成功构建
- epoch 数量(
n_epochs) - 当前所选事件标记编号
该结果可直接用于后续的特征提取、时频分析或功能连接分析。
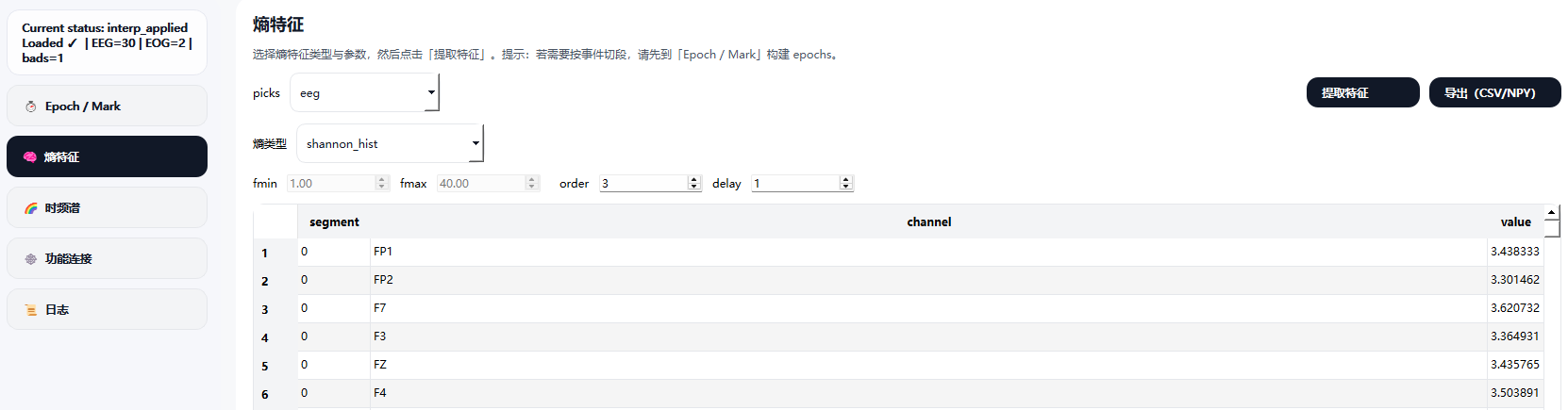
5.4.2 时域特征:熵特征
该模块用于从 EEG 信号中提取多种熵类特征,以量化信号的复杂性与不确定性。
用户可根据研究需求选择不同的熵类型(如 Shannon 熵、谱熵、排列熵等),并设置相应参数后执行特征计算。
在确认参数配置后,点击 提取特征 按钮即可完成熵特征提取,结果将以通道为单位进行汇总,并可导出用于后续统计分析或机器学习建模。
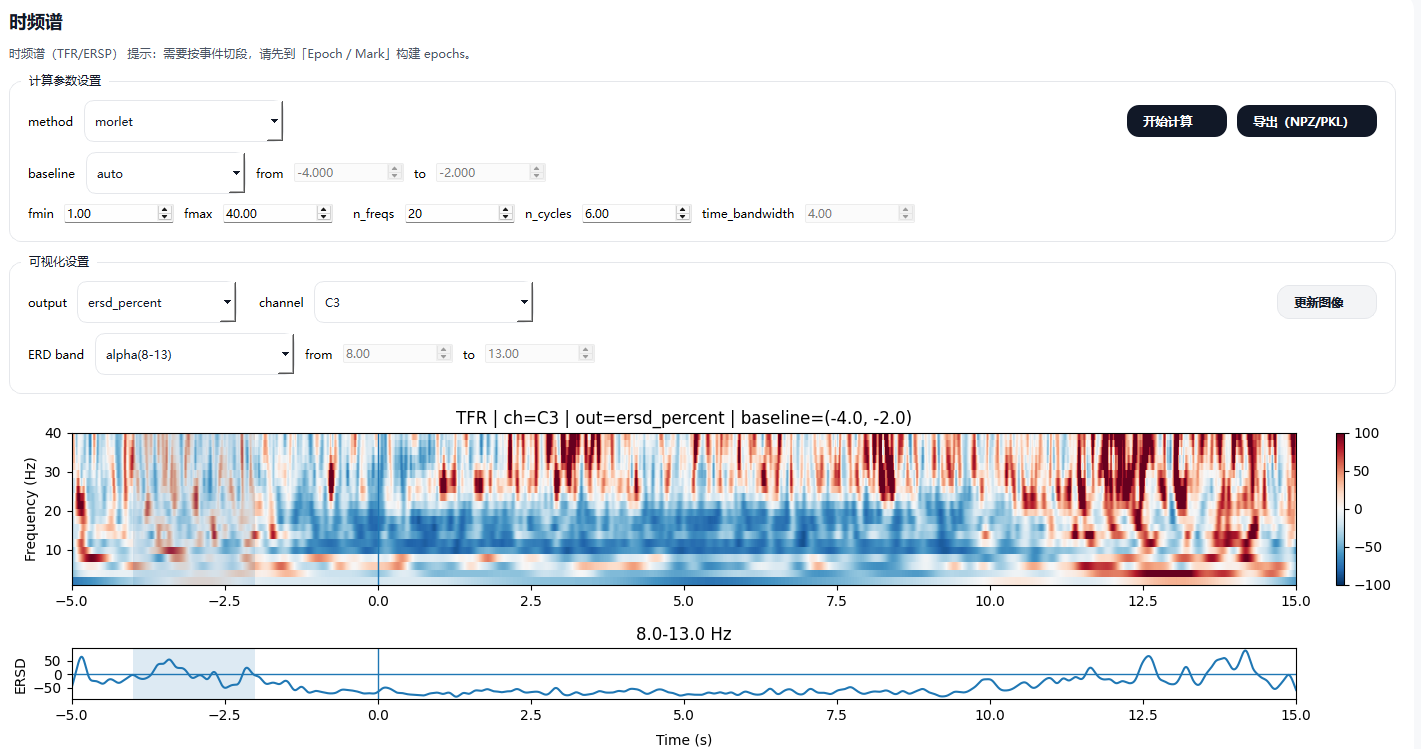
5.4.3 时频特征(Time--Frequency / ERSP):时频谱
该模块用于对 EEG 信号进行时频分析,以刻画不同频段功率随时间的动态变化,常用于事件相关去同步/同步(ERD/ERS)分析。
1)计算方法
- 可选择 Morlet 小波变换 或 Multitaper 对 epochs 数据进行时频分解
- 支持自定义:
- 频率范围(
fmin--fmax) - 频率分辨率(
n_freqs) - 小波周期数(
n_cycles) - 时间带宽参数
- 频率范围(
2)基线校正
- 支持自动或自定义基线区间
- 可输出相对基线的功率变化(如百分比形式)
3)可视化与输出
- 支持按单通道或通道平均方式显示结果
- 可选择特定频段(如 α、β 波段)进行 ERD/ERS 分析(选择后需点击**
更新图像** 按钮) - 结果可导出用于后续统计分析或机器学习建模
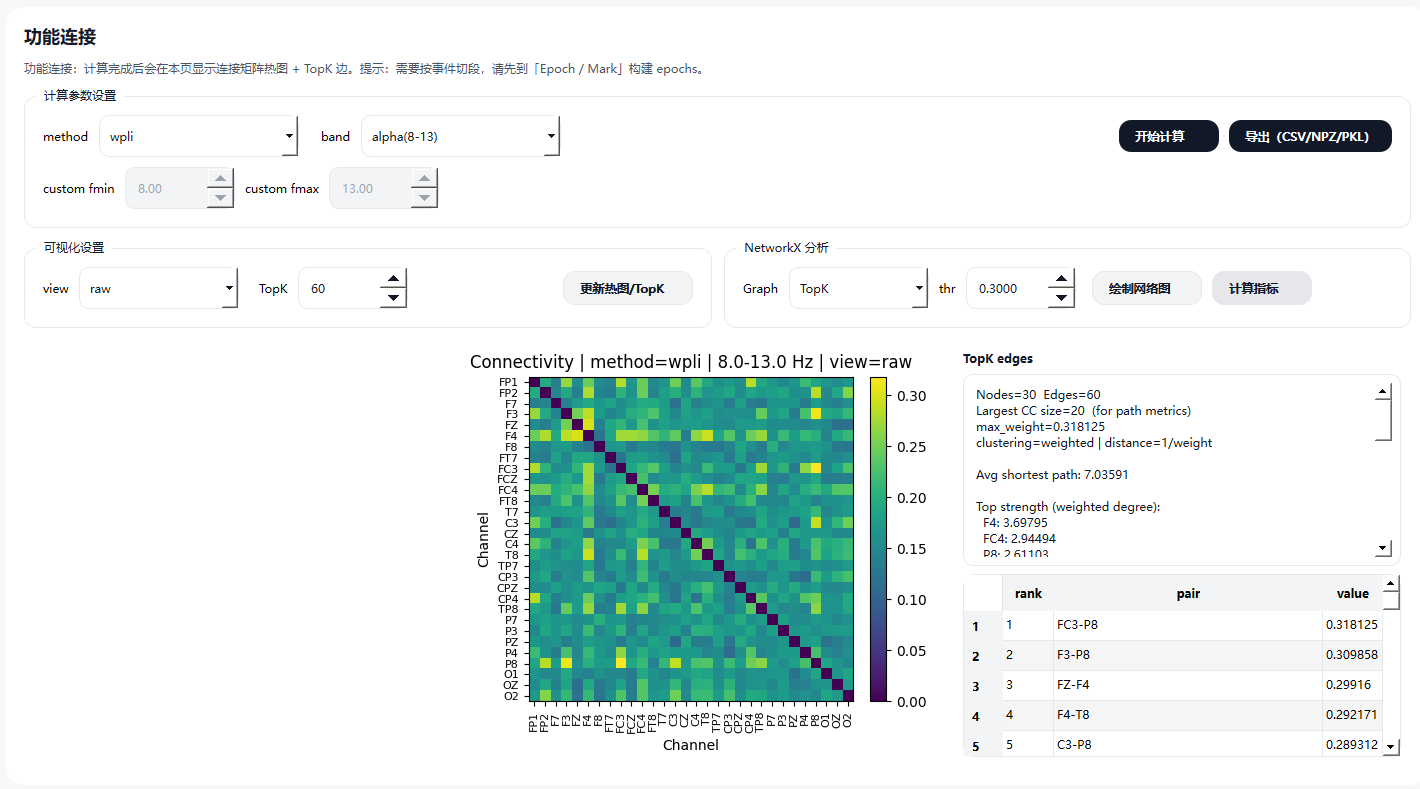
5.4.4 功能连接
该模块用于计算 EEG 通道间的功能连接关系,以刻画脑区之间的同步性与信息交互模式。
可选择预定义频段(δ、θ、α、β 等)或自定义频率范围
1) 连接指标
-
Coherence(相干性)
基于频域的线性相关度量,反映两个通道在特定频段内的幅值与相位一致性。
适用于分析稳定的振荡同步,但对体积传导较为敏感。
-
COH(Complex Coherence)
复数形式的相干性,保留相位差信息,可用于更精细的频域连接分析。
-
PLV(Phase Locking Value)
基于相位一致性的非线性指标,用于衡量信号间相位同步程度,对幅值变化不敏感,常用于事件相关或任务态分析。
-
PLI(Phase Lag Index)
基于相位滞后的连接指标,可在一定程度上抑制零时滞同步带来的体积传导影响,更强调具有方向性的相位关系。
-
wPLI(Weighted Phase Lag Index)
PLI 的加权改进版本,通过增强稳定相位滞后成分,提高对噪声的鲁棒性,在功能脑网络研究中被广泛采用。
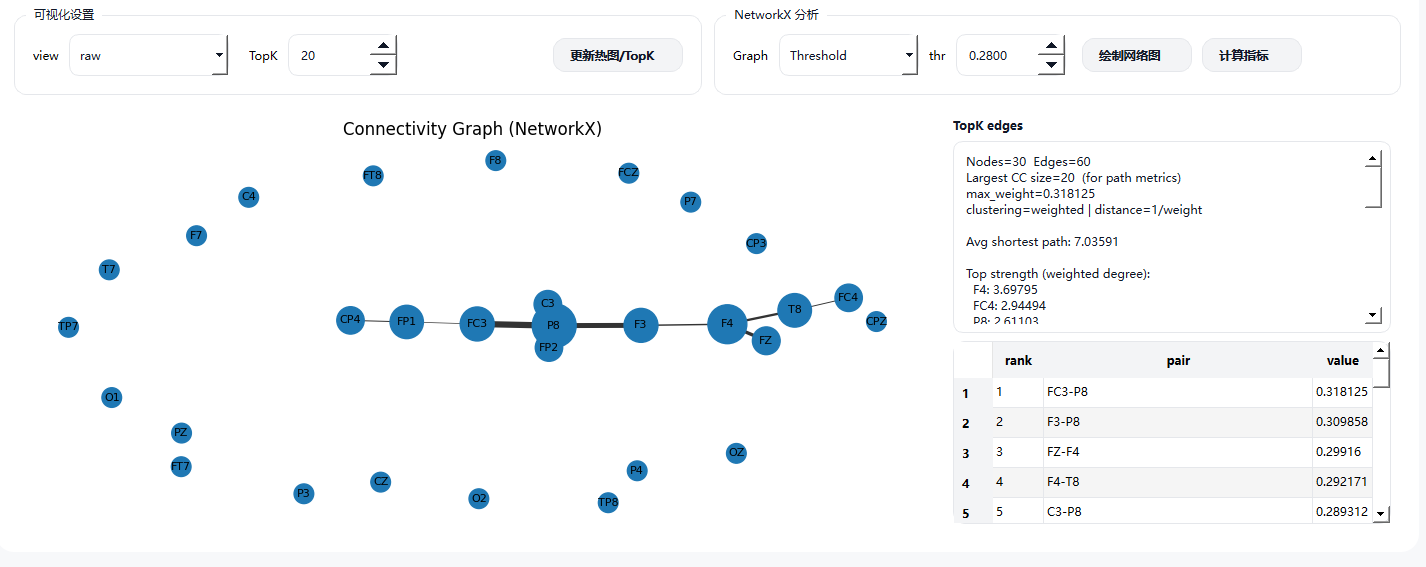
2) 连接矩阵与可视化
- 计算结果以通道 × 通道的连接矩阵形式表示
- 支持显示连接强度热图
- 可提取 Top-K 强连接边 以突出主要功能连接模式
3) 网络分析
- 基于连接矩阵构建功能脑网络
- 支持阈值或 Top-K 方式构图
- 可进一步计算图论指标(如节点度、聚类系数等),用于脑网络拓扑分析
4)日志(Log)
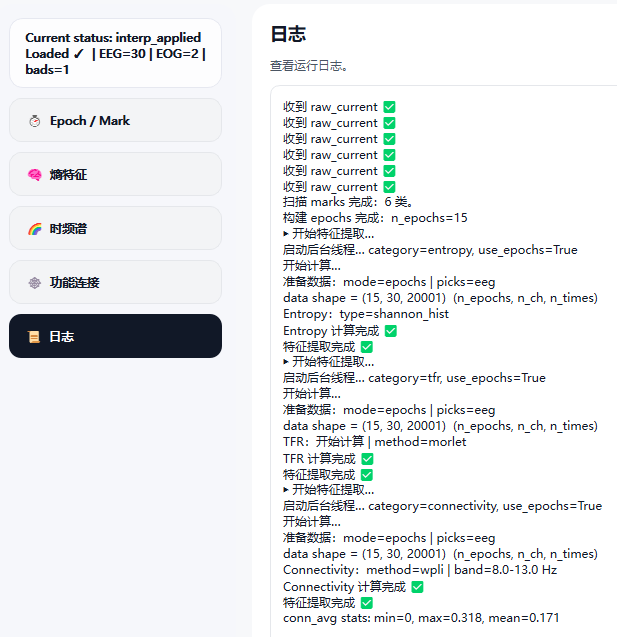
日志模块用于记录用户在软件中的关键操作与数据处理过程,以增强分析流程的可追溯性与结果的可复现性。
日志信息包括但不限于:
- 数据载入与状态更新(如 raw / epoch / 插值状态)
- 事件标记与 epoch 构建结果
- 各类特征计算的启动与完成状态
- 特征类别、所用方法及关键参数配置
- 数据维度信息(如 epoch 数、通道数、时间点数)
- 功能连接与网络分析的统计摘要
日志内容按时间顺序实时更新,可作为后续结果核查、参数复现及科研记录的重要参考。
6. fNIRS模块使用说明
本模块用于完成 fNIRS 数据的载入、预处理、事件切分及特征提取,支持基于血红蛋白浓度变化的任务相关分析。
6.1 载入数据(Load Data)
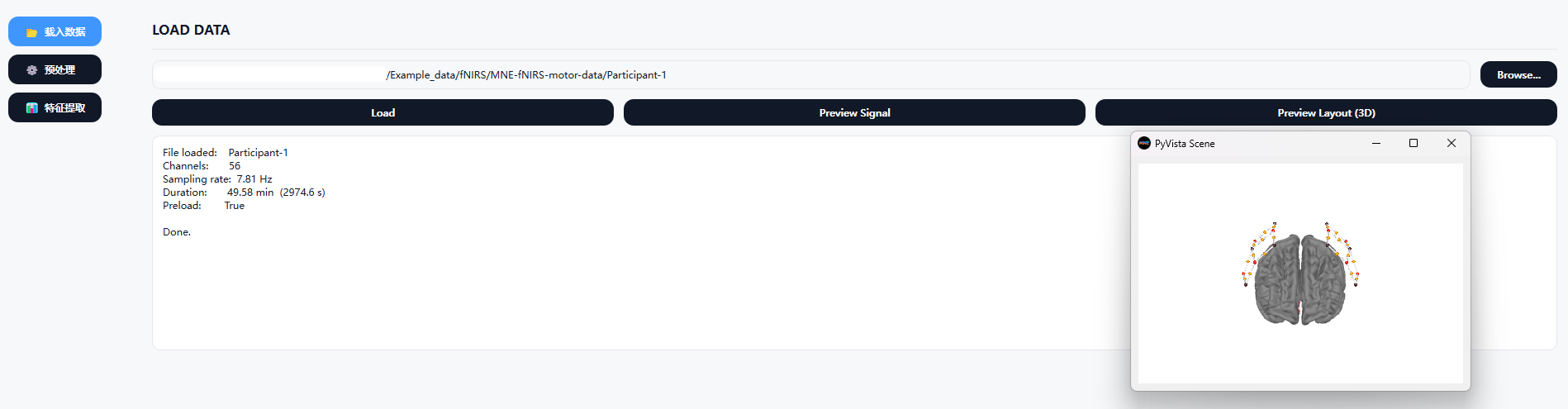
在 Load Data 页面中,选择并加载 fNIRS 数据文件。
系统将自动读取通道信息、采样率及记录时长,并初始化数据状态。
加载完成后,可预览原始信号及探头空间布局(3D),用于初步检查数据质量与通道分布。
6.2 预处理(Preprocessing)
6.2.1 转换、质量控制与血红蛋白计算
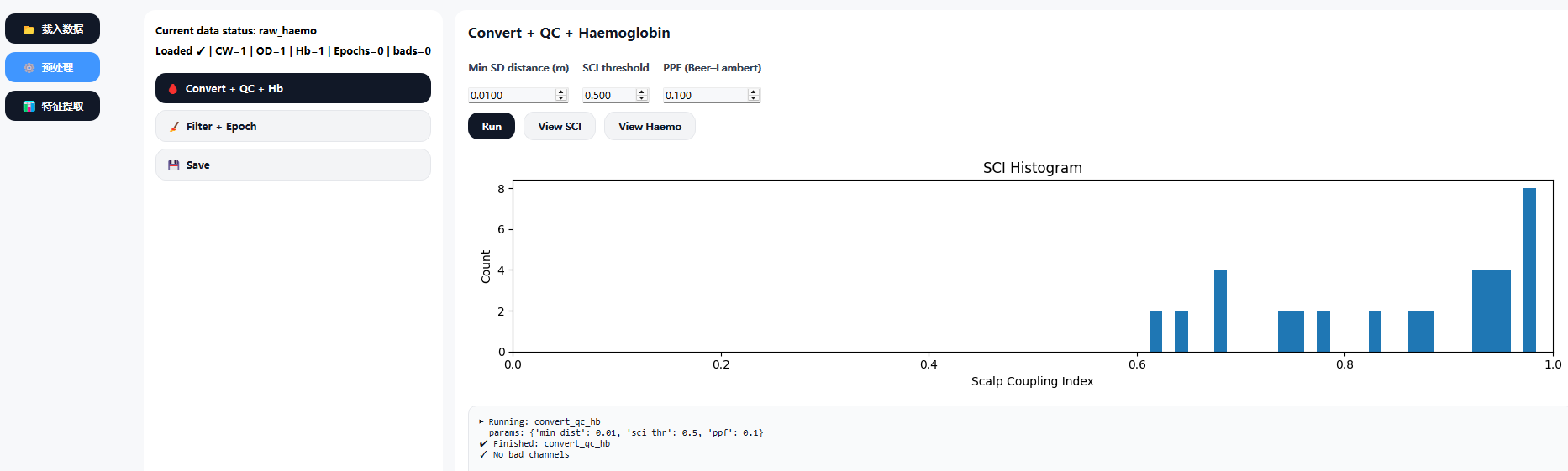
在 Convert + QC + Hb 页面中,系统依次执行:
- 光密度(Optical Density)转换
- 基于源--探测器距离与 SCI(Scalp Coupling Index)的质量控制
- 基于 Beer--Lambert 定律计算血红蛋白浓度变化(HbO / HbR)
处理过程中可查看 SCI 分布直方图与血红蛋白信号,以辅助评估通道质量。
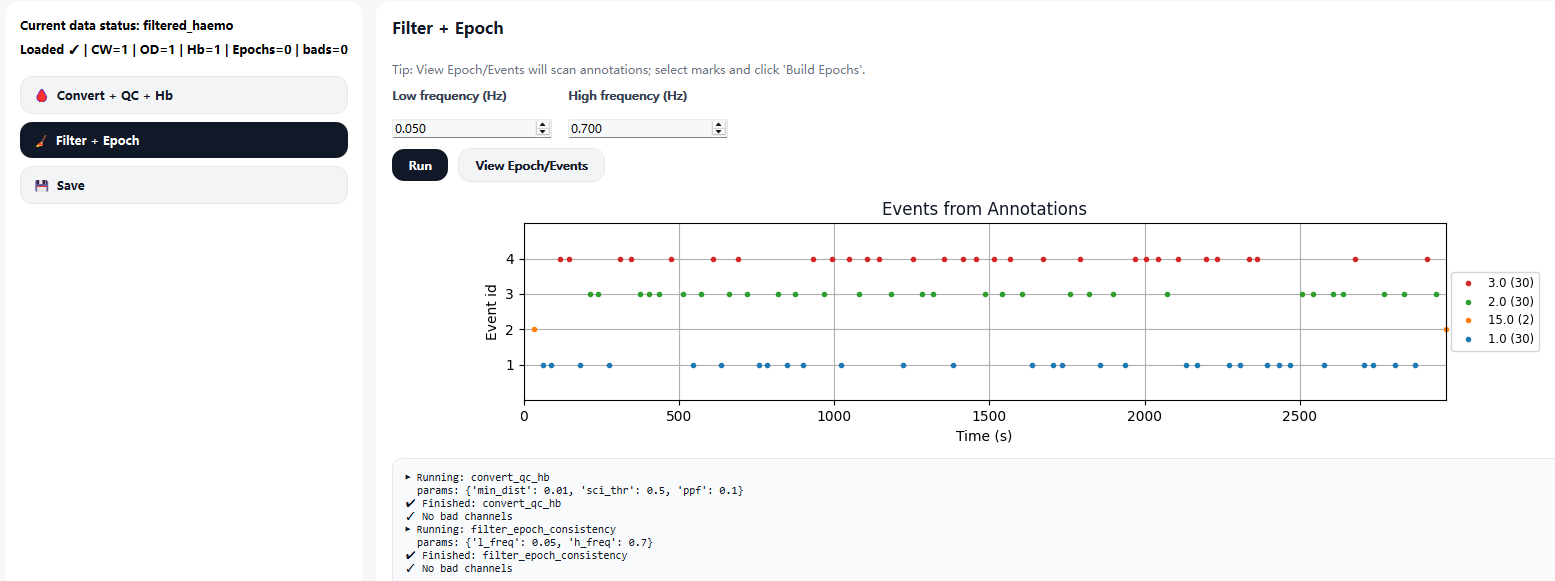
6.2.2 Filter + Epoch 可视化(Epoch Visualization)
该模块用于对 fNIRS 血红蛋白信号进行带通滤波,以抑制低频漂移与高频生理噪声,保留与任务相关的血氧动力学变化。
- 低频截止(Low frequency):用于去除缓慢基线漂移(如仪器漂移)
- 高频截止(High frequency):用于抑制心率、呼吸等高频生理噪声
滤波完成后,可通过 View Epoch/Events 按钮查看事件标记在时间轴上的分布,用于确认事件与信号时序的一致性。
在确认参数设置后,点击 Run 按钮即可执行滤波操作,结果将用于后续的事件切分与特征提取流程。
6.3 特征提取(Feature Extraction)
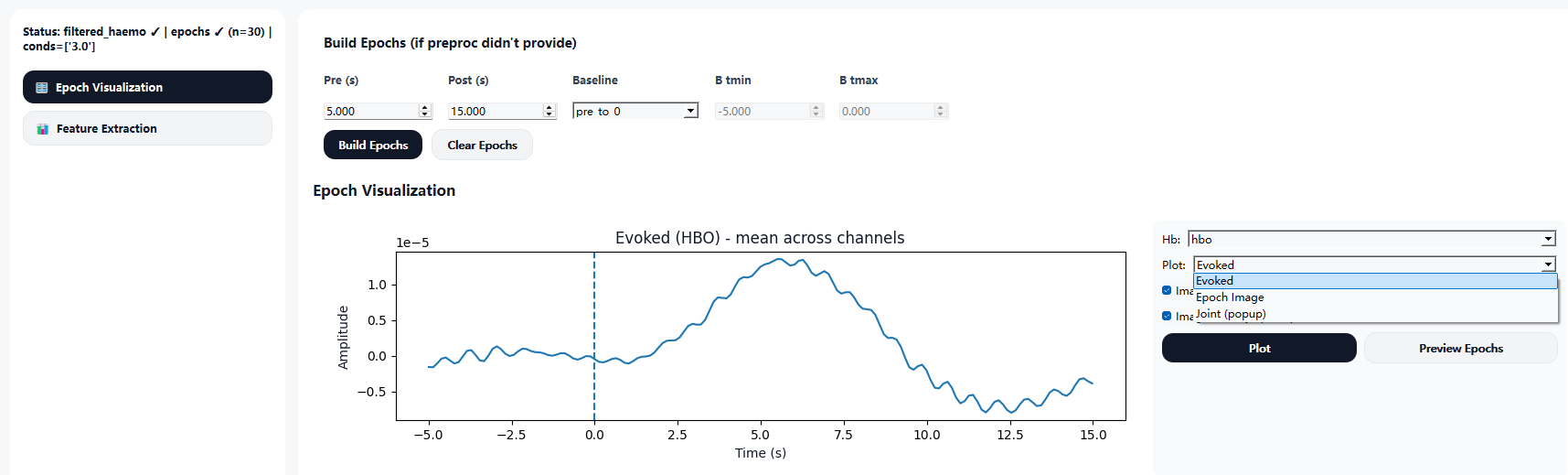
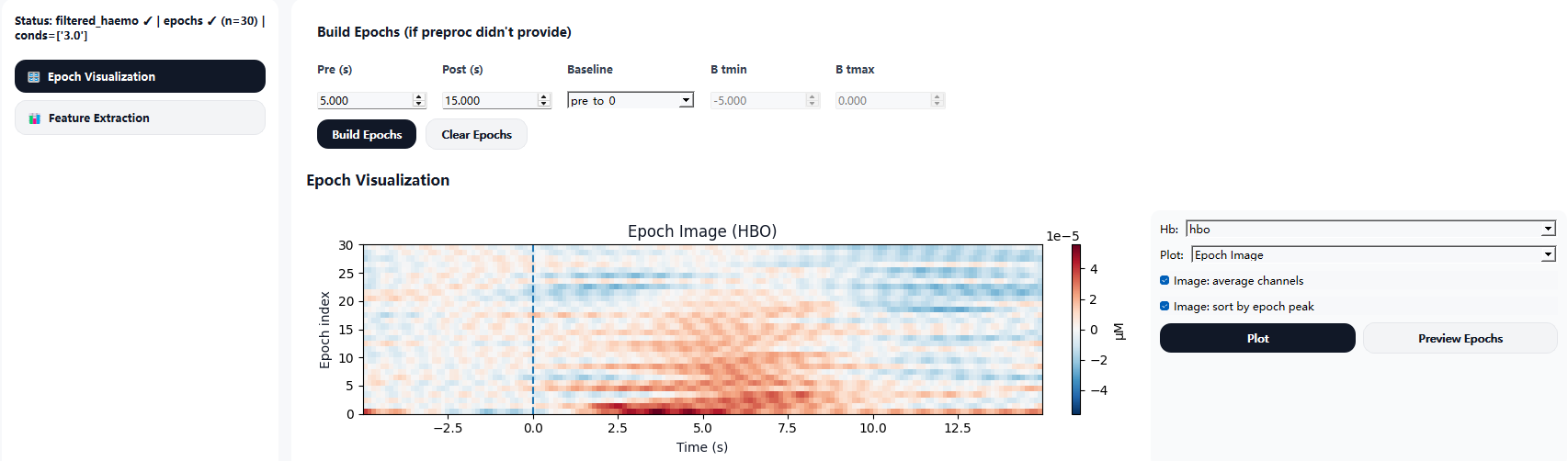
6.3.1 Epoch 可视化(Epoch Visualization)
在 Epoch Visualization 页面中,可构建 epochs ,并对epochs进行检查与展示:
- 支持选择血红蛋白类型(如 HbO)
- 提供 epoch 热图与联合视图(Joint plot)
- 显示基线位置与任务相关响应变化
该步骤有助于快速评估任务诱发的血氧动力学响应特征。
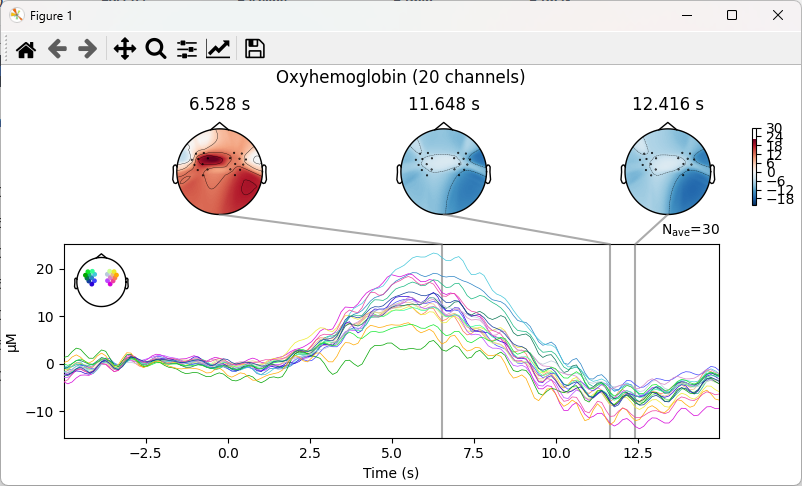
6.3.2 特征提取:Feature Extraction
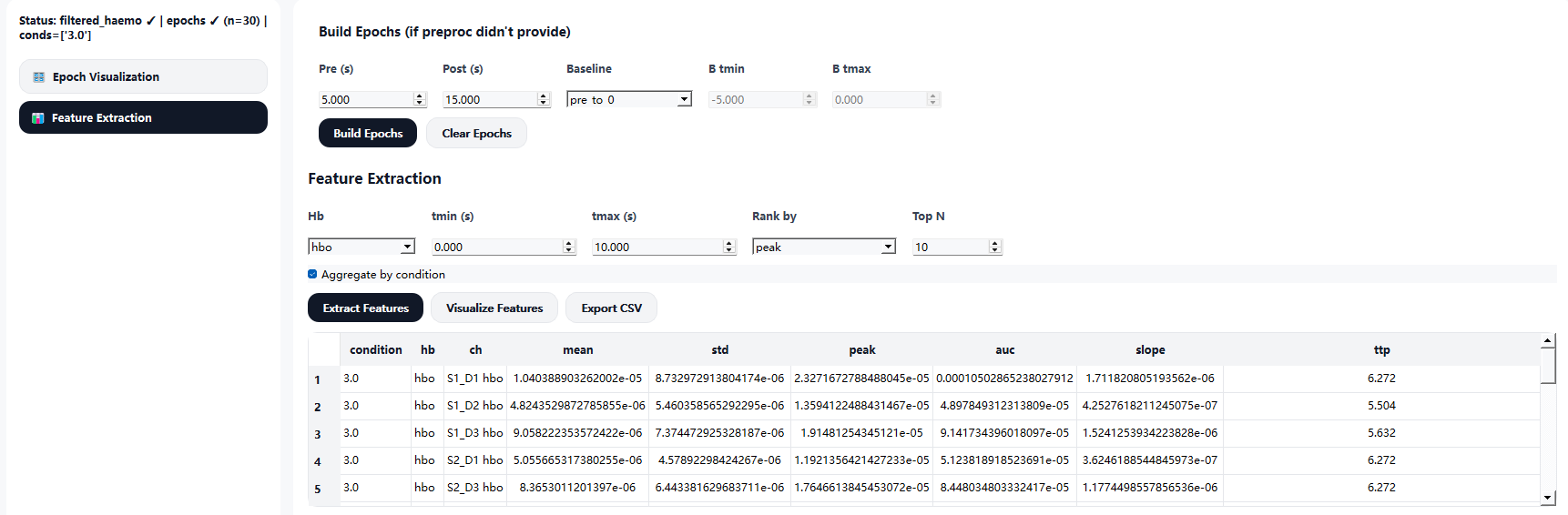
在 Feature Extraction 页面中,可从 epochs 中提取常用 fNIRS 特征:
- 支持按条件(condition)聚合
- 提取特征包括:
- 均值(mean)
- 标准差(std)
- 峰值(peak)
- 曲线下面积(AUC)
- 上升斜率(slope)
- 峰值时间(TTP)
特征结果以表格形式展示,并可导出为 CSV 文件,用于后续统计分析或机器学习建模。
6.4 数据保存(Save)
预处理及特征提取完成后,可将结果数据保存到本地。保存操作不会修改原始数据文件,确保数据安全与可追溯性。
7. sEMG模块使用说明
待更新......
8. 常见问题(FAQ)
Q1:双击软件没有反应 / 一闪而过
解决方法:
- 确认操作系统为 64 位 Windows
- 安装 Microsoft Visual C++ Redistributable 2015--2022(x64)
- 确保未删除
_internal文件夹
Q2:软件提示"某某库未安装"
这是软件内部依赖问题,并非用户操作错误。
解决方法:
- 请联系软件开发者获取更新版本
- 不需要、也无法在 exe 中执行
pip install
Q3:处理大文件时运行较慢
- 属于正常现象
- 建议:
- 使用性能更高的计算机
- 避免同时运行大量程序
9. 注意事项与已知限制
- 当前版本主要面向单文件交互式处理
- 暂不支持批量自动化处理
- 暂不支持 Linux / macOS 系统
10. 版本信息与联系方式
- 软件名称:NeuroSignal Preprocessor
- 当前版本:v1.0.0
- 发布日期:2025-12-31
如有问题或建议,请联系开发者。邮箱:wsh_cathy@163.com
© 2025 NeuroSignal Preprocessor