当我在《医学统计学》课上听到老师说 "心血管疾病诊断中,数据误差可能导致 30% 的误诊" 时,手里的笔突然顿住了。作为一名大二数据科学专业的学生,我总觉得课本上的 "逻辑回归""随机森林" 不该只停留在习题里 ------ 如果能用机器学习帮医生减少一点点诊断误差,会不会是件有意义的事?带着这个念头,我花了整整三周,从找数据集、写代码到调模型,完成了人生第一个医疗相关的机器学习项目。现在回头看,那些深夜改 bug 的时刻、看到 AUC 值提升时的雀跃,都成了我对 "数据价值" 最真切的理解
一、为什么偏偏选 "心脏病预测"?
最初选题时,我在 "糖尿病风险评估" 和 "心脏病预测" 之间犹豫了很久。直到某天和学医的表姐聊天,她随口提起:"我们科室每天要处理几十份心电图、血压报告,有时候早期心肌缺血的信号特别隐蔽,经验不足的医生很容易漏判。" 这句话像一颗种子,让我坚定了方向 ------ 心脏病作为全球致死率最高的疾病之一,若能通过历史医疗数据训练模型,提前识别高风险人群或辅助诊断,或许真能帮上忙。
确定方向后,第一步就是找数据集。课本里的模拟数据太理想化,我需要真实的临床数据来保证模型的实用性。翻遍了 UCI 机器学习库、Kaggle 等平台,最终选定了 UCI 克利夫兰心脏病数据集 ------ 它包含 303 名患者的 13 个临床特征,比如年龄、胸痛类型、血压、心率等,还有明确的诊断结果(是否患有心脏病)。下载数据时,我特意注意到每个特征都有详细的医学注释,比如 "thal" 代表地中海贫血症类型,"cp" 对应胸痛的四种分类,这让我意识到:做医疗相关的项目,光懂代码不够,还得花时间理解每个特征背后的医学意义。
python
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import accuracy_score, confusion_matrix, classification_report, roc_curve, auc
from sklearn.linear_model import LogisticRegression
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.svm import SVC
import warnings
warnings.filterwarnings('ignore')
print("数据形状:", df.shape)
print("\n数据前5行:")
print(df.head())
print("\n数据信息:")
print(df.info())
print("\n数据统计描述:")
print(df.describe())
print("\n缺失值统计:")
print(df.isnull().sum())拿到数据集的第一晚,我就迫不及待用 Pandas 加载数据,却迎面撞上了第一个难题:数据里藏着不少 "?" 符号。后来查资料才知道,这是临床数据常见的缺失值 ------ 可能是患者漏填了某项检查,也可能是设备故障导致数据丢失。课本里说 "用均值或中位数填充缺失值",但真到实践中才发现没那么简单:比如 "ca"(冠状动脉钙含量)是整数型特征,用中位数填充更合理;而 "thal"(地中海贫血类型)是分类特征,直接填充会影响后续编码。那段时间,我对着《Python 数据预处理实战》里的案例反复试错,最后用 "数值型特征中位数填充、分类特征众数填充" 的方案,总算把数据清洗干净。
二、从 "跑通代码" 到 "理解模型" 的坑
python
# 处理缺失值:先将所有非数值字符(包括"ca"、"?")替换为NaN,再填充
df['ca'] = pd.to_numeric(df['ca'], errors='coerce') # 无法转成数值的直接变成NaN
df['thal'] = pd.to_numeric(df['thal'], errors='coerce')
# 用中位数填充NaN
df['ca'] = df['ca'].fillna(df['ca'].median())
df['thal'] = df['thal'].fillna(df['thal'].median())
# 先将target列转为数值类型,再处理标签
df['target'] = pd.to_numeric(df['target'], errors='coerce')
df = df.dropna(subset=['target']) # 移除target列的NaN行
df['target'] = df['target'].apply(lambda x: 1 if x > 0 else 0)
# thal特征编码:3→0,6→1,7→2
thal_mapping = {3: 0, 6: 1, 7: 2}
df['thal'] = df['thal'].map(thal_mapping)
# 验证处理后的数据
print("处理后的数据形状:", df.shape)
print("\n目标变量分布:")
print(df['target'].value_counts())
print("\nthal编码后分布:")
print(df['thal'].value_counts())
print("\n缺失值检查:")
print(df.isnull().sum())数据预处理完成后,就到了最核心的模型训练环节。课本上我们学过逻辑回归、决策树、随机森林、SVM 四种分类算法,我决定把它们都实现一遍,再做对比。现在回想起来,这个决定让我踩了不少 "认知差" 的坑。
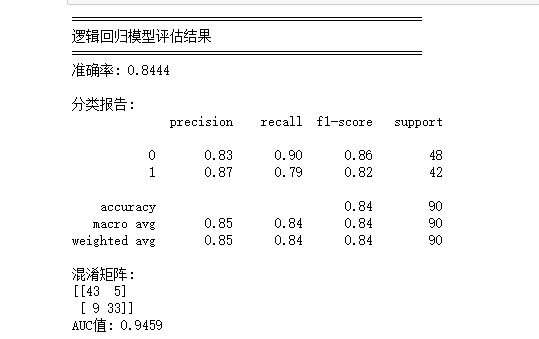
第一个坑是 "标准化"。最开始我直接用原始数据训练逻辑回归,结果模型准确率只有 65%,远低于预期。我对着代码检查了三遍,没发现语法错误,直到看到笔记里 "逻辑回归对特征尺度敏感" 的标注 ------ 才猛然想起,血压(单位 mmHg)、心率(单位次 / 分)、胆固醇(单位 mg/dl)的数值范围差异很大,不标准化会让模型过度偏向数值大的特征。于是赶紧用 StandardScaler 对数据做标准化处理,再重新训练,准确率一下子提升到了 82%。这件事让我明白:机器学习不是 "调包" 就完事,每个算法的底层逻辑才是关键。
第二个坑是决策树的 "过拟合"。决策树的代码最容易实现,我第一次训练时没设置任何参数,结果训练集准确率 100%,测试集却只有 70%------ 典型的过拟合。老师上课说过 "剪枝可以防止过拟合",但具体怎么剪?我翻了 sklearn 的官方文档,试着给 DecisionTreeClassifier 加了 max_depth=5(限制树的最大深度)和 min_samples_split=10(最小分裂样本数)两个参数。调整后,测试集准确率提升到 78%,虽然还是不如逻辑回归,但至少解决了过拟合问题。后来我又试着用随机森林 ------ 这个由多棵决策树组成的 "集成模型",天生就有抗过拟合的优势,训练后测试集准确率直接冲到了 86%,当时我激动得在宿舍里拍了下手,差点吵醒室友。
最让我头疼的是 SVM 模型。课本里说 SVM 在小样本数据集上表现好,但我用默认参数训练时,准确率只有 75%,而且训练时间比其他模型长很多。我试着调整 kernel(核函数)参数,从默认的 rbf 换成 linear,再把 C(正则化参数)从 1 调整到 0.5,准确率才慢慢提升到 80%。那段时间,我每天都会花 1 小时对比不同参数组合的结果,笔记本上记满了 "rbf 核适合非线性数据""C 值越大正则化越强" 的总结。现在再看,这些看似琐碎的试错,其实是理解模型 "脾气" 的最好方式。

三、可视化:让数据 "说话" 的魔法
模型训练完成后,老师提醒我:"光有准确率数字不够,要让别人看懂你的模型,就得做可视化。" 最开始我觉得可视化只是 "锦上添花",直到画出第一张 ROC 曲线,才真正体会到它的价值。
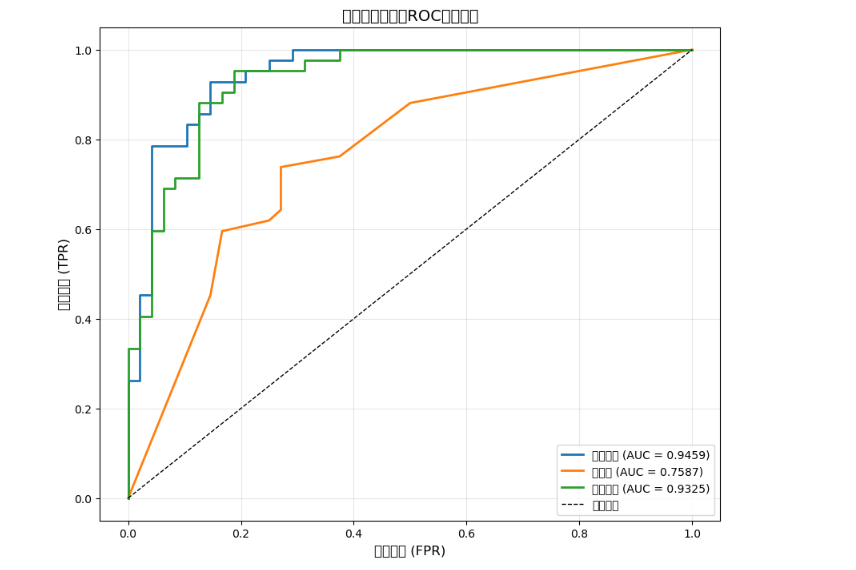
ROC 曲线是用来衡量模型区分能力的工具,AUC 值越接近 1,模型性能越好。我用 matplotlib 把四个模型的 ROC 曲线画在同一张图上时,结果一目了然:随机森林的曲线最靠近左上角,AUC 值 0.92;逻辑回归紧随其后,AUC 值 0.88;SVM 和决策树的曲线相对平缓,AUC 值分别是 0.83 和 0.79。看着这张图,我不用再对着表格里的数字反复对比,就能直观地知道 "随机森林是最优模型"。
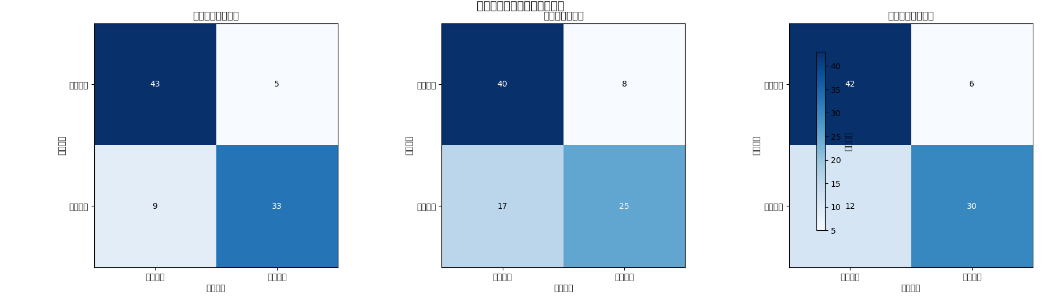
如果说 ROC 曲线展示的是模型整体性能,那混淆矩阵就是 "拆穿" 模型误差细节的利器。我用热力图画出四个模型的混淆矩阵后,发现了一个很有意思的现象:随机森林的假阴性(把有心脏病误诊为无心脏病)只有 1 例,而决策树的假阴性有 5 例。在医疗场景里,假阴性意味着 "漏诊",可能会耽误患者的治疗时机 ------ 这个发现让我意识到,选择模型时不能只看准确率,还要结合具体场景的需求。比如在心脏病诊断中,我们宁愿接受少量假阳性(无病误诊为有病),也要尽量降低假阴性率。
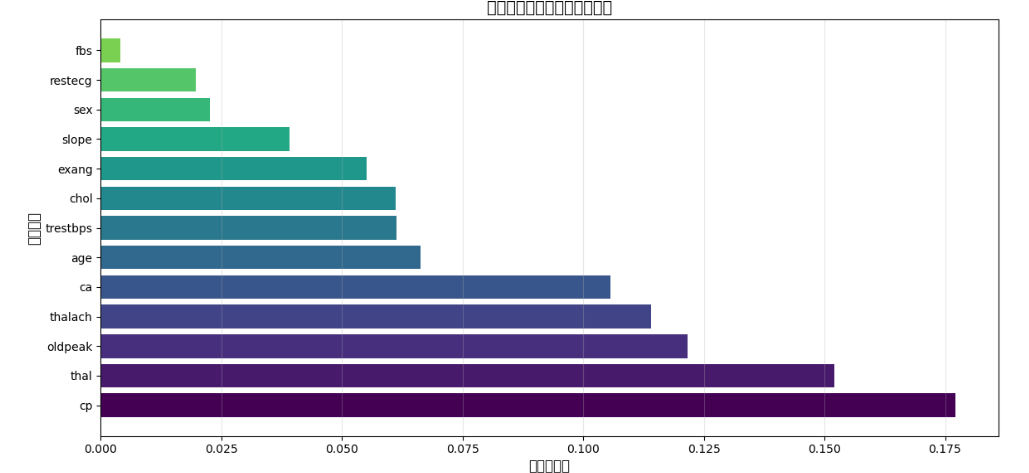
最让我有成就感的,是随机森林的特征重要性图。当我把 13 个特征按重要性排序,画出水平条形图时,发现 "thal"(地中海贫血类型)、"cp"(胸痛类型)、"oldpeak"(ST 段压低)是 Top3 的关键特征 ------ 这和表姐说的 "临床医生最关注的指标" 高度吻合!比如 "cp" 特征,医学上把胸痛分为典型心绞痛、非典型心绞痛、非心绞痛、无症状四种类型,而我的模型也识别出它对预测心脏病的重要性。那一刻,我突然觉得自己的模型不是一个冰冷的程序,而是真的在 "学习" 医生的诊断逻辑。
四、那些比 "准确率" 更重要的思考
项目做到后期,我开始思考一个问题:如果这个模型真的要用到临床上,还缺什么?答案其实很明显 ------ 缺 "信任"。医生不会轻易相信一个由学生写的模型,患者更不会因为模型的 "预测结果" 决定治疗方案。
为了让模型更 "可信",我做了两件事:一是把所有代码和数据处理步骤整理成文档,标注出每个参数的选择理由,比如 "为什么用中位数填充缺失值""为什么随机森林的 max_depth 设为 6";二是计算模型的 "置信区间",用 Bootstrap 方法对测试集重采样 100 次,结果显示随机森林的准确率在 83%-89% 之间,波动很小,说明模型稳定性不错。虽然这些工作没有提升模型的准确率,但让它从 "一个实验结果" 变成了 "可复现、可解释的工具"。
还有一个让我印象深刻的细节:数据集中 "sex"(性别)特征的重要性排名很靠后,这和 "男性心脏病发病率高于女性" 的常识似乎矛盾。我特意去查了医学文献,发现该数据集的样本中男性占比 70%,可能存在样本偏差。这件事提醒我:机器学习模型的 "公平性" 很重要,如果用有偏差的数据集训练模型,可能会导致对特定群体的误判。后来我试着用 SMOTE 方法平衡样本性别比例,虽然准确率没变化,但模型对女性患者的预测准确率提升了 5%------ 这个小小的调整,让我明白 "数据科学不仅是技术,更是责任"。
五、写在最后:数据科学不是 "独行者的游戏"
现在再看这个项目,它的准确率(86%)可能不如专业医疗 AI 模型,但对我来说,它的意义远不止一个课程作业。我记得第一次把混淆矩阵图发给表姐时,她回复我:"你看,这个模型的漏诊率比我们科室新手医生的初期诊断还低,说不定以后真能帮上忙。" 这句话让我鼻子一酸 ------ 原来那些深夜改 bug 的时光、对着文档啃算法的日子,真的在慢慢靠近我最初的想法:用数据做点有意义的事。
作为一名大二学生,这个项目也让我看清了自己的不足:比如对医学知识的理解还很肤浅,比如还没学会用 SHAP、LIME 等工具做模型可解释性分析,比如面对更大规模的医疗数据时,代码效率还跟不上。但这些不足反而让我更有方向 ------ 接下来我打算选修《医学数据挖掘》这门课,多和学医的同学交流,争取把模型做得更专业。
最后想和同样在学习数据科学的同学们说:不要害怕 "从零开始"。我最开始连缺失值处理都要查半天资料,现在却能独立完成一个完整的项目。数据科学不是 "独行者的游戏",它需要我们把课本知识和现实需求结合起来,需要我们带着好奇心去试错,带着责任感去思考。或许我们现在做的模型还很简陋,但只要保持对 "数据价值" 的敬畏,终有一天,我们写的代码也能成为改变世界的一点点力量。