GPU工具分类
实话实说,暂时只有英伟达的GPU才能实现比较方便的基因组分析集成化解决方案,其他卡还需要努力呀,或者需要商业公司或学术团体的努力开发呀!FPGA等这种专用卡的解决方案也是有的,比如某测序仪厂家,某大厂,专门做加速方案的提供商,以及CPU加速方案提供商等。
这里,就先看下普通人可及的英伟达啦,AI的普及,以及黑神话的爆火,让大家都有了相对较好的卡,有的实验室新配服务器也加装了一般的GPU算力卡,除了做AI,也可以考虑下做下加速分析啦!当然,如我这种,没有大显存的卡,没有大显存的服务器,我们依然可以租卡呀,专门租gpu的公司可以成本低至一块多一小时,也是极划算的啦!当然,自己买张便宜的卡一举多得也不错啦,又多了一个上装备的理由啦!当然,禁运和制裁是个问题,应该不跑大项目也不涉及这些,国货当自强呀!
- Clara Parabricks
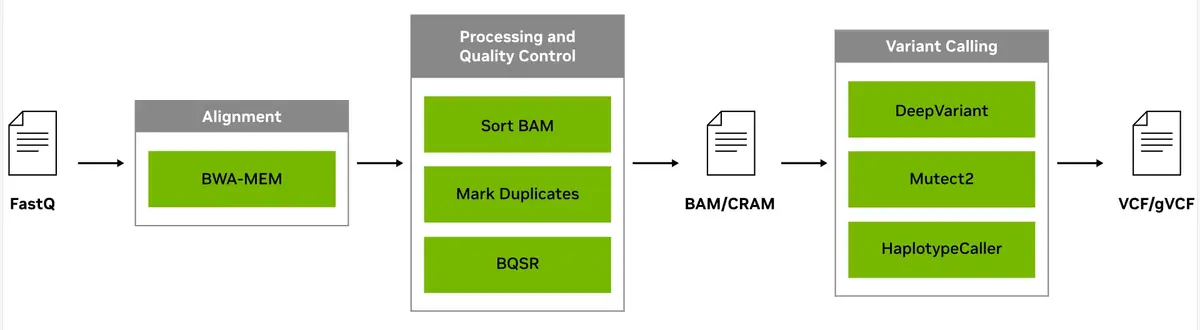
短读段测序可生成短 DNA 序列,并因速度快且经济实惠而受到重视。Clara Parabricks是英伟达基于GPU卡开发用于加速call变异的工具套件,支持GATK haplotypecaller和deepvariant 2种call 变异的方式,相比原版速度有大幅提升,从而有助于快速分析短读段测序数据。可用于处理来自 Element、Illumina、MGI、Singular、Thermo Fisher 和 Ultima 测序仪的数据。从v4.0开始,学术机构用户可免费使用。
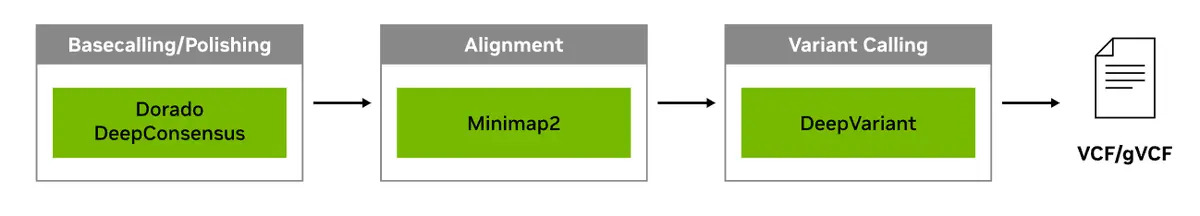
- 长读段测序有助于识别结构变异,并可用于根据多个样本来组合基因组,从而推动群体遗传多样性的研究,可用于处理来自 Oxford Nanopore 和 PacBio 长读段测序仪的数据。
- NVIDIA RAPIDS™ 可加速单细胞数据处理和可视化工作流,并具有与 scverse Python 库类似的可用性模型。cunnData 为 GPU 提供了 AnnData 对象的超小型、轻量级版本,有助于快速操作和处理数据,RAPIDS cuGraph 和 cuML 库随后则会开展下游分析工作。

暂时手上只有基因组的数据,于是先测试下基因组的分析啦!
先用自己的3050 Ti Laptop(4G VRAM)测试
先说下结论安装是可以成功的,报错也是会的 ,当然,分析小基因组也是极好的,话说回来,小基因组也就不需要加速啦!我是Win11系统,已经装了WSL2子系统,docker和驱动已经装好,以上这些这里不表啦!当然,最新的驱动已经可以使用RAM当显存,前提是牺牲速度,所以,我的笔记本加到64G RAM也应该可以跑得动的,慢速的。
根据官方文档安装是个小挑战,众所周知的原因啦,费了好久,几乎要放弃时,在github上找到了新的方法,这里分享一下!
Request nvidia-docker2 debian download not from repository · Issue #635
安装nvidia-docker2
git clone下载,更方便,避免官方网站步骤的各种报错,亲测可以成功!
LOCALDIR=/var/lib/nvidia-docker-repo
mkdir -p $LOCALDIR && cd $LOCALDIR
git clone -b gh-pages https://github.com/NVIDIA/libnvidia-container.git
git clone -b gh-pages https://github.com/NVIDIA/nvidia-container-runtime.git
git clone -b gh-pages https://github.com/NVIDIA/nvidia-docker.git
# ubuntu系统,应该是18.04以上通用
sudo tee /etc/apt/sources.list.d/nvidia-docker.list <<< \
"deb file://$LOCALDIR/libnvidia-container/stable/ubuntu18.04/amd64 /
deb file://$LOCALDIR/nvidia-container-runtime/stable/ubuntu18.04/amd64 /
deb file://$LOCALDIR/nvidia-docker/stable/ubuntu18.04/amd64 /"
# 更新
sudo apt-key add $LOCALDIR/nvidia-docker/gpgkey
sudo apt-get update
sudo apt-get install -y nvidia-container-toolkit
sudo apt-get install -y nvidia-docker2docker镜像修改
sudo vi /etc/docker/daemon.json
# 修改为以下内容:
{
"runtimes": {
"nvidia": {
"path": "nvidia-container-runtime",
"runtimeArgs": []
}
},
"registry-mirrors": [
"https://docker.1panel.live",
"https://hub.rat.dev"
]
}
# 参考自:Docker更换镜像源 https://patzer0.com/archives/configure-docker-registry-mirrors-with-mirrors-available-in-cn-mainland使用测试
想了下,参考基因组应该是不能切分的,所以官方宣称的人类基因组需要16G显存的要求应该是真实的和合理的。
docker run --gpus all --rm --volume $(pwd):/workdir \
--volume $(pwd):/outputdir \
nvcr.io/nvidia/clara/clara-parabricks:4.3.2-1 \
pbrun fq2bam \
--ref /workdir/reference/felCat8.fa.gz \
--in-fq /workdir/cat_10000_1.fq.gz /workdir/cat_10000_2.fq.gz \
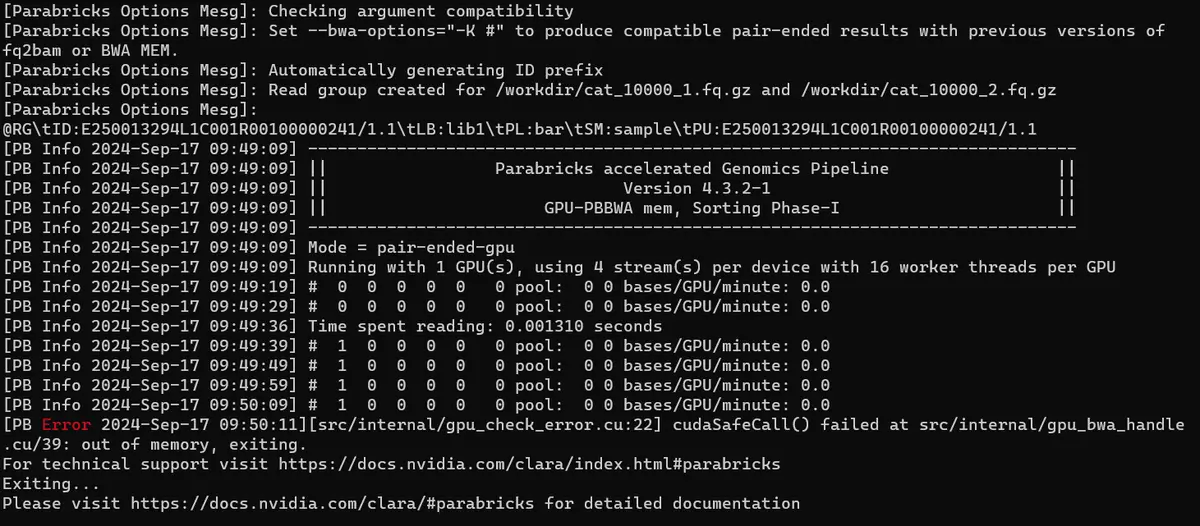
--out-bam /outputdir/fq2bam_output.bam报错信息如下:
分染色体运行
http://hpc.ncpgr.cn/app/098-parabricks/#fen-ran-se-ti-yun-xing
这里还有个高校的经验介绍,部分基因组较大或深度较深的数据,运行 pbrun haplotypecaller 时可能会出现显存不够的报错 Out of memory,此时可以分染色体来跑,最后再合并。
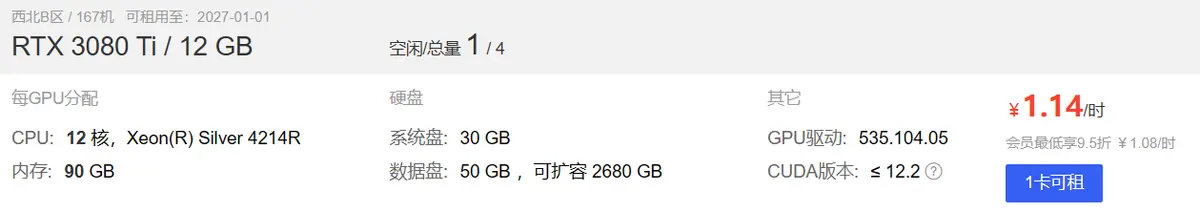
租卡测试
看起来价格很诱人的样子,有空了再实测速度和感受怎样,欢迎交流相关经验。