在单细胞RNA测序分析中,可视化是揭示数据背后生物学模式的关键步骤。Seurat作为单细胞分析的主流工具,虽然功能强大,但在可视化方面仍有提升空间。今天就为大家介绍Seurat的增强包------SeuratExtend,它能让你的单细胞图表瞬间提升一个档次!
单细胞转录组、空间转录组、转录组(mRNA-seq、miRNA-seq、lncRNA-seq、circRNA-seq)、宏基因组、16S扩增子、基因组、ChIP-seq、等组学、TCGA/GEO数据挖掘、影像组、病理组、研究学习免费交流群:18983376561
什么是SeuratExtend?
SeuratExtend是基于Seurat开发的扩展包,专注于增强可视化功能。它保留了Seurat原有的使用习惯,同时添加了大量自定义参数,让你的图表既能满足科研需求,又能拥有 publication 级别的美观度。
注意:下边的例子基于SeuratExtend 1.2.3版本
核心功能亮点
增强版降维散点图(DimPlot2)
告别Seurat中DimPlot(离散变量,如细胞类型,分组等)和FeaturePlot(连续变量,如基因表达水平等)的区分,DimPlot2一键搞定所有类型变量的可视化:
r
library(Seurat)
library(SeuratExtend)
# 基础用法 离散分类
DimPlot2(pbmc)
# 同时展示多种变量
DimPlot2(pbmc, features = c("cluster", "orig.ident", "CD14", "CD3D"))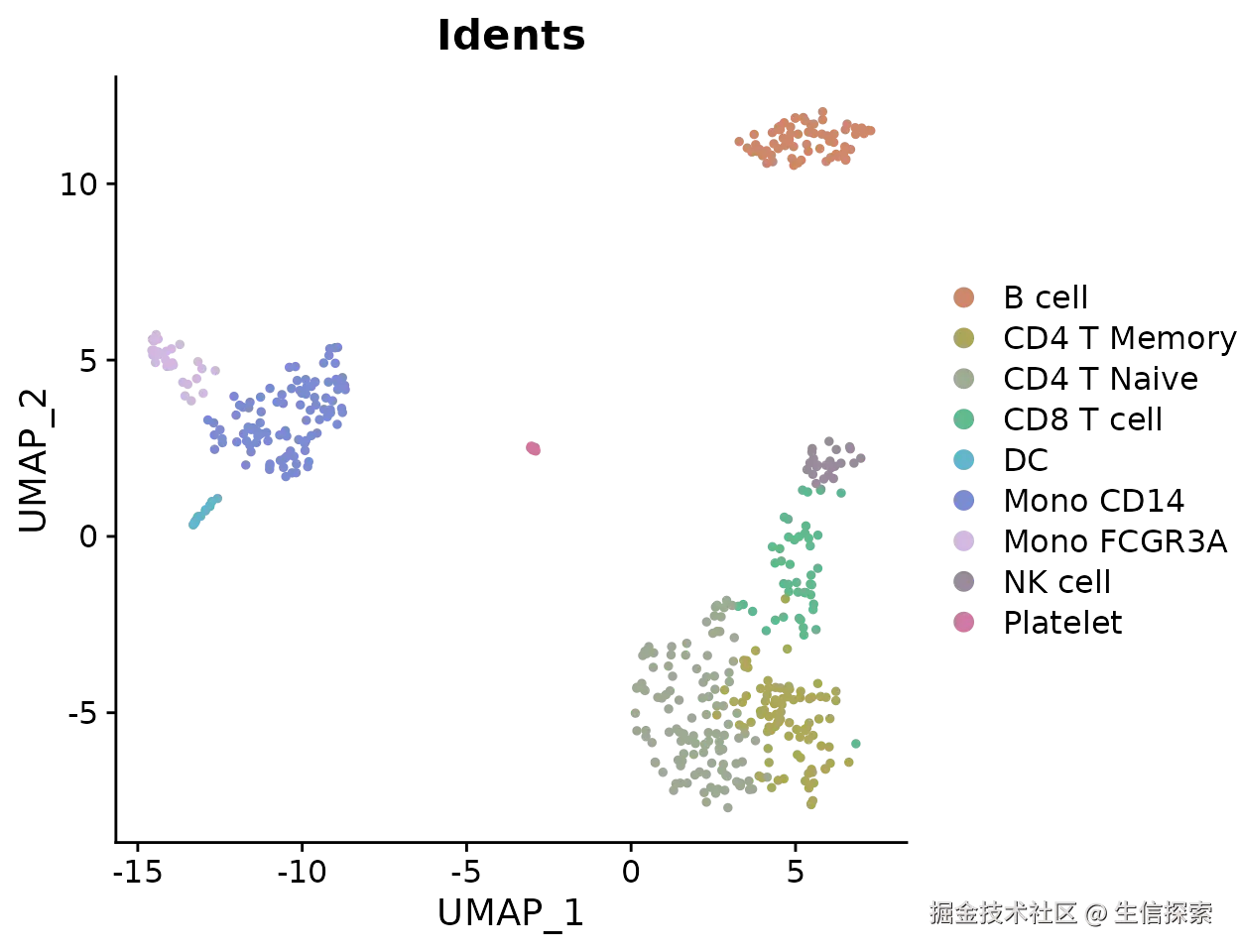
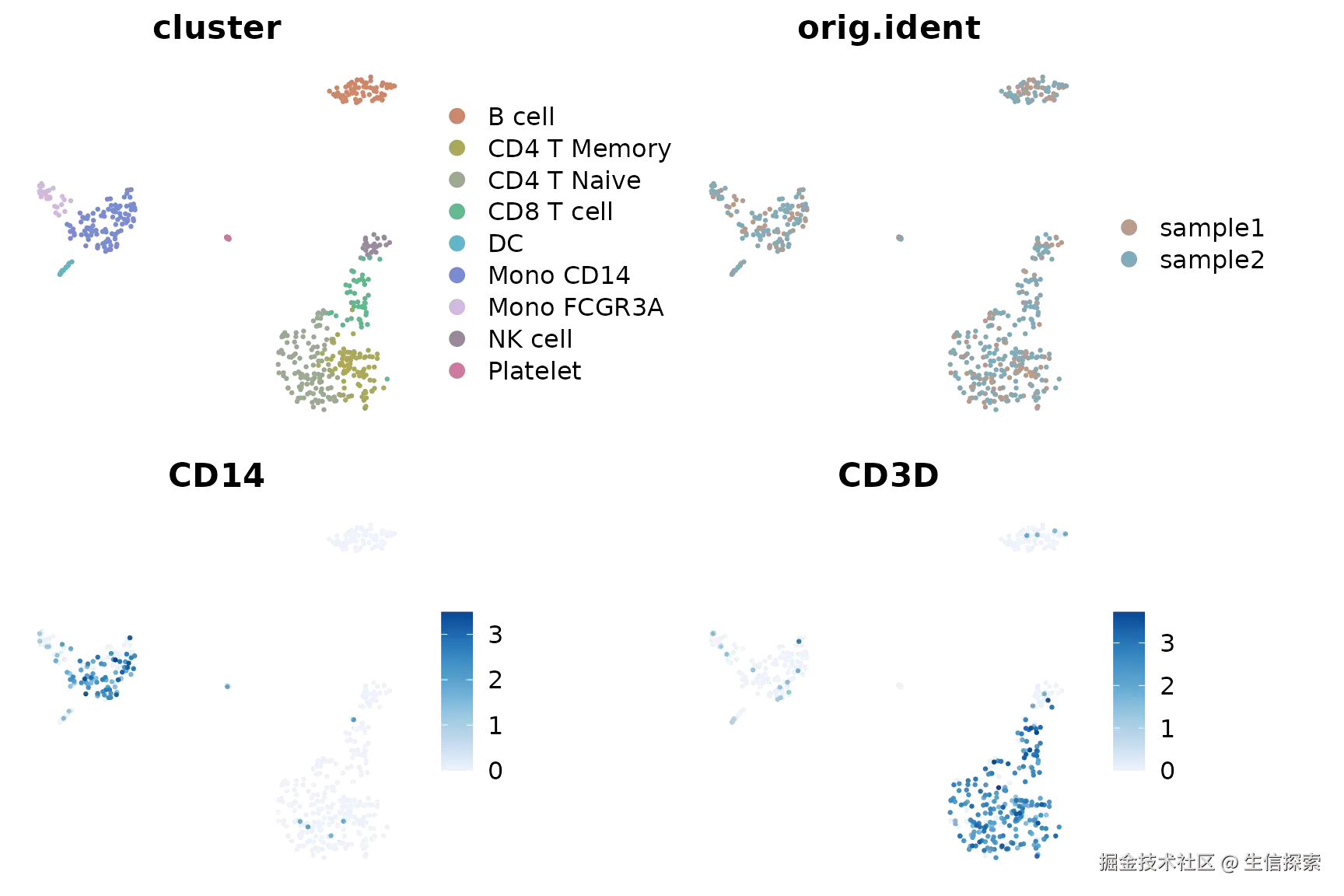
特色功能:
- 自动识别变量类型,无需切换函数
- 支持高亮特定细胞群(
cells.highlight参数) - 缩小 UMAP 坐标轴箭头(
p + theme_umap_arrows())
FeaturePlot3
新增三特征同时展示(FeaturePlot3),通过RGB或RYB混色原理呈现三个基因的表达分布
r
FeaturePlot3(pbmc, color = "ryb", feature.1 = "CD3D", feature.2 = "CD14", feature.3 = "CD79A", pt.size = 0.5)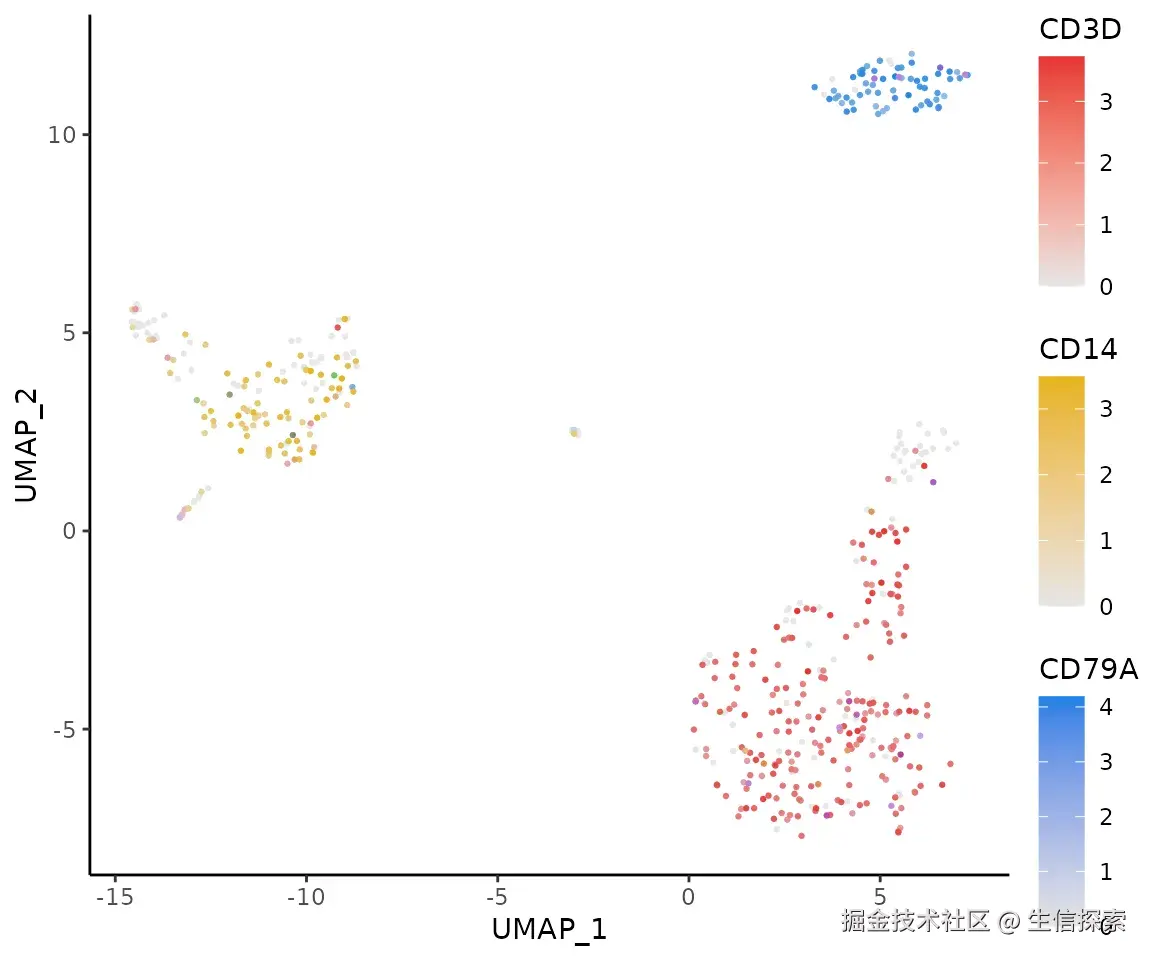
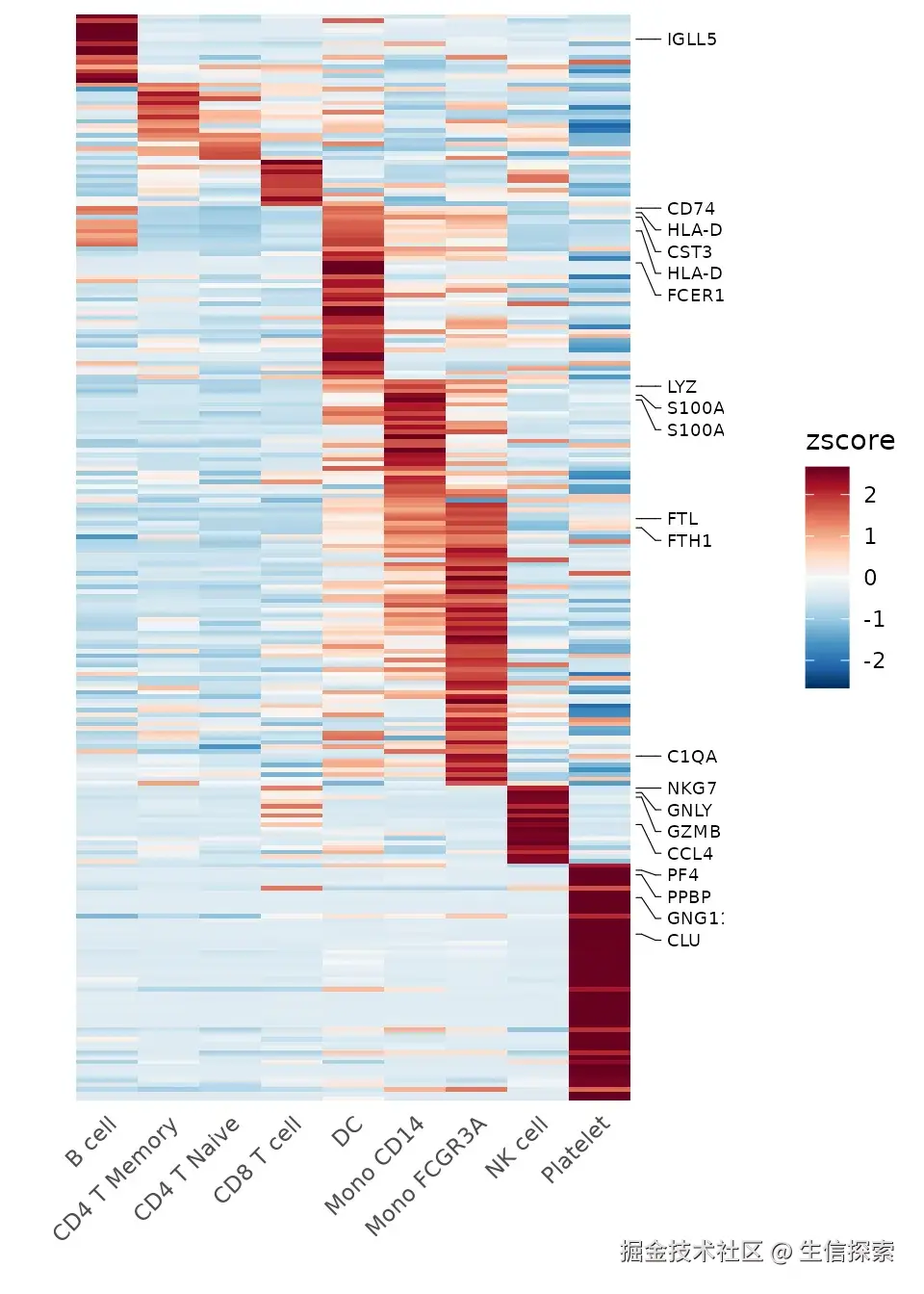
高颜值热图(Heatmap)
热图是展示大量基因在不同集群中表达模式的利器,SeuratExtend的Heatmap函数让颜色搭配更专业:
r
# 计算变量基因的z-score
genes <- VariableFeatures(pbmc)
toplot <- CalcStats(pbmc, features = genes, method = "zscore")
# 基础热图
Heatmap(toplot, lab_fill = "zscore")
# 只标注部分基因
toplot2 <- CalcStats(pbmc, features = genes[1:500], method = "zscore", order = "p")
Heatmap(toplot2, lab_fill = "zscore", feature_text_subset = genes[1:20], expand_limits_x = c(-0.5, 11))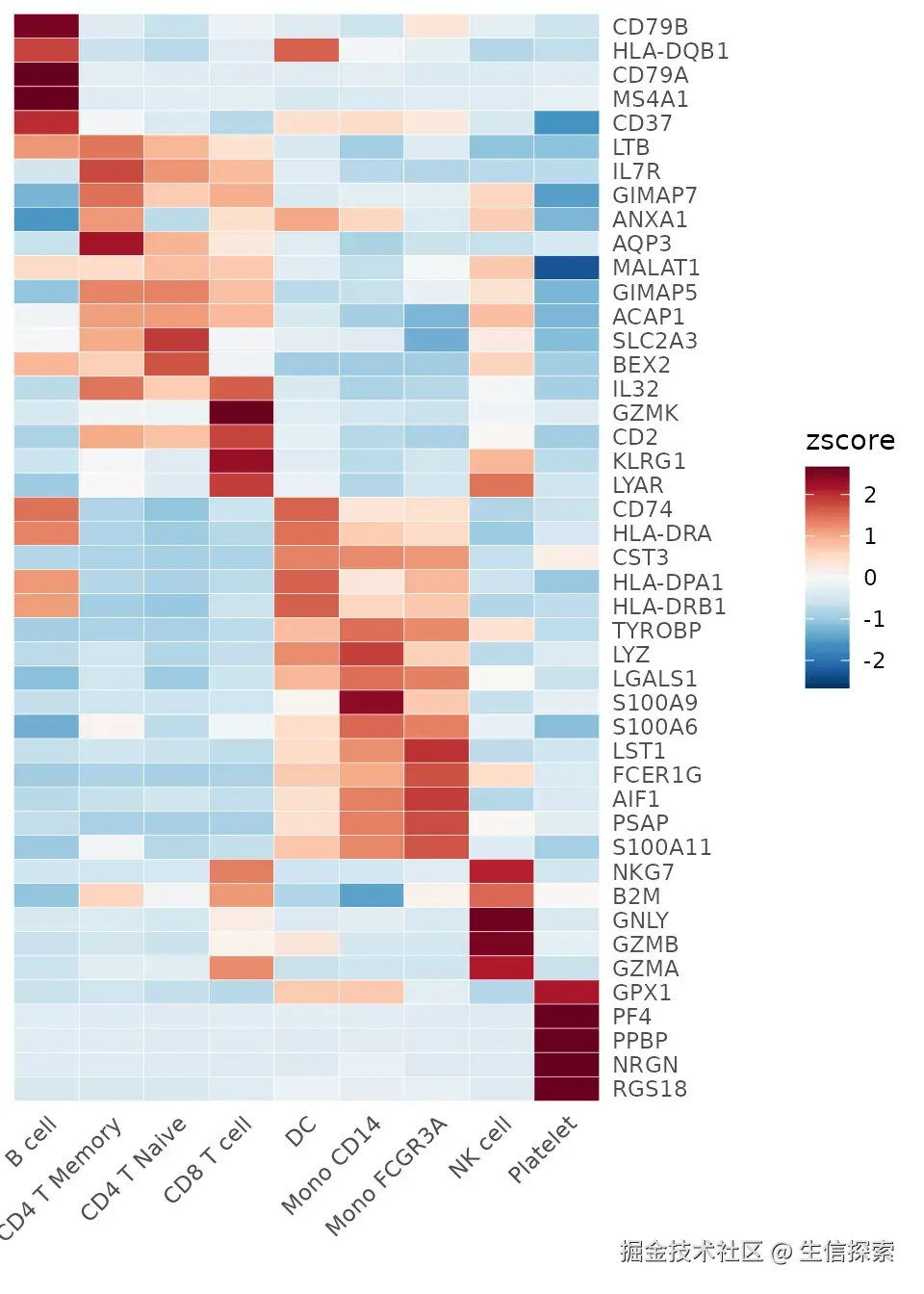
增强版点图(DotPlot2)
点图能同时展示基因表达量(颜色)和表达细胞比例(大小),是单细胞分析的必备图表:
r
DotPlot2(pbmc,
features = grouped_features,
color_scheme = "BuRd",
border = FALSE, # Remove dot borders
show_grid = FALSE, # Remove grid lines
flip = TRUE) # Flip coordinates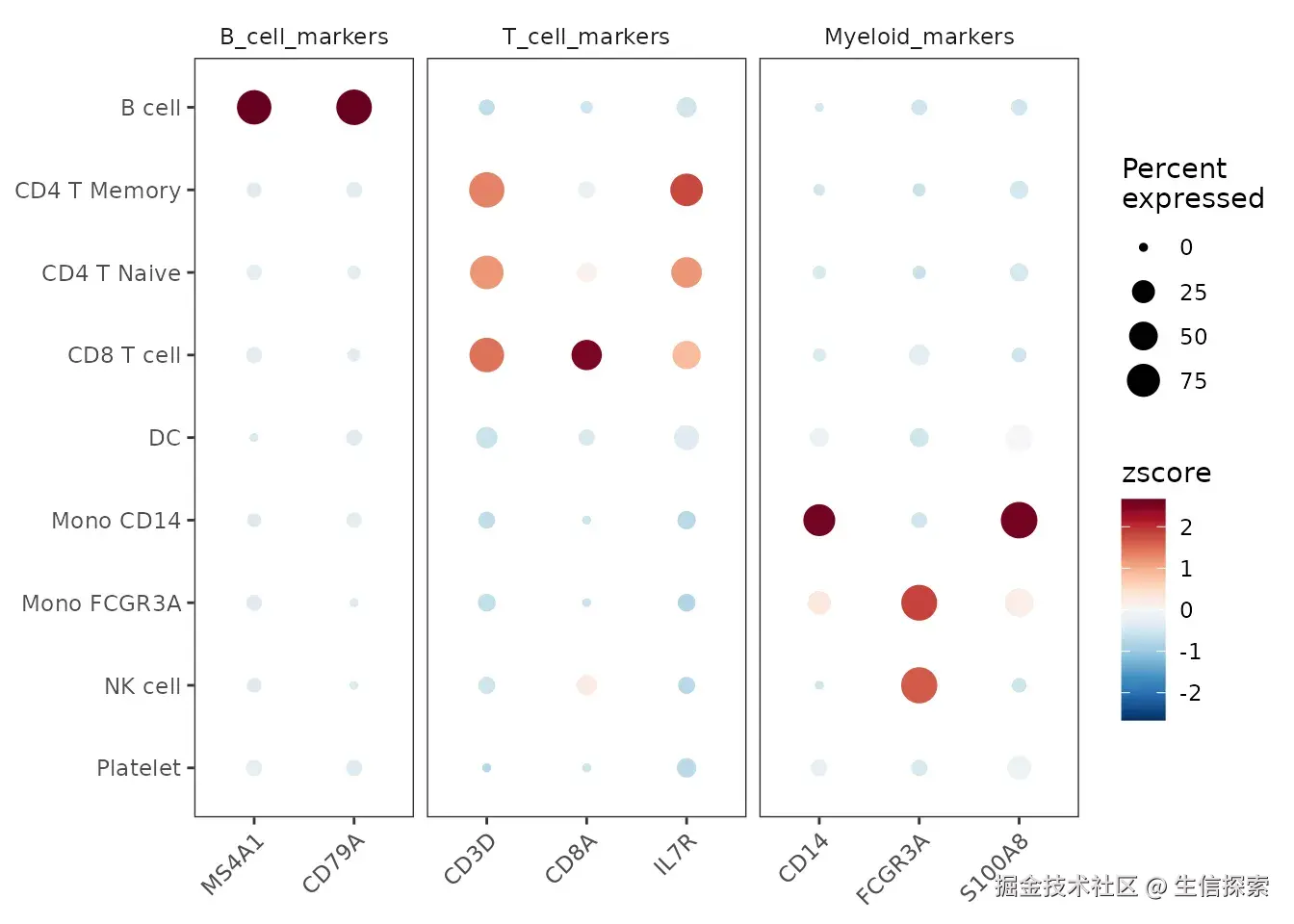
增强版小提琴图(VlnPlot2)
小提琴图能展示基因表达的分布特征,VlnPlot2在此基础上增加了统计分析功能:
r
# 基础用法
VlnPlot2(pbmc, features = c("CD3D","CD14","CD79A"))
# 高级功能:添加统计学标注
VlnPlot2(pbmc, features = "CD3D",
group.by = "cluster",
stat.method = "wilcox.test", # 威尔科克森检验
hide.ns = TRUE) # 隐藏不显著结果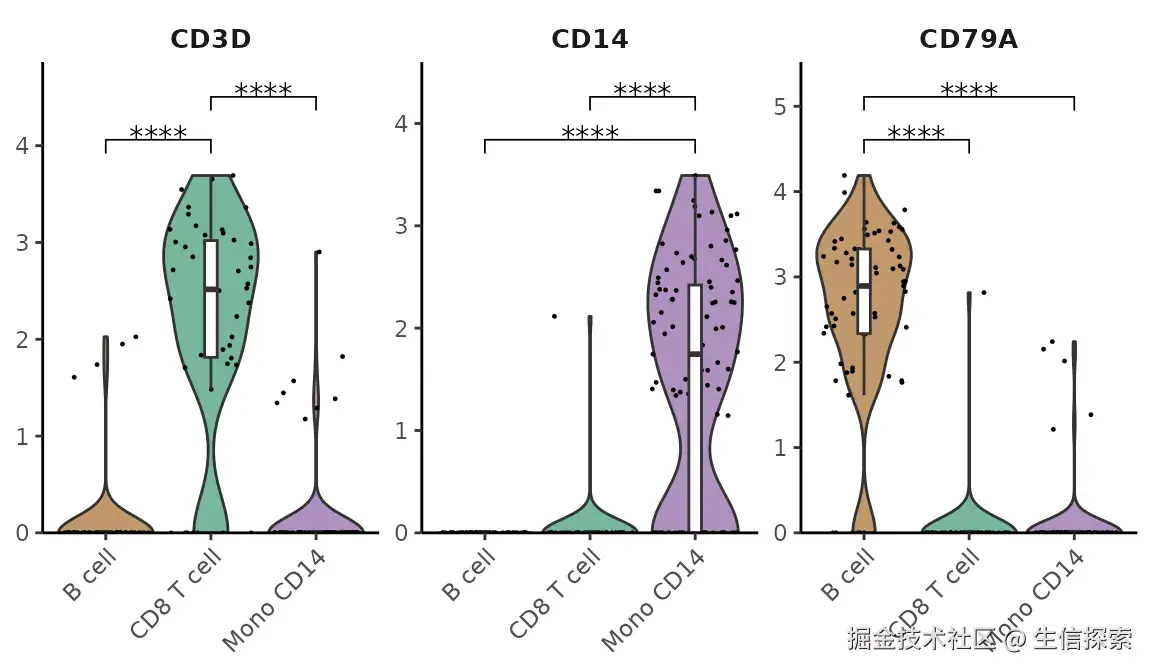
集群分布柱状图(ClusterDistrBar)
想知道每个样本中各细胞集群的比例?这个函数能帮你快速可视化:
r
# 展示各样本中集群的分布比例
ClusterDistrBar(origin = pbmc$orig.ident, cluster = pbmc$cluster)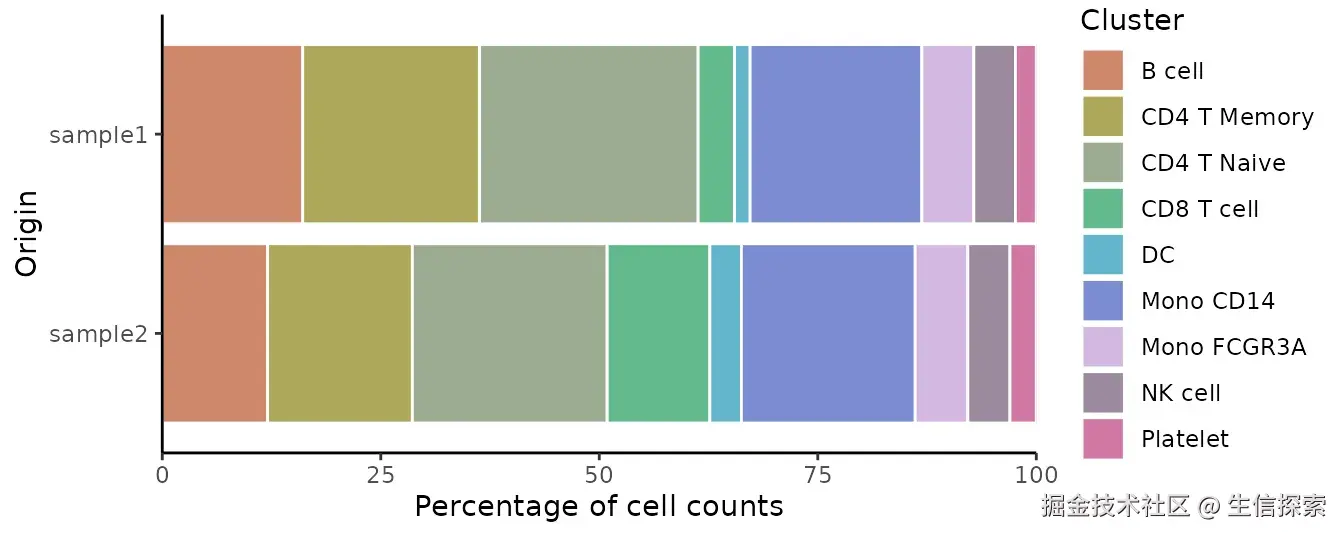
瀑布图(WaterfallPlot)
瀑布图非常适合展示两个细胞群之间的差异基因或通路:
r
# 比较两个细胞群的通路活性差异
WaterfallPlot(matr, f = pbmc$cluster, ident.1 = "Mono CD14", ident.2 = "CD8 T cell")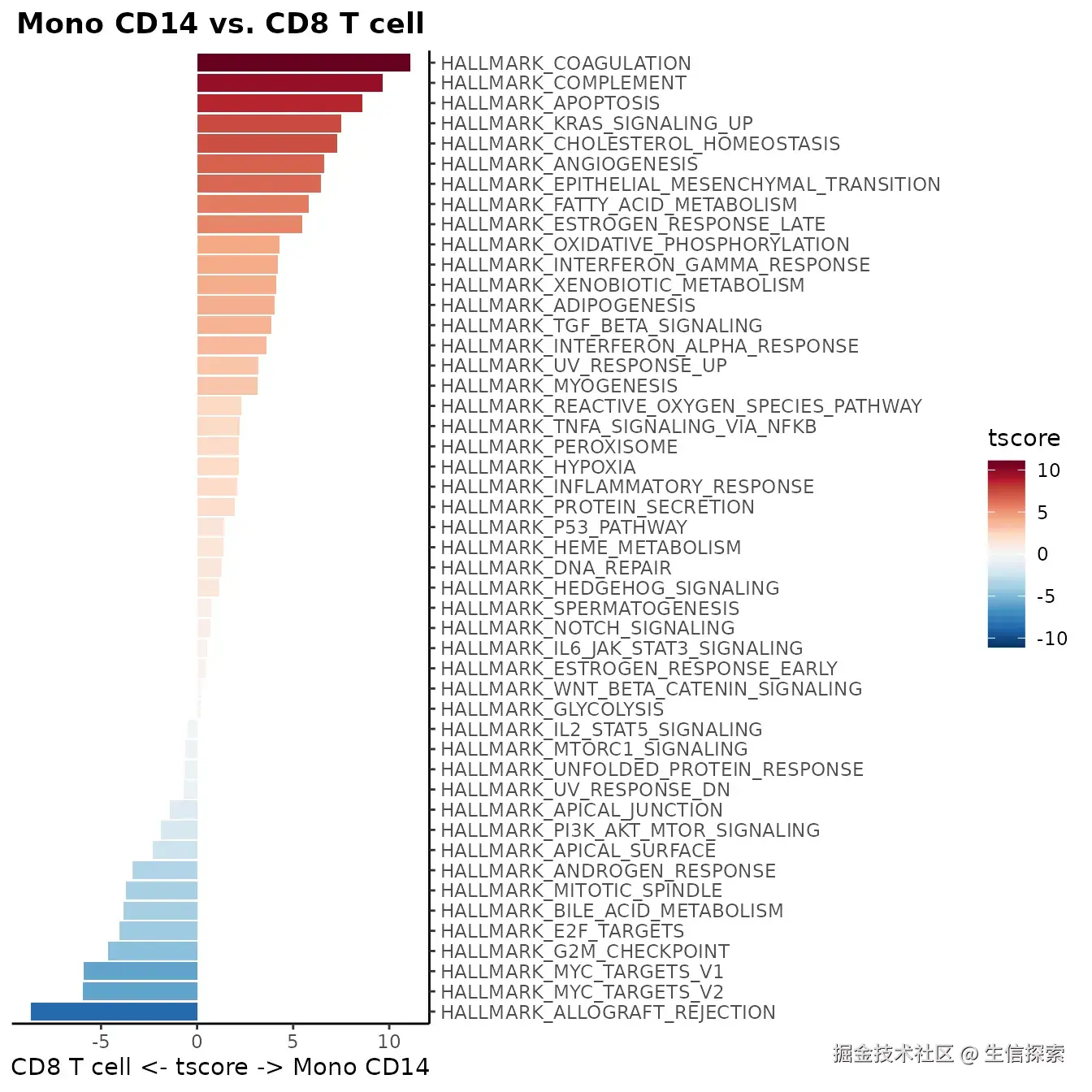
火山图(VolcanoPlot)
火山图能同时展示差异的大小(x轴)和显著性(y轴),是筛选关键基因的好帮手:
r
VolcanoPlot(pbmc,
ident.1 = "B细胞", ident.2 = "CD8 T细胞",
x.threshold = 0.5, # logFC阈值
y.threshold = 2) # -log10(p值)阈值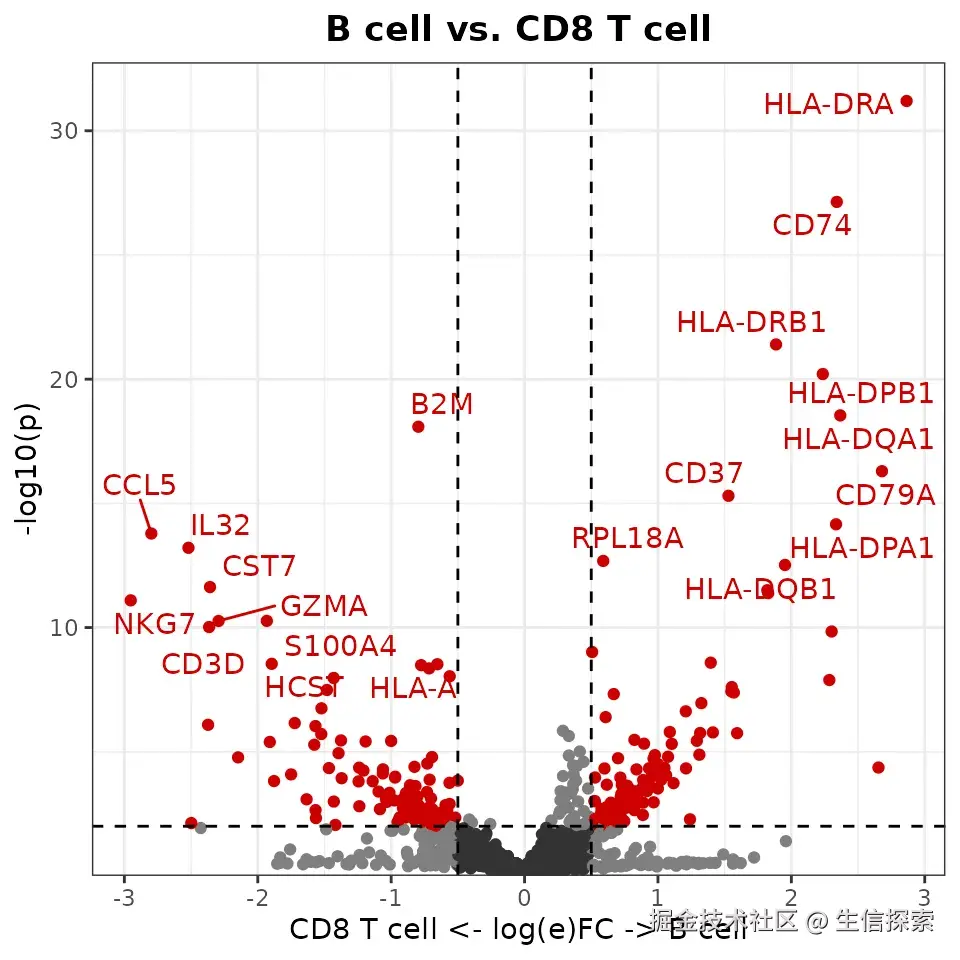
配色方案指南
SeuratExtend提供了丰富的配色方案,让你的图表既专业又美观:
- 离散变量(如集群、样本):推荐使用"default"(深色)或"light"(浅色)方案
- 连续变量(如基因表达):推荐使用"Blues"(蓝色渐变)或"BuRd"(蓝红渐变)
r
# 为离散变量自定义颜色
DimPlot2(pbmc, cols = "pro_blue") # 专业蓝色系
# 为连续变量自定义颜色
FeaturePlot(pbmc, feature = "CD14", cols = "OrRd") # 橙红渐变选择合适的可视化方式
不同图表有不同的适用场景,选择合适的图表类型能让你的结果更有说服力
| 图表类型 | 最佳用途 | 优势 |
|---|---|---|
| 热图 | 比较多个特征在多组间的模式 | 紧凑展示大规模数据 |
| 点图 | 同时展示表达量和表达比例 | 信息更丰富 |
| 小提琴图 | 展示表达分布细节 | 可添加统计分析 |
| 瀑布图 | 展示两组间的差异排序 | 清晰呈现最显著差异 |
| 火山图 | 展示差异大小与显著性 | 快速筛选关键特征 |
Reference
r
https://huayc09.github.io/SeuratExtend/articles/Visualization.html