推理模型的训练通常需依托形式化指定系统(如代码或符号数学)中的验证机制。然而,在生物学等开放领域,我们通常无法获取可支持大规模形式化验证的精确规则,因此往往需要通过实验室中的假设测试来评估预测结果的有效性。通过开展真实实验进行验证不仅过程缓慢、成本高昂,而且本质上无法与计算规模同步扩展。
CZI的研究员提出:无需额外实验数据,可将生物学世界模型或其他先验知识作为生物学知识的"近似先知",并将其用作"软验证器"来训练推理系统。rbio1以预训练大语言模型(LLM)为基础,通过强化学习进行后训练,并在训练过程中利用已习得的生物学模型获取生物学知识,以实现验证功能。软验证能够成功将生物学世界模型的知识提炼到rbio1中:例如,在PerturbQA基准测试的扰动预测任务中,rbio1的性能超越了当前最先进的模型,达到领先水平;同时,还证实了通过组合多种验证器,可训练出泛化能力更强的rbio系列模型。rbio1是一种新的范式:无需依赖实验数据,可利用生物学模型的预测结果(即通过模拟方式)来训练高性能的推理模型。
rbio1 - training scientific reasoning LLMs with biological world models as soft verifiers
https://github.com/czi-ai/rbio
Rbio 包含多种变体,这些变体基于强化学习期间用作验证器的数据类型或模型。其中,TF(Transcriptformer)是一个single-cell基础模型。
| 模型变体名称 | 任务、用途或描述 | 访问 URL 或 AWS 下载链接 |
|---|---|---|
| Rbio1-EXP | 使用直接实验数据作为"硬验证器"进行后训练,以在相关任务上实现最高准确率。 | s3://czi-rbio/rbio1-EXP/ |
| Rbio1-MLP | 使用特定于任务的 MLP 作为"软验证器"进行后训练,展示从较小世界模型进行知识迁移的能力。 | s3://czi-rbio/rbio1-MLP/ |
| Rbio1-TF | 使用来自 Transcriptformer 基础模型的信号(例如 PMI 分数)作为"软验证器"进行后训练。 | s3://czi-rbio/rbio1-TF/ |
| Rbio1-GO | 使用基因本体 (GO) 知识库作为"软验证器"进行后训练,通过 ROUGE 指标使用已建立的生物学事实指导模型。包含来自 GO 本体所有子集的信息:GO-F(分子功能)、GO-P(生物过程)和 GO-C(细胞成分) | s3://czi-rbio/rbio1-GO/ |
| Rbio1-GO-C | 使用基因本体 (GO) 知识库细胞成分 (GO-C) 作为"软验证器"进行后训练,通过 ROUGE 指标使用已建立的生物学事实指导模型。 | s3://czi-rbio/rbio1-GO-C/ |
| Rbio1-GO-F | 使用基因本体 (GO) 知识库分子功能 (GO-F) 作为"软验证器"进行后训练,通过 Rouge 指标使用已建立的生物学事实指导模型。 | s3://czi-rbio/rbio1-GO-F/ |
| Rbio1-GO+EXP | 使用实验数据作为当前任务的"硬验证器",并使用基因本体(GO-all:GO-P + GO-C + GO-F)知识库作为生物事实一致性的"软验证器"进行后训练。 | s3://czi-rbio/rbio1-GO+EXP/ |
| Rbio1-TF+EXP | 使用实验数据作为当前任务的"硬验证器",并使用PMI分数将Transcriptformer基础模型作为"软验证器"进行后训练。 | s3://czi-rbio/rbio1-TF+EXP/ |
| Rbio1-TF+GO+EXP | 使用以下方法进行后训练:实验数据作为当前任务的"硬验证器";Transcriptformer基础模型作为使用PMI分数的"软验证器";以及基因本体(GO-all:GO-P + GO-C + GO-F)知识库作为生物事实一致性的"软验证器"。 | s3://czi-rbio/rbio1-TF+GO+EXP/ |
| Rbio1-TF+GO+MLP | 后训练使用:MLP 充当从较小模型视角观察的世界知识的"软验证器";Transcriptformer 基础模型作为使用 PMI 分数的"软验证器";以及基因本体(GO-all:GO-P + GO-C + GO-F)知识库作为生物事实一致性的"软验证器" | s3://czi-rbio/rbio1-TF+GO+MLP/ |
| Rbio1-TF+GO+MLP+EXP | 后训练使用:实验数据充当当前任务的"硬验证器"; Transcriptformer 基础模型作为使用 PMI 分数的"软验证器";基因本体(GO-all:GO-P + GO-C + GO-F)知识库作为生物事实一致性的"软验证器";MLP 作为通过较小模型呈现的世界知识的"软验证器"。 | s3://czi-rbio/rbio1-TF+GO+MLP+EXP/ |
目录
背景概述
虚拟细胞模型系统的核心前景在于:构建强大的生物学预测模型,无需依赖实验数据,就能对细胞的任意状态转换(如从疾病状态到健康状态,反之亦然)进行预测。目前,大多数基础模型均基于特定的实验模态构建,例如转录组学、成像、蛋白质组学、基因组学等;同时,也有部分研究开始探索基于多模态数据训练的模型(ChatNT)。
要实现真正的虚拟细胞模型范式(即通过一个共享的表征空间整合所有这些生物学世界模型的知识),CZI认为核心挑战在于如何整合那些基于不同数据集、且可能属于独立模态训练出的独立模型(模型整合)。为缓解这些问题并构建真正通用的虚拟细胞系统,CZI将研究目标设定如下:开发一种方法,以语言作为连接模态,将多个生物学世界模型整合到一个公共空间中。这种方法的优势在于,它还能将复杂的生物学模型转化为对话式模型 ------ 这类模型可通过自然语言与用户交互,从而让实验研究者和计算领域使用者都能轻松与生物学世界模型进行互动,并获取其中的知识。通过将生物学模型的知识提炼到大型语言模型(LLM)中,我们能够提取其结构中所编码的、源自实验数据的知识,并将其转化为交互式、人类可理解对话的自然语言模型。这一过程也可视为推理型大型语言模型(reasoning LLM)与生物学世界模型的对齐(alignment)。
传统生物学模型往往需要专业知识才能解读,与之不同的是,对话式模型为查询和理解系统行为提供了便捷的交互界面。大型语言模型(LLMs)通常还具有更强的泛化能力 ------ 相较于生物学模型,它们能适应更广泛的问题和场景,尤其是当这些大型语言模型在包含不同领域知识的大规模语言语料库上进行过预训练时。此外,对话式模型的灵活性使其成为假设生成、结果解释和科学发现的有力工具,进而拓展了我们与生物学知识交互、并从中提取洞见的方式。
为实现研究目标,借助推理型大型语言模型(reasoning LLMs)------ 这类语言模型极具潜力,已在医学、化学等领域展现出良好应用前景,且能够通过对话模拟复杂的生物学相互作用。受近期这一研究进展的启发,CZI的研究员提出采用强化学习(reinforcement learning, RL)技术(具体为 GRPO 算法),促使模型行为与生物学世界模型所习得的生物学洞见保持一致;这一设计可提升科学推理的准确性,并实现更有效的知识交互。此外,CZI采用了一种新的学习范式 ------ 由特定领域的生物学模型制定奖励机制 ------ 来引导模型的学习过程。该范式无需单纯依赖大量带标签数据,而是利用模型预测结果(作为训练信号)。在通用强化学习文献及黑箱优化领域中,"利用替代模型(surrogate model)的预测结果而非带标签数据" 这一理念已有所应用。
将生物学世界模型中的信息提炼到大型语言模型(LLMs)中,这一方法背后的核心思路是:基础模型确实能够充分捕捉生物数据背后的流形结构,且通过这种捕捉能力,我们可以对该流形进行 "遍历",从而获取更多数据点及其具有实际意义的表征。基于此,我们能够开展合理的比较与预测,而这些比较和预测结果最终可作为 "软" 评分信号,用于评估大型语言模型生成的文本推理轨迹。此外,这种方法与"针对生物学特定文本优化大型语言模型的下一个 token 预测能力" 存在本质区别。图 1 中展示了所提方法的示意图。在模型能力测试方面,考虑到基因扰动预测在理解细胞状态转换中的核心地位,将其作为下游任务 ------ 具体而言,该任务是预测敲除某个基因后,细胞内其他基因所产生的结果。
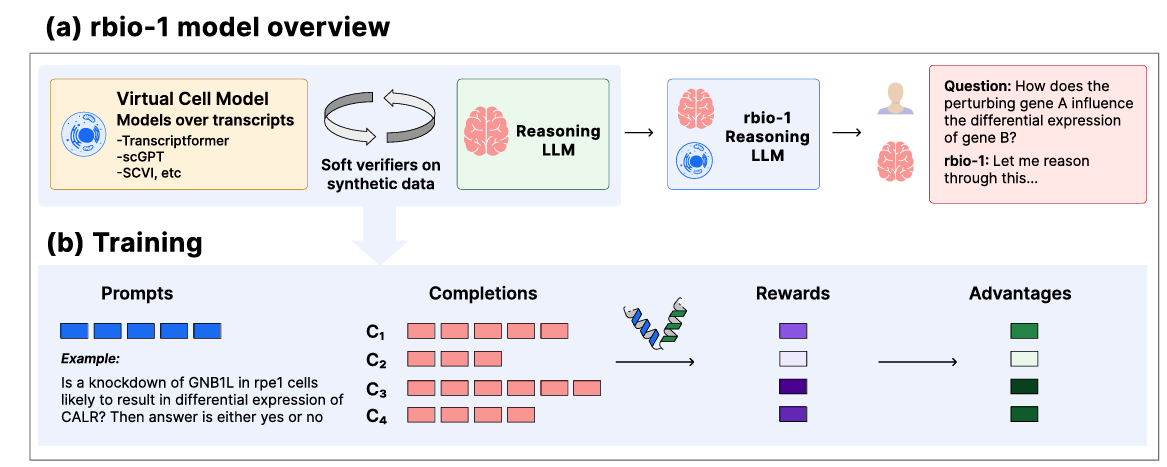
- 图a)rbio1 整体介绍:rbio1 通过软验证将虚拟细胞模型中的生物学信息提炼到推理型大型语言模型中 ------ 该过程以模拟结果而非实验数据作为训练信号。最终得到的是一个具备生物学知识的推理模型,用户可通过自然语言与之对话。
- 图b)基于 GRPO 的训练流程示意图:对于每个prompt,大型语言模型生成 N 个补全结果,每个结果根据虚拟细胞模型(VCM)的响应获得相应奖励。奖励被转化为优势值,用于指导训练信号的生成。
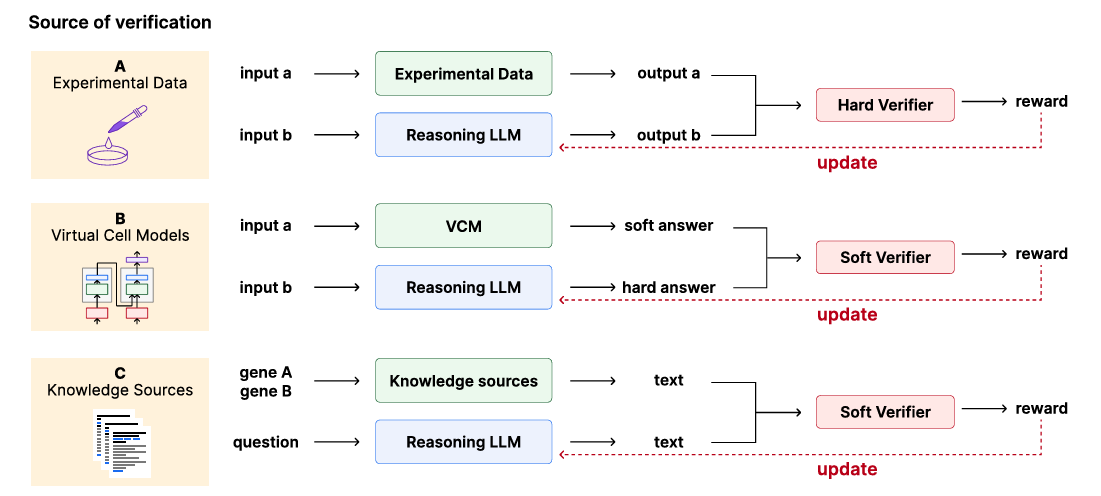
- 图c:通用软监督示例(基于知识源):借助基因本体(GO)知识库,将基因扰动信息转化为自然语言表述的事实。软验证器(基于关键词、ROUGE 分数、似然估计)接收推理型大型语言模型回答中的推理部分,结合基因本体提供的描述,输出奖励。
本质上,rbio1关注的是差异表达(differential expression)------ 即受基因敲低(gene knockdown)影响的基因,其表达水平发生的变化(上调或下调)。rbio1将这一研究问题转化为以下自然语言查询:
- Is a knockdown of gene A in this particular cell line likely to result in differential expression of gene B?(在该特定细胞系中敲低基因 A,是否可能导致基因 B 的差异表达?)
在转录组学领域,这类特定任务相关的问题通常通过以下两种方法解决:(1)借助基于特定任务数据训练的定制模型;(2)利用在大规模语料库上预训练的基础模型 ------ 这类模型通过微调或零样本学习能力,适配下游任务。
对于扰动预测任务,近期研究表明(Deep-learning-based gene perturbation effect prediction does not yet outperform simple linear baselines),定制模型的性能往往优于第二类模型(即基础模型)。构建定制模型通常需要大量专用训练数据,而这些数据需通过实验室实验获取,这使得模型训练受限于数据的可获得性与资源条件。随着转录组学基础模型的兴起,利用模型在大规模预训练过程中习得的生物学知识,并将其应用于扰动预测等特定任务问题以辅助预测,具有巨大潜力。然而,对于仅基于生物数据(而非语言数据)训练的生物学模型,要使其应对分布外任务(即模型未见过的数据或任务场景),直接通过提示词引导的方式并非易事。
而rbio1的实验表明,在 PerturbQA 数据集(该数据集包含四种癌细胞系 ------RPE1、K562、HEPG2、JURKAT------ 的单基因扰动敲低数据)上,以软验证器作为奖励信号训练的推理模型,能够在分布外数据集与任务上实现泛化,且无需依赖实验数据进行训练。
示例

- 扰动预测任务的推理轨迹示例。该示例针对从测试集中随机抽取的一个问题展开回答,展示了不同思维链(chain-of-thought)技术对应的多种回答示例。所有输出均来自一个基于多软验证器组合训练的模型,即 rbio-TF+GO+MLP 模型。

- 基于基因共表达数据训练的 rbio-TF 模型针对非扰动相关问题的回答示例。
rbio1的用法
在https://github.com/czi-ai/rbio/blob/main/inference.py中:
python
def run_rbio_inference(
aws_s3_bucket: str,
base_model_name: str,
rbio_model_ckpt: str,
results_output_folder: str,
results_output_filename: str,
):
# These can be changed
CoT_suffix = " The Biologist provides the reasoning step-by-step."
system_prompt_orig = "A conversation between User and Biologist. The user asks a question, \
and the Biologist solves it. The biologist first thinks about the reasoning process in the mind and \
then provides the user with the answer. The reasoning process and answer are enclosed within \
<think> </think> and <answer> </answer> tags, respectively, i.e., <think> reasoning process here </think> <answer> answer here </answer>."
system_prompt_orig_CoT = system_prompt_orig + CoT_suffix
questions = [
"Is a knockdown of CPAMD8 in rpe1 cells likely \
to result in differential expression of SPARC? The answer is either yes or no. "
]
inference_fn(
aws_s3_bucket=aws_s3_bucket,
base_model_name=base_model_name,
rbio_ckpt=rbio_model_ckpt,
system_prompt=system_prompt_orig_CoT,
system_prompt_type="system_prompt_orig_CoT",
questions=questions,
results_output_folder=results_output_folder,
results_output_filename=results_output_filename,
)问题属于是或否的问答形式:"Is a knockdown of CPAMD8 in rpe1 cells likely to result in differential expression of SPARC? The answer is either yes or no. "(在 rpe1 细胞中敲低 CPAMD8 是否可能导致 SPARC 的差异表达?答案是肯定的,也可能是否定的。)