基于深度学习的胸部 CT 肺癌诊断项目 --- 设计与实现(含Web端)
一套从模型推理到网页交互的完整应用:上传胸部 CT 图像,返回四类诊断结果(肺腺癌/大细胞未分化癌/正常/鳞状细胞癌),并支持历史记录、批量预测与可视化扩展。
项目亮点
- 端到端:模型加载、预测推理、Flask Web 界面、历史记录一体化。
- 高可用回退:权重加载失败时,自动切换至 ImageNet 预训练 EfficientNetB3 作为备选,保证服务可运行。
- 易扩展:已预留仪表盘/可视化挂点,便于后续接入 ECharts 等前端图表。
- 跨平台路径处理 :上传文件以相对
static路径生成 URL,避免 Windows 路径导致的 404。
目录结构
text
code/
app.py # Flask 应用后端(模型加载/预测/API/页面路由)
requirements.txt # 依赖列表
efficientnetb3_notop.h5 #(可选)完整模型文件(若为整模型)
efficientnetb3_base_best_weights.h5 #(可选)训练权重
efficientnetb3_base_best_weights2.h5 #(可选)备用训练权重
Data/ # 数据集(train/valid/test)
static/ # 前端静态资源(CSS/JS/图片等)
img/predict_test/ # 上传预测图片存放目录
templates/ # Jinja2 页面模板(index、dashboard、history 等)
test_*.py # 若干测试脚本(模型/接口测试)
README.md # 本文档提示:
static/img/predict_test/用于存放上传的预测图片,后端会生成/static/...形式的可访问 URL。
技术栈
- 后端框架:Flask
- 深度学习:TensorFlow 2.x、Keras(EfficientNetB3)
- 图像处理:Pillow、OpenCV(可选)
- 前端:Bootstrap、jQuery、ECharts(已包含依赖,可直接扩展)
- 数据库存储:SQLite(用户与预测历史)
环境准备
1) Conda 环境(推荐)
bash
conda create -n lungct python=3.8 -y
conda activate lungct2) 安装依赖
bash
pip install -r requirements.txt如未包含全部依赖,可补充安装:
bash
pip install tensorflow pillow numpy opencv-python flask启动与使用
演示图片
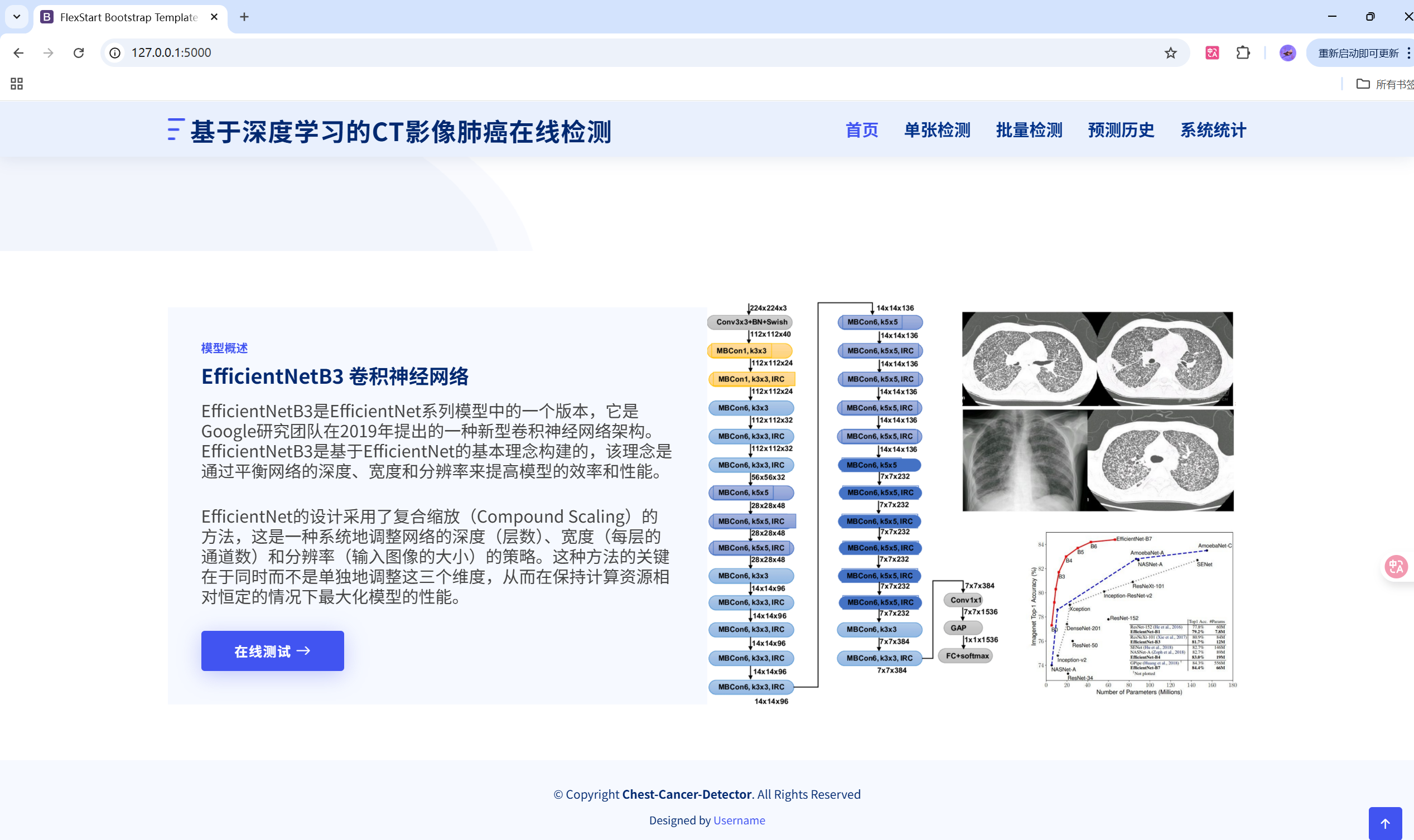
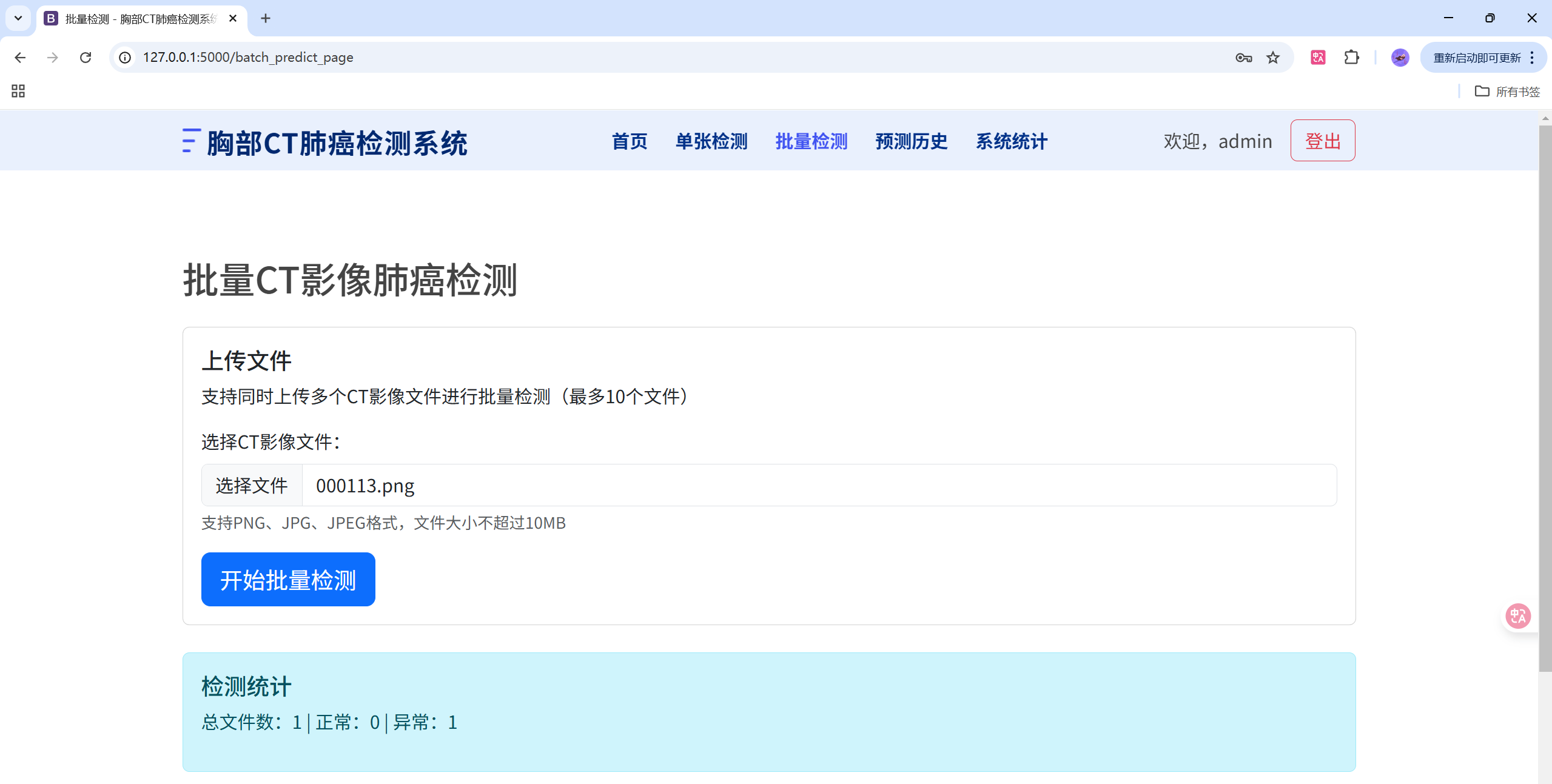
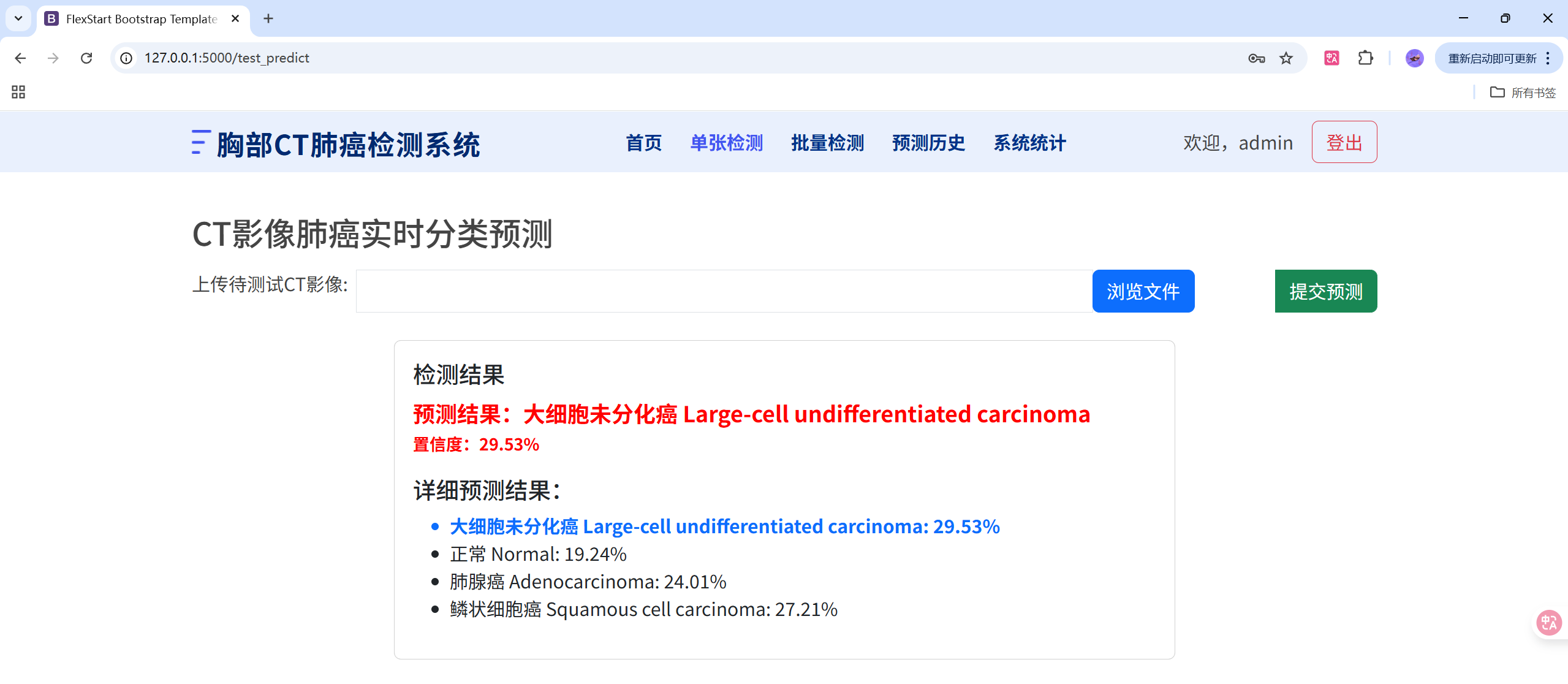
启动服务
bash
python app.py
# 输出类似:Running on http://127.0.0.1:5000访问页面
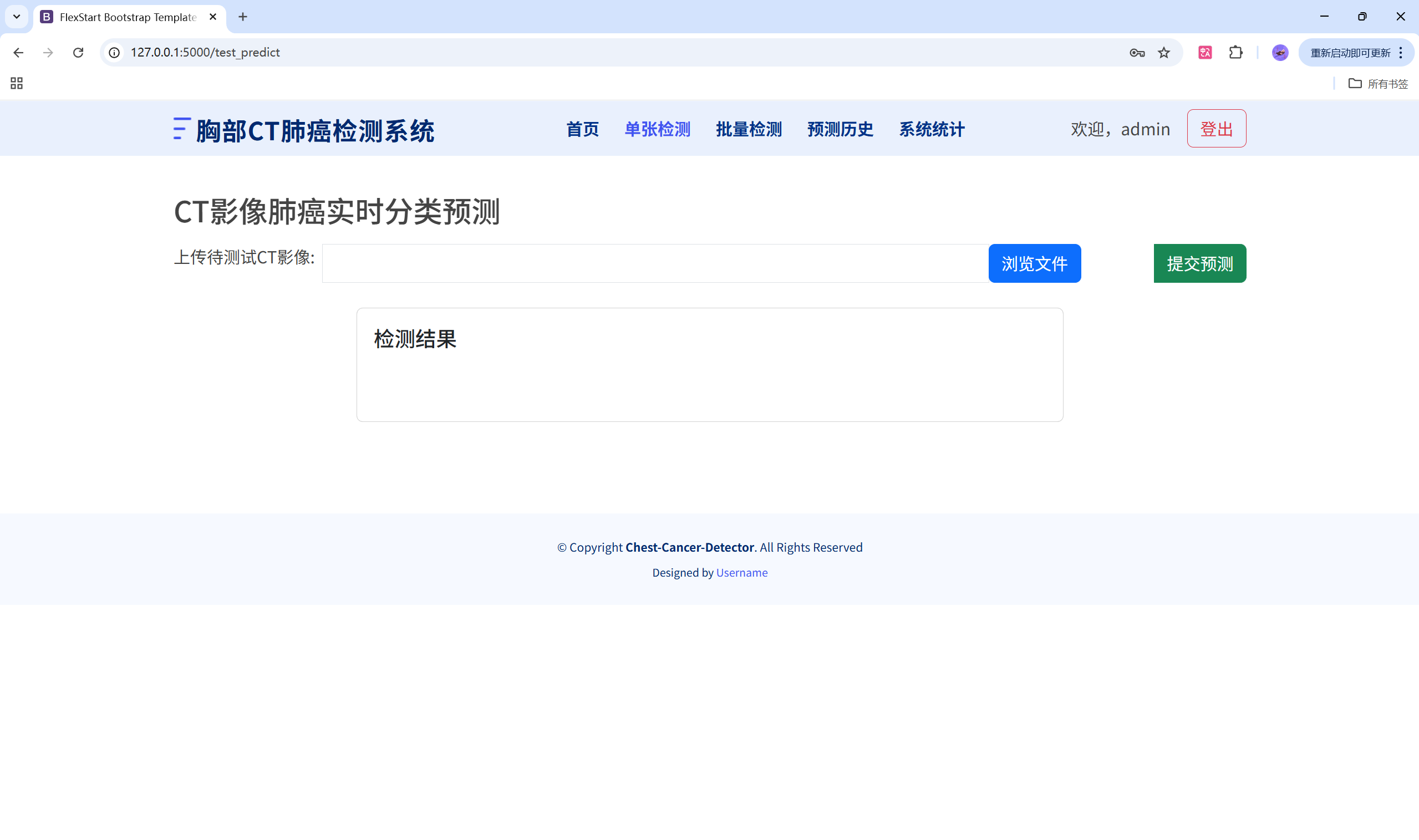
- 首页与单图预测:
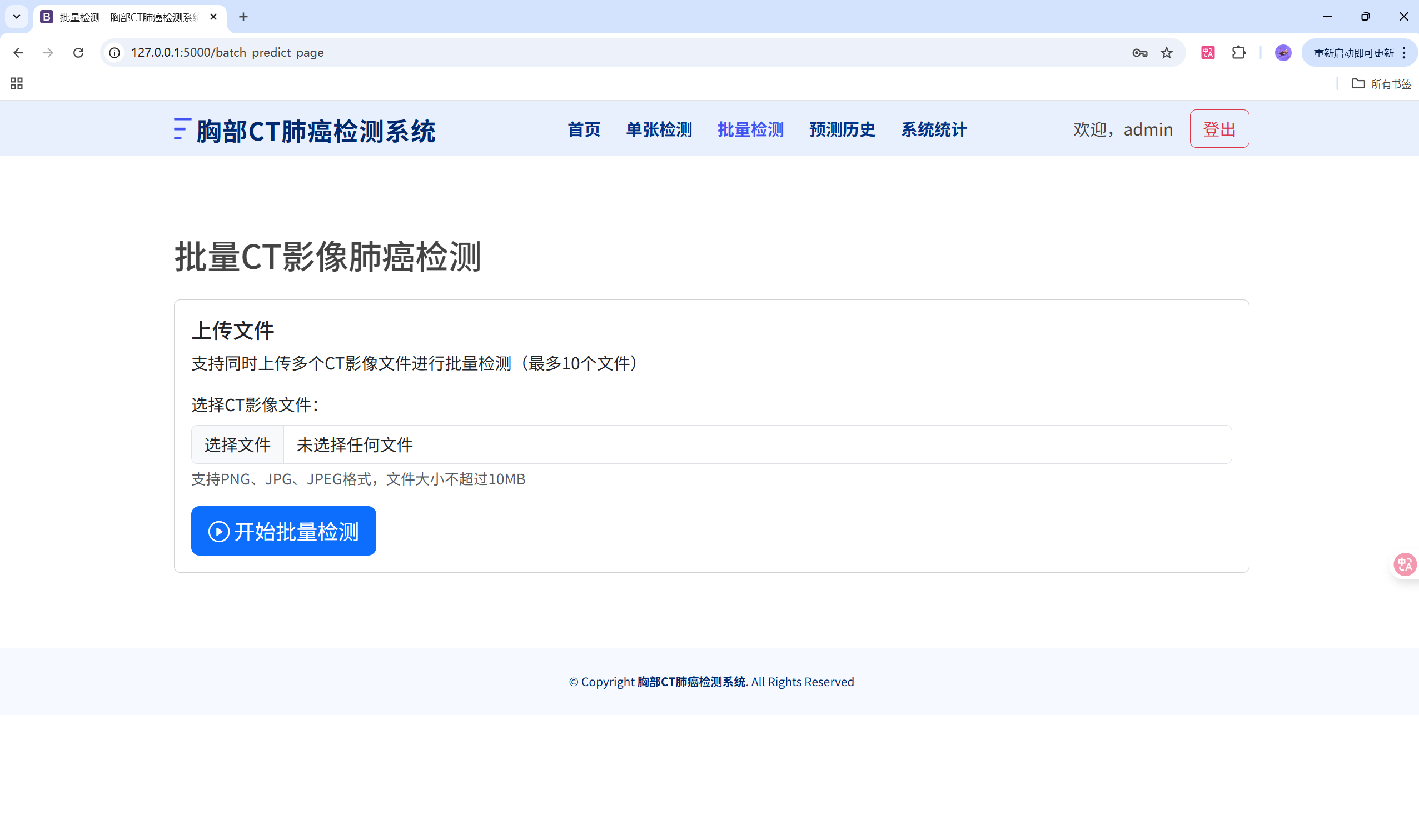
http://127.0.0.1:5000/ - 批量预测:
http://127.0.0.1:5000/batch_predict_page - 历史记录:
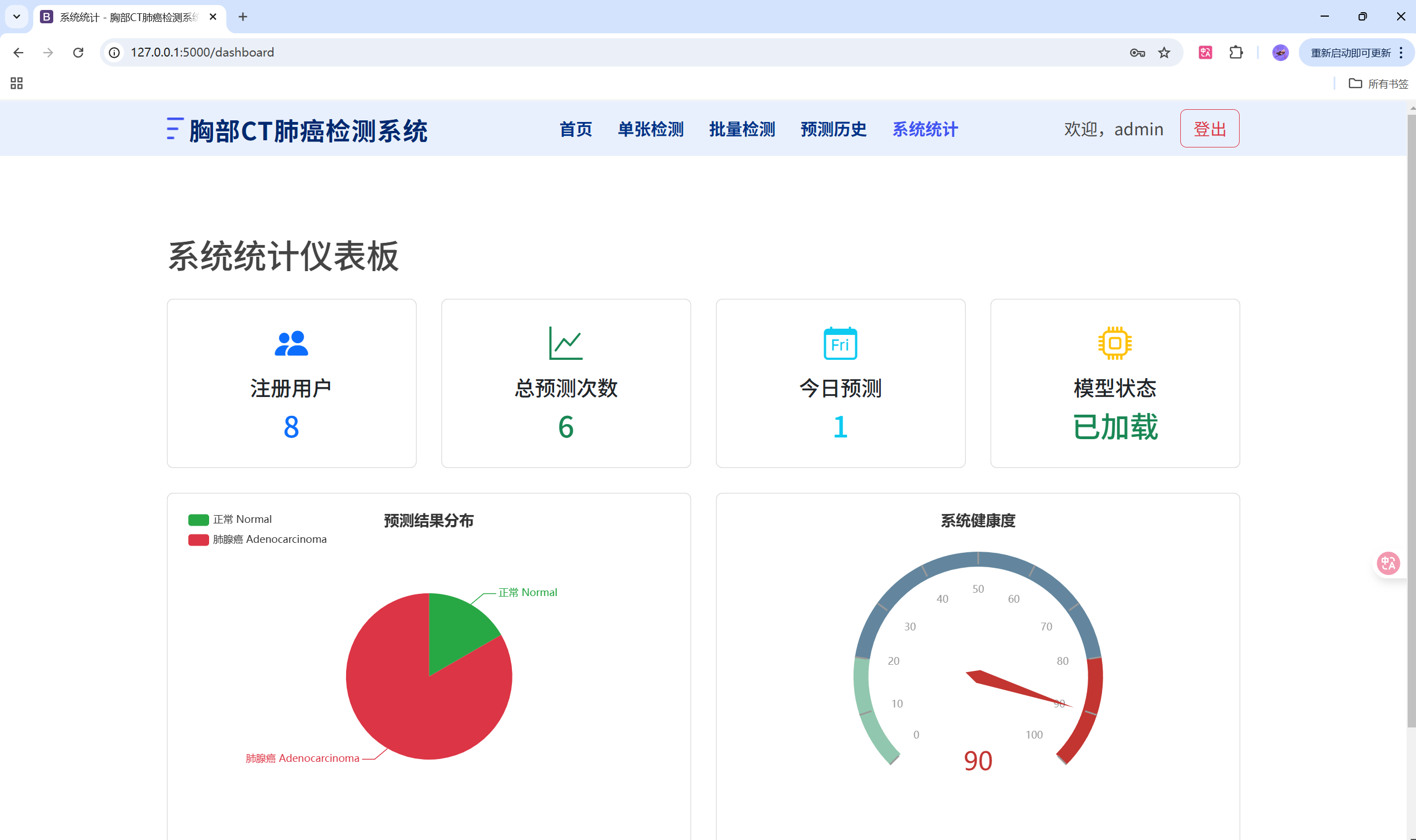
http://127.0.0.1:5000/history - 仪表盘(可视化预留):
http://127.0.0.1:5000/dashboard
模型与加载策略
项目默认使用 EfficientNetB3 作为特征提取骨干,输入大小为 (224\times224\times3)。
模型头(分类层)
python
# 伪代码展示(推理端结构需与训练端保持一致)
from tensorflow.keras.applications import EfficientNetB3
from tensorflow.keras.layers import Dense, GlobalAveragePooling2D
from tensorflow.keras.models import Model as KerasModel
height, width = 224, 224
base_model = EfficientNetB3(weights=None, include_top=False, input_shape=(height, width, 3))
x = base_model.output
x = GlobalAveragePooling2D()(x)
x = Dense(1024, activation='relu')(x)
x = Dense(512, activation='relu')(x)
predictions = Dense(4, activation='softmax')(x)
model = KerasModel(inputs=base_model.input, outputs=predictions)加载策略(多级回退)
- 尝试加载整模型
efficientnetb3_notop.h5(若它确为model.save(...)导出的整模型)。 - 若失败,构建相同结构后加载权重
efficientnetb3_base_best_weights.h5。 - 若仍失败,尝试备用权重
efficientnetb3_base_best_weights2.h5。 - 最终回退:加载
weights='imagenet'的 EfficientNetB3 冻结骨干,添加同样分类头并编译,以保证服务可用。
可选增强:当层名一致但层数不完全匹配时,尝试按名加载并跳过不匹配层:
python
model = build_same_structure_model()
model.load_weights('efficientnetb3_base_best_weights.h5', by_name=True, skip_mismatch=True)核心后端逻辑(节选)
路由概览
/:首页,上传单图预测/test_predict:测试页面/history:历史记录/batch_predict_page:批量预测页面/dashboard:仪表盘(可插入可视化图表)
单图预测接口
python
@app.route('/submit_and_predict', methods=['POST'])
def submit_and_predict():
# 1) 保存上传图片到 static/img/predict_test/
# 2) PIL 读取与预处理(RGB、resize 到 224x224)
# 3) model.predict 得到 4 类概率,取最大值和标签
# 4) 结果写入 SQLite(若用户已登录)
# 5) 以 url_for('static', filename=...) 返回可访问图片 URL
return jsonify(result)返回示例:
json
{
"upload_image": "/static/img/predict_test/000109 (4).png",
"predict": "鳞状细胞癌 Squamous cell carcinoma",
"confidence": 26.11,
"all_predictions": {
"肺腺癌 Adenocarcinoma": 23.92,
"大细胞未分化癌 Large-cell undifferentiated carcinoma": 24.32,
"正常 Normal": 25.64,
"鳞状细胞癌 Squamous cell carcinoma": 26.11
}
}批量预测接口
python
@app.route('/batch_predict', methods=['POST'])
def batch_predict():
# 多文件循环保存、读取、预测
# 为每张图片生成 image_path 的静态 URL,前端即可直接展示
return jsonify({'results': results, 'status': 'ok'})代码详解与关键片段解释
1) 模型加载的多级回退机制
目的:保证在不同部署环境中仍能运行,即便权重/整模型不可用。
python
def load_model_safely():
# 方案1:优先尝试加载整模型(若 h5 确实是整模型)
base_model = load_model('efficientnetb3_notop.h5', compile=False)
# 方案2:构建相同结构后加载权重(与训练端保持一致)
model = create_model()
model.load_weights('efficientnetb3_base_best_weights.h5')
# 方案3:备用权重
model.load_weights('efficientnetb3_base_best_weights2.h5')
# 方案4:最终回退,使用 ImageNet 预训练骨干,冻结后添加分类头
return create_working_model()关键点:
- 整模型与仅权重的区别:整模型包含结构+权重;仅权重需要先构建完全相同的结构。
- 若层数量不匹配但层名基本一致,可尝试
by_name=True, skip_mismatch=True按名加载。
2) 上传图片路径与可访问 URL 的生成
目的:避免 Windows 绝对路径导致浏览器 404,统一返回可访问的 /static/... URL。
python
test_file_path = os.path.join(current_dir, 'static', 'img', 'predict_test', filename)
# 关键转换:把磁盘路径转为相对 static 的路径,再用 url_for 生成 URL
static_dir = os.path.join(current_dir, 'static')
rel_path = os.path.relpath(test_file_path, static_dir).replace('\\', '/')
image_url = url_for('static', filename=rel_path)要点:
relpath将绝对路径转相对static,replace('\\','/')统一分隔符。- 返回
image_url给前端直接<img src="...">使用。
3) 预测输出与标签映射
python
predictions = model.predict(test_X) # shape: (1, 4)
pred_labels = np.argmax(predictions, 1)[0] # 取最大概率的类别索引
confidence = float(np.max(predictions[0])) * 100
predict_class = class_name_dict[pred_labels]
all_predictions = {
class_name_dict[i]: round(float(predictions[0][i]) * 100, 2)
for i in range(len(class_name_dict))
}要点:
- 置信度输出为百分比;
all_predictions便于前端做可视化或表格展示。
4) 历史记录写入(SQLite)
目的:记录用户最近的预测行为,便于回溯与数据分析。
python
# 若不存在则创建表
CREATE TABLE prediction_history(
id INTEGER PRIMARY KEY AUTOINCREMENT,
username CHAR(256),
filename CHAR(256),
prediction CHAR(256),
confidence REAL,
timestamp DATETIME DEFAULT CURRENT_TIMESTAMP
);
# 插入记录(当用户已登录)
INSERT INTO prediction_history (username, filename, prediction, confidence)
VALUES (?, ?, ?, ?)要点:
timestamp默认当前时间,前端可按时间倒序展示最近 20 条。- 可扩展字段:如模型版本、推理耗时、图像来源等。
5) 批量预测流程
python
for file in files:
# 保存 → 读取(PIL) → 预处理(resize 到 224x224) → model.predict
# 生成 image_url → 追加到 results
return jsonify({'results': results, 'status': 'ok'})要点:
- 对每张图片独立生成
image_url,前端可预览每张预测结果。 - 若单张失败,记录
status='error'与错误信息,不影响其他图片处理。
前端可视化(预留)
本项目已包含 ECharts 等前端依赖,可在 templates/dashboard.html 中挂载图表:
html
<!-- 在 dashboard.html 中添加占位 div -->
<div id="prediction-distribution" style="height:360px;"></div>
<script>
const chart = echarts.init(document.getElementById('prediction-distribution'));
fetch('/get_system_stats').then(r => r.json()).then(stats => {
const dist = stats.prediction_distribution || {};
const data = Object.keys(dist).map(k => ({ name: k, value: dist[k] }));
chart.setOption({
title: { text: '预测结果分布' },
tooltip: { trigger: 'item' },
series: [{ type: 'pie', radius: '55%', data }]
});
});
window.addEventListener('resize', () => chart.resize());
// 可继续添加:今日预测趋势、用户增长、词云等
</script>你也可以在 history.html 中用表格展示最近 20 条预测记录,或结合 ECharts Timeline 展现时间序列。
数据与预处理
- 输入:胸部 CT 切片图像(PNG/JPG)
- 预处理:
- PIL 读取,确保 RGB 模式
- resize 到 (224\times224)
- 堆叠 batch 维度后进入模型
model.predict
如需归一化/标准化,请与训练阶段保持一致,并在推理前加入同样的预处理步骤。
常见问题(FAQ)
1. 权重无法加载,日志提示 Layer count mismatch 或 UTF-8 解码失败?
- 确认
efficientnetb3_notop.h5是否为整模型(model.save('xxx.h5')),若不是请重新导出整模型或仅提供权重并构建相同结构后再load_weights。 - 若结构基本一致但不完全匹配,可尝试
by_name=True, skip_mismatch=True按名加载跳过不匹配层。
2. Windows 上传图片后 404?
- 已修复:返回的图片链接统一为
url_for('static', filename=...)生成的/static/...相对 URL,避免GET /f:/...的绝对路径访问。
3. GPU/CPU 运行问题?
- 代码已兼容 CPU 运行;若有 GPU,默认按需分配显存。必要时可在启动前设置
TF_CPP_MIN_LOG_LEVEL来减少日志噪音。
4. 如何评估模型质量?
- 在
test_*.py中可扩展:单图/批量评测、混淆矩阵、ROC 曲线等;前端仪表盘亦可接入可视化评估结果。
测试与示例
单图预测(cURL 示例)
bash
curl -X POST \
-F "file=@static/img/predict_test/your_ct.png" \
http://127.0.0.1:5000/submit_and_predict批量预测(cURL 示例)
bash
curl -X POST \
-F "files=@static/img/predict_test/a.png" \
-F "files=@static/img/predict_test/b.png" \
http://127.0.0.1:5000/batch_predict部署建议
- 生产环境建议使用
gunicorn/uwsgi+nginx部署,关闭 Flask 调试模式。 - 静态资源建议由
nginx托管,后端仅提供 API。 - 将权重/模型文件挂载到只读目录,并对上传目录设置最小可写权限。
维护规划(可选)
- 加入模型版本管理(e.g.,
v1,v2目录分版),热更新模型。 - 引入统一的配置文件(YAML/JSON),管理端口、模型路径、阈值等。
- 增加日志与监控:接口耗时、模型推理耗时、错误率监控等。
版权与鸣谢
- 模型骨干:EfficientNetB3(ImageNet 预训练)
- 前端框架与资源:Bootstrap、ECharts 等
如使用本项目进行科研或产品化,请遵守相关依赖的开源许可。
联系方式
码界筑梦坊各大平台同名