一.k-means算法
k均值聚类算法是一种迭代求解算法,一般采用常见的欧氏距离作为样本距离度量准则。
每个样本点到本簇中心的距离的平方和也称为误差平方和(SSE)
1.k-means流程
(1).初始化聚类中心:从所有数据点中,随机选择k个不重复的点作为初始的k个聚类中心(k需提前指定)。
(2).分配数据点:计算每个数据点到k个聚类中心的距离(常用欧氏距离),将该点分配到距离最近的聚类中心所在的簇
(3).更新聚类中心:针对每个簇,计算簇内所有数据点的均值(即各维度坐标的平均值),将这个均值作为该簇新的聚类中心。
(4). 判断迭代终止:重复步骤2和步骤3,直到聚类中心的位置变化小于预设的阈值(如移动距离极小),或迭代次数达到设定上限,算法停止。
例子:用k-means算法将下面九个点聚类为两个簇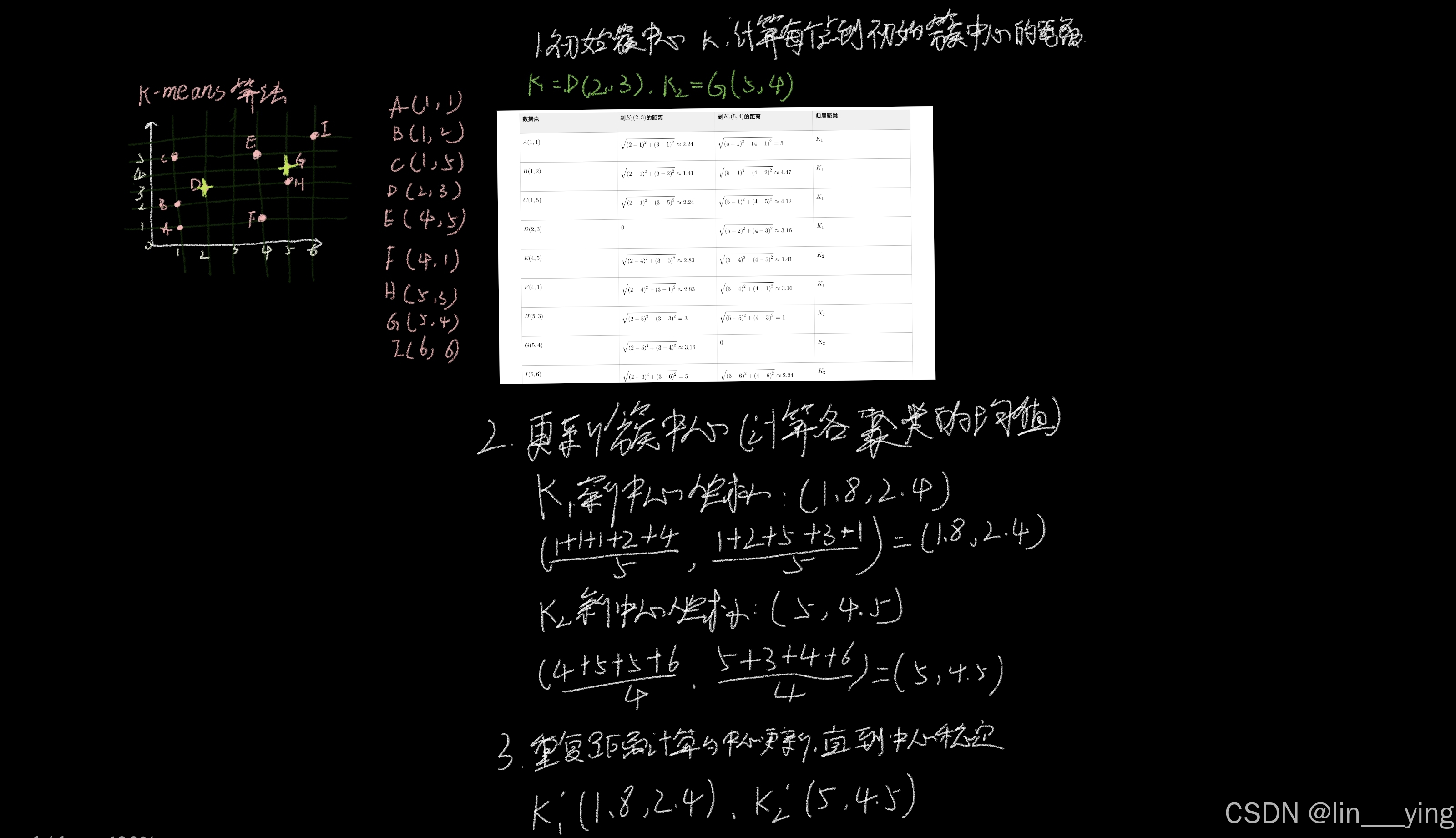
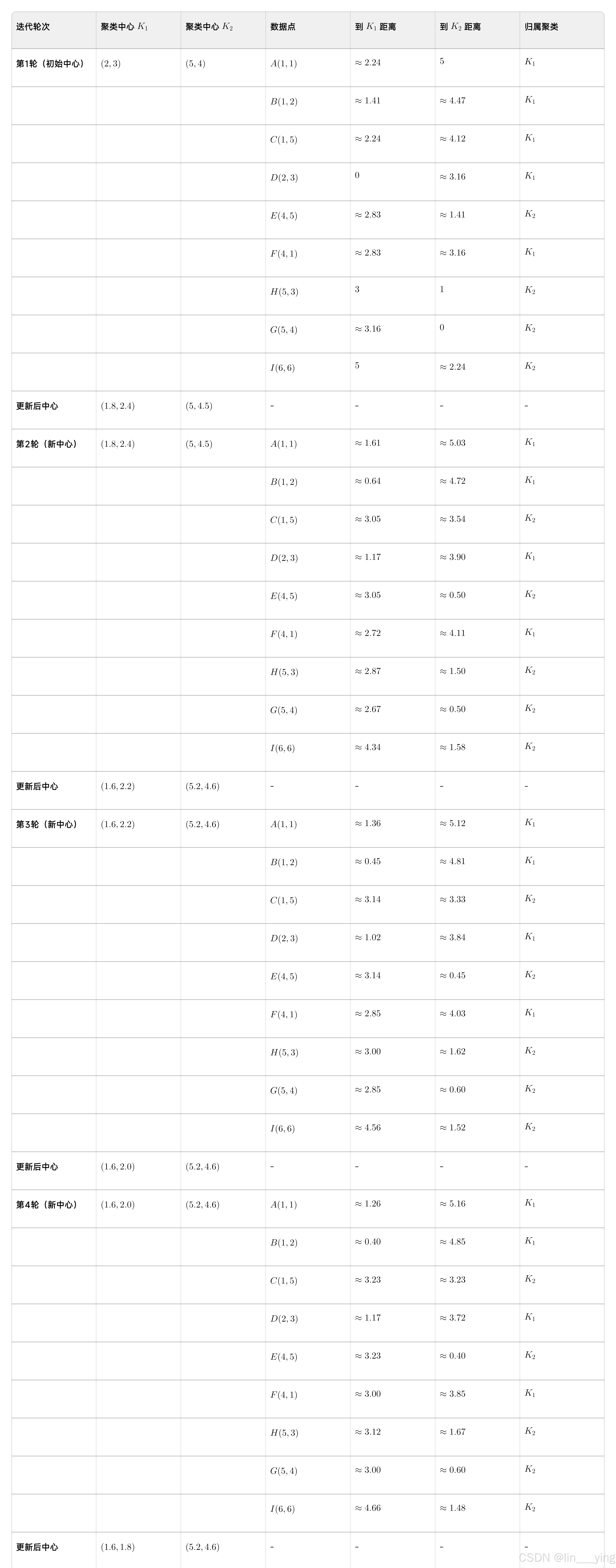
最终稳定的聚类为K1:ABDF K2:CEHGI
2.用python实现k-means算法
python
# -*- coding: utf-8 -*-
"""
Created on Thu
k-means clustering algorithm
@author:zzy
"""
import numpy as np
import matplotlib.pyplot as plt
def L2(vecXi,vecXj):
'''
计算欧氏距离
param vecXi:
param vecXj:
return:
'''
return np.sqrt(np.sum(np.power(vecXi -vecXj,2)))
def kMeans(S,k,distMeans=L2):
'''
k均值聚类
param S:
param K:
param distMeans:
return SSE:误差平方和
'''
m = np.shape(S)[0]
sampleTag = np.zeros(m)
n = np.shape(S)[1]
clusterCents = np.array([[-1.93964824,2.33260803],[7.79822795,6.72621783],[10.64183154,0.20088133]])
sampleTagChanged = True
SSE = 0.0
while sampleTagChanged:
sampleTagChanged = False
SSE = 0.0
for i in range(m):
minD = np.inf
minIndex = -1
for j in range(k):
d = distMeans(clusterCents[j,:],S[i,:])
if d < minD:
minD = d
minIndex = j
if sampleTag[i] != minIndex:
sampleTagChanged = True
sampleTag[i] = minIndex
SSE += minD**2
print(clusterCents)
plt.scatter(clusterCents[:,0].tolist(),clusterCents[:,1].tolist(),c='r',marker='^',linewidths=7)
plt.scatter(S[:,0],S[:,1],c=sampleTag,linewidths=np.power(sampleTag+0.5,2))
plt.show()
print(SSE)
#重新计算簇中心
for i in range(k):
ClustI = S[np.nonzero(sampleTag[:]==i)[0]]
clusterCents[i,:] = np.mean(ClustI,axis=0)
return clusterCents,sampleTag,SSE
if __name__=='__main__':
samples = np.loadtxt(r"D:\360MoveData\Users\Administrator.DESKTOP-E5JRNOM\Desktop\jqxx\kmeansSamples.txt")
clusterCents, sampleTag,SSE = kMeans(samples,3)
plt.show()
print(clusterCents)
print(SSE)kmeansSamples.txt8.764743691132109049e+00 1.497536962729086341e+01 4.545778445909218313e+00 7.394332431706460262e+00 5.661841772908352333e+00 1.045327224311696668e+01 6.020055532521467967e+00 1.860759073162559929e+01 1.256729723000295529e+01 5.506569916803323750e+00 4.186942275051188211e+00 1.402615035721461290e+01 5.726706075832996845e+00 8.375613974148174989e+00 4.099899279500291094e+00 1.444273323355928795e+01 2.257178930021525254e+00 1.977895587652345855e+00 4.669135451288612515e+00 7.717803834787531070e-01 8.121947597697801058e+00 7.976212807755792555e-01 7.972277764807800260e-02 -1.938666197338206221e+00 8.370047062442882435e+00 1.077781799178707622e+01 6.680973199869320922e+00 1.553118858170866545e+01 5.991946943553537963e+00 1.657732863976965021e+01 5.641990155271871643e+00 1.554671013661827672e+01 -2.925147643580102041e+00 1.108844569740028163e+01 4.996949605297930752e+00 1.986732057663068707e+00 3.866584099986317025e+00 -1.752825909916766900e+00 2.626427441224858939e+00 2.208897582166075324e+01 5.656225833870900388e+00 1.477736974879376675e+01 -3.388227926726261607e-01 5.569311423852095544e+00 1.093574481611491223e+01 1.124487205516641275e+01 4.650235760178413003e+00 1.278869502885029341e+01 8.498485127403823114e+00 9.787697108749913610e+00 7.530467091751554598e+00 8.502325665434069535e+00 6.171183705302398792e+00 2.174394049079376856e+01 -9.333949569013078040e-01 1.594142490265068712e+00 -6.377004909329702542e+00 3.463894089865578341e+00 7.135980906743346175e+00 1.417794597480970609e+01
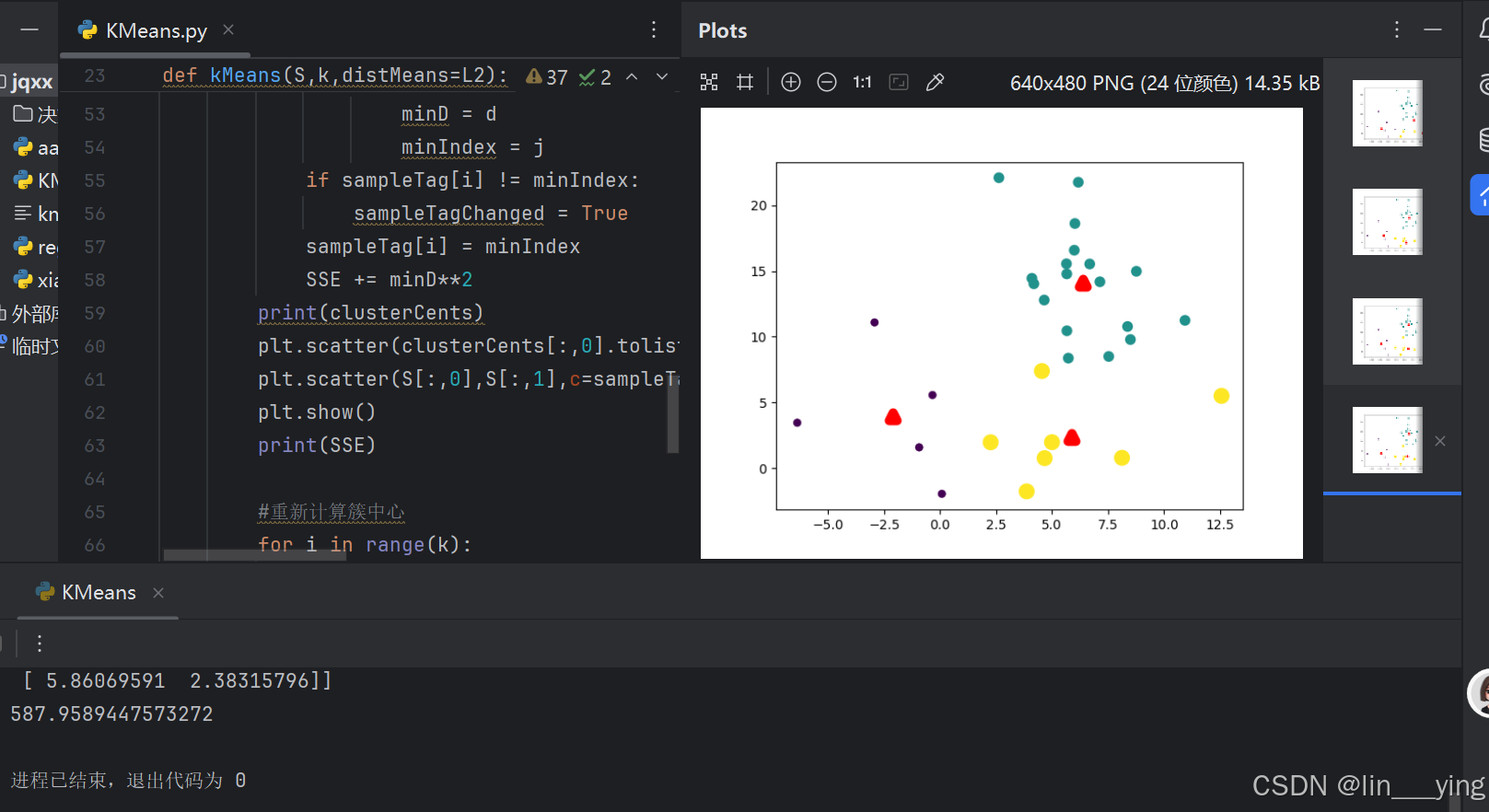
运行得到
3.k-means算法的优缺点
优点:
(1).计算复杂度低,可解释性强。算法逻辑简单直观,时间复杂度为 O(nkt) (其中 n 是样本数, k 是聚类数, t 是迭代次数),在处理中大规模数据时效率较高,且聚类结果容易理解。
(2).收敛性有保障。只要迭代次数足够,算法能保证收敛到一个局部最优解。
(3).对数值型数据适配性好。适用于连续型数值数据的聚类分析,在许多领域如客户分群、图像分割等场景中应用广泛。
缺点:
(1).需预先指定聚类数 k。实际应用中,用户往往难以提前确定最优的 k 值,若 k 选择不当,会严重影响聚类效果。
(2).对初始聚类中心敏感。不同的初始中心可能导致不同的聚类结果(局部最优而非全局最优),如前面示例中若初始中心选择不同,最终聚类可能有差异。
(3).对非球形簇、不同密度或大小的簇适应性差。仅能识别球形且密度、大小相近的簇,对于不规则形状(如环形、月牙形)或密度差异大的簇,聚类效果不佳。
(4).**对噪声和离群点敏感。**离群点会显著影响聚类中心的计算,导致聚类结果偏离真实分布。
(5).只能处理数值型数据。无法直接处理类别型或混合类型的数据,若要处理这类数据,需先进行数值化转换(如独热编码),但可能引入偏差。
二.改进算法
二分k-means算法试图克服k-means算法收敛于局部最优值的缺陷。核心思路是通过"二分拆分"逐步构建k个聚类
1.二分k-means算法流程
(1). 初始化聚类集合
将所有样本点合并为1个初始聚类,记为聚类集合 C = \{C_1\} ( C_1 包含所有样本)。
(2). 计算初始SSE
计算当前聚类集合中每个聚类的误差平方和(SSE),总SSE为各聚类SSE之和。
SSE定义:聚类内所有样本到该聚类中心的欧氏距离的平方和
(3). 迭代拆分聚类
循环执行以下步骤,直到聚类集合的大小等于目标 k :
选择待拆分的聚类:从当前聚类集合 C 中,选择SSE最大的聚类(记为 C_i )------因为SSE大意味着该聚类内部样本差异大,更适合拆分。
拆分选中的聚类:对 C_i 执行普通K-means(k=2),得到2个子聚类 C_{i1} 和 C_{i2} 。
更新聚类集合:从 C 中移除 C_i ,加入 C_{i1} 和 C_{i2} 。
更新总SSE:重新计算新聚类集合的总SSE(若总SSE降低,说明拆分有效)。
(4). 输出结果
当聚类集合的大小达到 k 时,停止迭代,输出最终的 k 个聚类。
2.k-means++算法流程
K-Means++的核心是通过"距离加权概率"选择初始中心
(1).随机选1个样本作为第一个聚类中心,避免完全无方向的初始化。
(2).计算每个样本到"已选中心"的最短距离(d(x,\mu)),衡量样本与现有中心的分散程度。
(3).用距离的平方(D(x)^2)作为权重计算选择概率(P(x)),距离越远的样本被选中的概率越高,保证初始中心的分散性。
(4).重复步骤2-3,直到选够k个初始中心,后续流程与普通K-Means一致。
3.k-medoids算法
k--medoids算法与k--means算法不同之处在于簇中心的计算方式不同。k-means:簇中心是虚拟的"均值点",由簇内所有数据点的特征均值计算得出,该点未必是数据集中的实际样本。k-medoids:簇中心是真实的"代表点"(medoid),从簇内所有实际数据点中选出,其特点是使簇内其他点到它的总距离最小。
k-medoids算法核心是选择实际数据点作为聚类中心(medoid),通过迭代替换中心来最小化簇内数据点到中心的总距离
k--medoids算法流程
(1). 初始化 :从所有数据点中随机选择k个不重复的点,作为初始medoid(聚类中心)。
(2).分配簇 :计算每个非medoid点到所有k个medoid的距离,将该点分配到距离最近的medoid所在的簇。
(3).尝试替换(Swap) :对每个簇,依次用簇内的非medoid点(候选点)替换当前medoid,计算替换后簇内的总距离(成本)。
(4).判断是否更新 :若替换后的总距离比替换前更小,则保留该候选点为新medoid;否则,维持原medoid不变。
(5). 迭代终止:重复步骤2-4,直到所有medoid都不再被替换,或达到预设的迭代次数,算法收敛。
4.Mini Batch k-means算法
Mini Batch k-means算法通过略微牺牲优化质量来取得显著减少计算时间的效果。它的基本思路是用随机抽取的代表样本来进行优化计算,而不是在全部样本上进行计算
Mini Batch k--means算法流程
(1). 初始化:随机选k个点作为初始簇中心。
(2). 抽批次:每次从全量数据中取一小部分样本(Mini Batch)。
(3). 更中心:将批次内样本分配到最近簇,用这批样本更新对应簇中心。
(4). 终止:重复2-3,直到簇中心变化极小或迭代次数达标。
三.聚类算法基础
1.聚类任务
聚类任务是无监督学习下的核心任务,核心是让算法自动将无标签数据,按"相似度/距离"分成若干组(簇),最终实现"簇内数据相似、簇间数据差异大"的效果。
2.样本点常用距离度量
(1)Lp距离
Lp距离(Lp Metric)是一类距离的通用表示框架,通过调整参数p的值,可涵盖欧氏距离、曼哈顿距离等常见距离,核心是衡量n维空间中两点的"差异程度"。
其数学定义为:

关键通过p值区分具体距离类型:
当 p=1 时,即曼哈顿距离,计算"直角折线"距离(如城市中沿街道行走的路径)。
当 p=2 时,即欧氏距离,计算"空间直线"距离(如两点间的最短路径)。
当 p→∞ 时,即切比雪夫距离,取两点各维度差值的最大值(如棋盘上国王的一步最大移动距离
(2).VDM距离
VDM距离,即值差异度量(Value Difference Metric),是一种用于衡量离散无序属性之间距离的方法,核心思想是将离散无序的数据转化为可以量化的差异度量,以进行比较和分析。在实际应用中,VDM距离常与闵可夫斯基距离结合,用于处理包含有序属性和无序属性的混合属性数据 。
对于定义域为{飞机,火车,轮船}这样的离散无序属性,不能直接在属性值上计算距离,此时可采用VDM
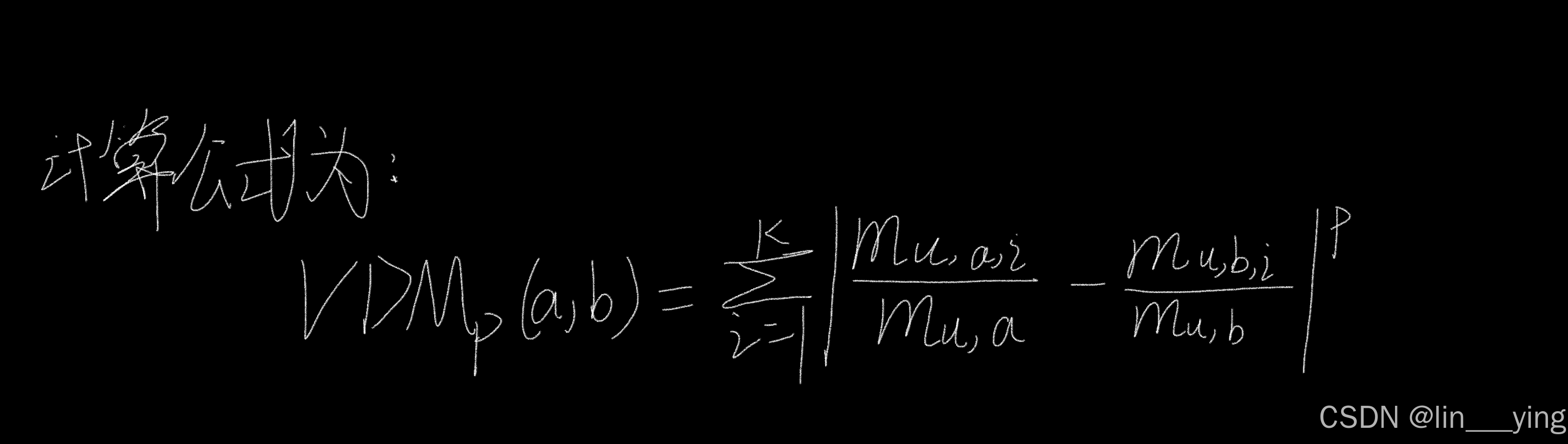
其中,m_{u,a}表示在属性u上取值为a的样本数,m_{u,a,i}表示在第i个样本簇中在属性u上取值为a的样本数,k为样本簇数。
(3).混合加权距离
混合加权距离是一种将多种距离度量方法结合起来,并为每种距离度量分配相应权重,从而计算数据点之间差异的方法,它可以更全面地反映数据点之间的相似性或差异性。

在实际应用中,混合加权距离常用于处理包含多种类型特征的数据。如在基于混合加权距离的KNN心衰患者死亡率评估模型中,就利用值差度量和曼哈顿距离混合计算样本间的距离,以提高模型性能。
(4). 余弦相似度
余弦相似度是衡量两个向量在空间中夹角余弦值的指标,核心用于判断向量方向的相似性,值越接近1表示方向越一致(相似度越高),越接近-1则方向越相反。

3.聚类算法评价指标
1.外部指标
(1).Jaccard系数(JC)
Jaccard系数(JC,又称交并比)是衡量两个集合相似度的指标,核心通过"交集大小与并集大小的比值"量化重合程度,广泛用于聚类、分类、文本匹配等场景。

(2).FM指数(FMI)
FM指数(Fowlkes-Mallows Index,FMI)是一种用于聚类结果外部评价的指标,核心通过"精确率"与"召回率"的几何平均,量化聚类分组与真实标签的匹配程度。
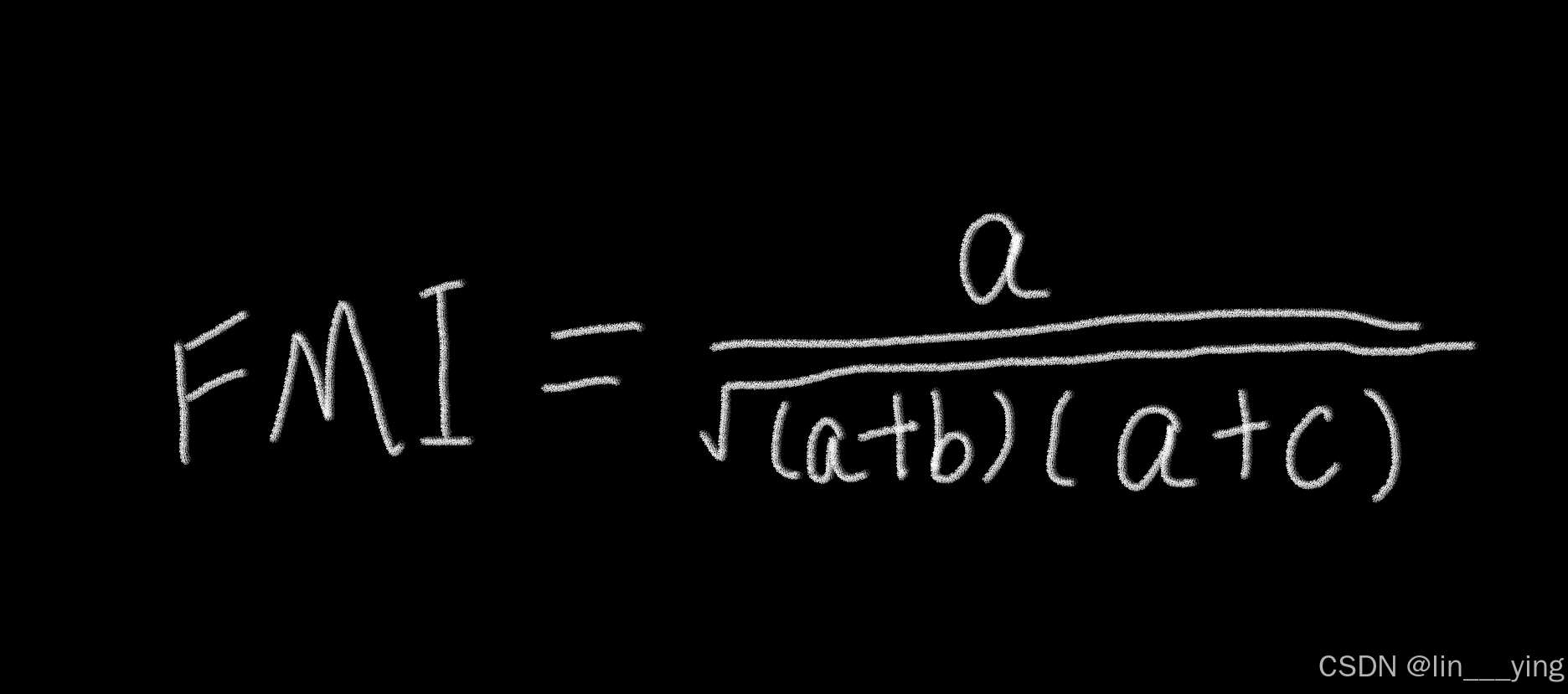
其中,a+b是出现在同一预先分组中样本对数,a+c是出现在同一算法分簇中的样本对数。该指数取值于0~1,值越大,算法分簇与预先分组越接近,值越小,算法分簇与预先分组越不相关。
2.内部指标
若某聚类算法给出的分簇C={C1,C2,C3,...,Ck},定义
(1) 样本xm与同簇Ci其他样本的距离:

该距离也称为xm的簇内平均不相似度
(2) 样本xm与 不同簇Cj内样本的平均距离:

该距离也称为xm与簇Cj的平均不相似度
(3).样本Xm与簇的最小平均距离:
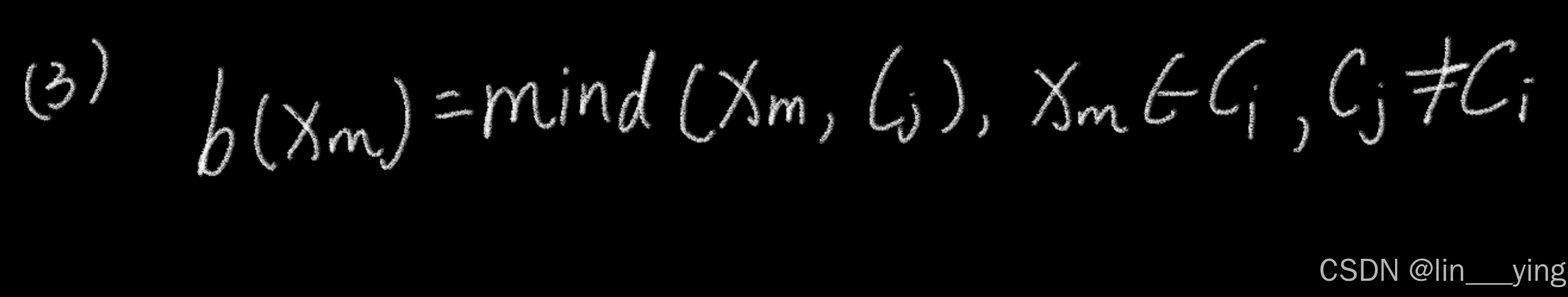
该距离是取xm与所有其他不同簇的平均距离中的最小值
(4).簇内样本平均距离:

(5).簇内样本最大距离:
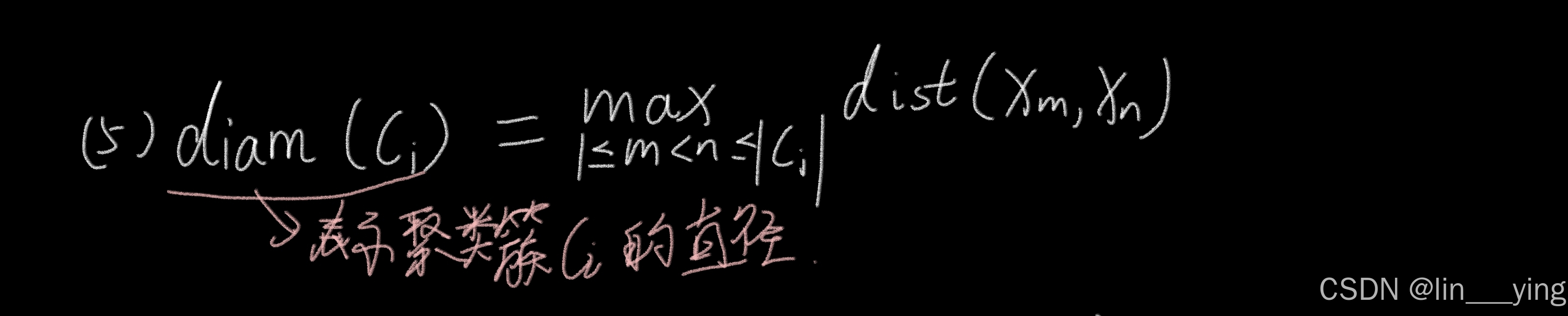
(6).簇最小距离:
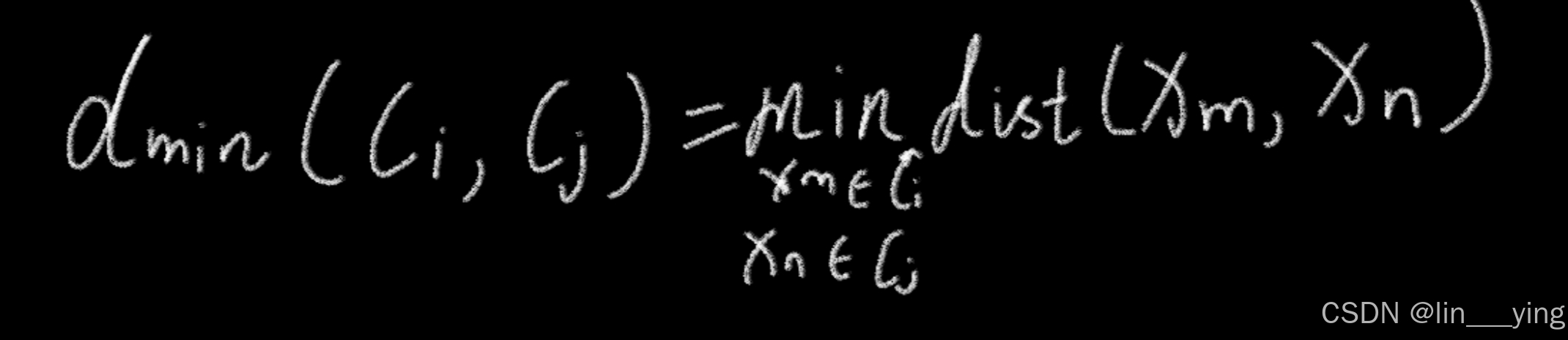
(7).簇中心距离:
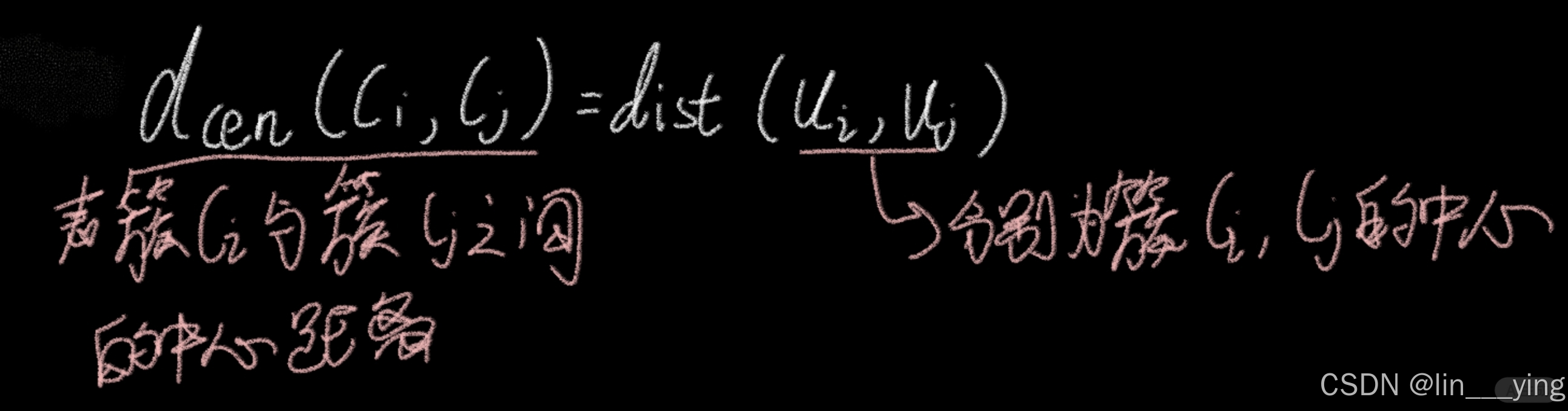
根据以上距离,可定义以下内部评价指标
(1)轮廓系数(SC)
单一样本xm的轮廓系数为:

一般使用的轮廓系数是对所有样本的轮廓系数取均值。CS值高表示簇内密集,簇间疏散。
(2).DB指数(DBI)
DB指数,即Davies-Bouldin Index,是一种用于评估聚类算法性能的内部指标。
它基于簇内的紧密性和簇间的分离性来衡量聚类的质量,其值越小,说明聚类效果越好。

(3).Dunn指数(DI)
Dunn指数(DI)是评估聚类效果的指标,通过计算"类间最小距离"与"类内最大直径"的比值,取各聚类对应值的最小值。其核心逻辑是衡量类间分离度与类内紧致度,数值越大代表聚类结果越优,适用于K-means等算法的结果评估。
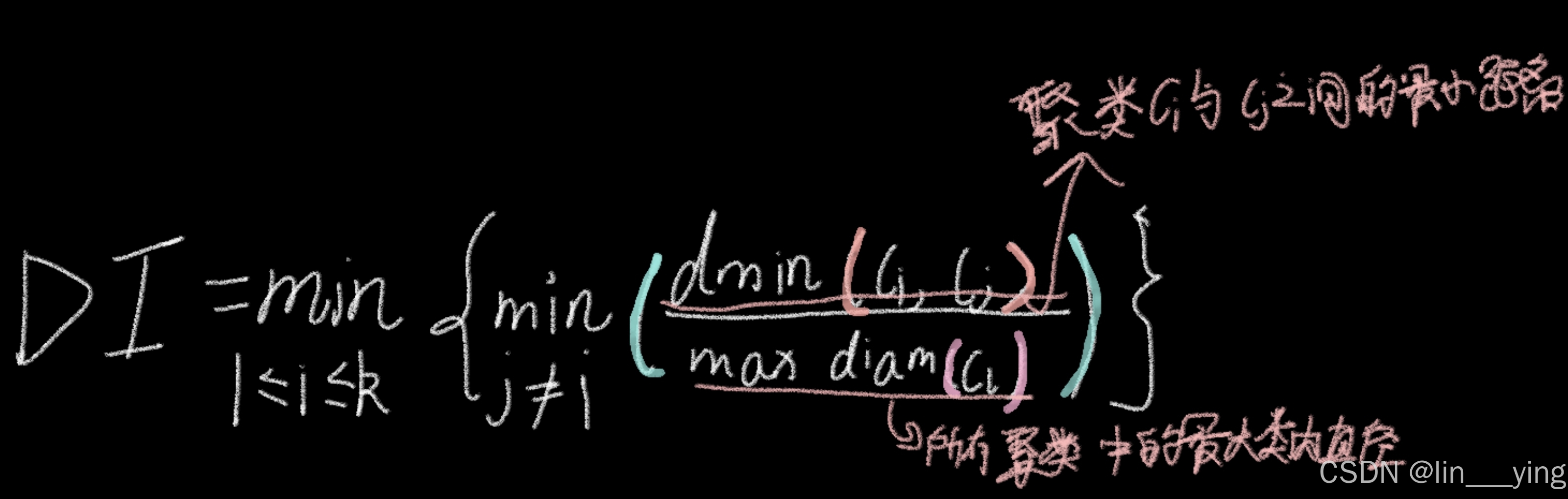
4. 聚类算法分类
(1).划分聚类
核心:将数据划分为预设数量的簇,通过迭代优化簇内相似度。
典型算法:K-means(基于距离的簇中心更新)、K-medoids(抗噪声,用实际样本点作中心)
(2).密度聚类
核心:基于样本密度划分簇,能发现任意形状的簇,自动识别噪声。
典型算法:DBSCAN(通过ε邻域和最小点数定义核心点)、OPTICS(优化DBSCAN的参数敏感性)
(3).层次聚类
核心:通过"凝聚(从单个样本开始合并)"或"分裂(从全样本开始拆分)"生成聚类树。
代表算法:AGNES(凝聚式)、DIANA(分裂式)。
(4).网络聚类
核心:依赖网络拓扑结构(而非节点属性),需同时考虑节点连接的"密度""权重""方向"(如无向/有
向图);部分场景会结合节点属性(如用户画像)优化聚类效果。
典型算法:GN算法(Girvan-Newman)、Louvain算法
(5).模型聚类
核心:基于概率模型或生成模型的聚类方法
典型算法:高斯混合模型(Gaussian Mixture Model, GMM)、贝叶斯高斯混合模型(Bayesian Gaussian Mixture Model)、隐马尔可夫模型(Hidden Markov Model, HMM)
四.DBSCAN及其派生算法
DBSCAN是经典的密度聚类算法
1.密度相关概念
(1).ε-邻域
对于数据集中的任意样本点 p ,以 p 为中心、以预设距离阈值ε 为半径的区域内,所有包含的样本点的集合,称为 p 的 ε-邻域,记为 Nε(p)

ε是DBSCAN算法需要指定的两个参数之一
(2).核心点和边界点
核心点:DBSCAN中,若点的ε-邻域内样本数≥预设最小点数阈值MinPts,该点为核心点,是聚类扩展的基础
边界点:自身非核心点,但落在某核心点的ε-邻域内,属于聚类却不驱动聚类生长
简而言之,核心点是"高密度核心",边界点是"核心周围的低密度边缘",二者共同构成DBSCAN的聚类结构
(3).直接密度可达
若点q在核心点p的ε-邻域内,且p是核心点(ε-邻域样本数≥MinPts),则q从p直接密度可达,是密度关系的"直接连接"
(4).密度可达
若存在点链p→p₁→p₂→...→q,每相邻两点直接密度可达,且p是核心点,则q从p密度可达,体现"传递性",是单向关系
(5).密度相连
若存在核心点o,点p和q均从o密度可达,则p与q密度相连,是双向关系,同一聚类内的点彼此密度相连。
2.DBSCAN算法流程
- 设定核心参数 :确定两个关键参数------ε(epsilon,邻域半径) 和 MinPts(邻域内最少样本数),这是算法的基础
- 识别核心点 :遍历所有样本点,若某点邻域(以该点为中心、ε为半径)内的样本数 ≥ MinPts,则标记为核心点;反之则为非核心点
- 扩展密度簇 :从任意未分类的核心点出发,将其所有直接/间接密度可达的样本(通过核心点连接的点)归为同一簇,并标记簇内所有点
- 标记噪声点:遍历完所有核心点后,剩余未被任何簇包含的非核心点,即为噪声点(异常值)
3.OPTICS算法
OPTICS(Ordering Points To Identify the Clustering Structure)算法是一种基于密度的聚类算法,它可以看作是DBSCAN算法的扩展。与DBSCAN直接输出聚类结果不同,OPTICS通过计算样本点的可达距离(reachability distance)和核心距离(core distance),生成一个有序的点序列和对应的可达距离图,从而帮助用户识别不同密度的聚类结构
-
核心距离(Core Distance):对于给定的参数 min_samples ,样本点成为核心点所需的最小距离(即第 min_samples 个最近邻的距离)。若样本点不是核心点,则核心距离未定义
-
可达距离(Reachability Distance):从核心点 p 到点 q 的可达距离,定义为 max(核心距离(p), 距离(p, q)) 。若 p 不是核心点,则可达距离未定义
OPTICS算法流程
初始化所有点为"未处理"状态
遍历每个未处理的点 p :
▪ p 是核心点,计算其所有 ε 邻域内的点,生成一个有序的种子列表(按可达距离排序)。
▪从种子列表中选取可达距离最小的点 q ,标记为"已处理",并将其加入结果序列。
▪若 q 是核心点,计算其邻域内点相对于 q 的可达距离,更新种子列表。
3. 重复步骤2,直到所有点都被处理,生成最终的有序序列和可达距离
4.AGNES算法
(1).簇之间的距离变量
①簇最小距离
簇最小距离是两个簇成员之间的最小距离
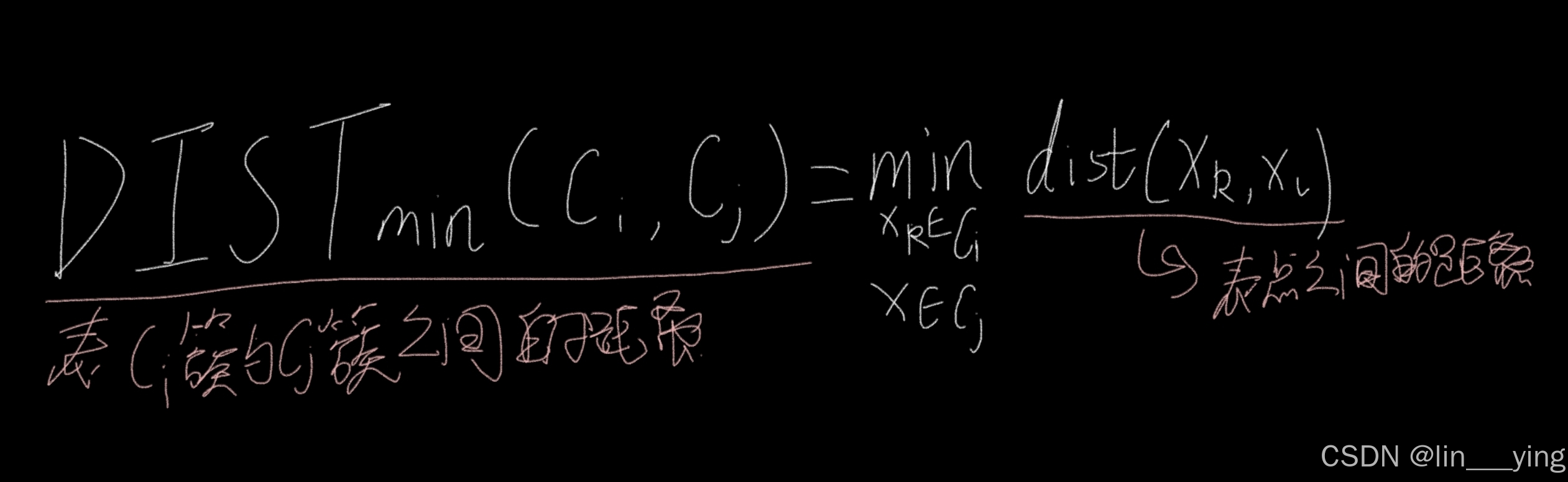
②簇最大距离
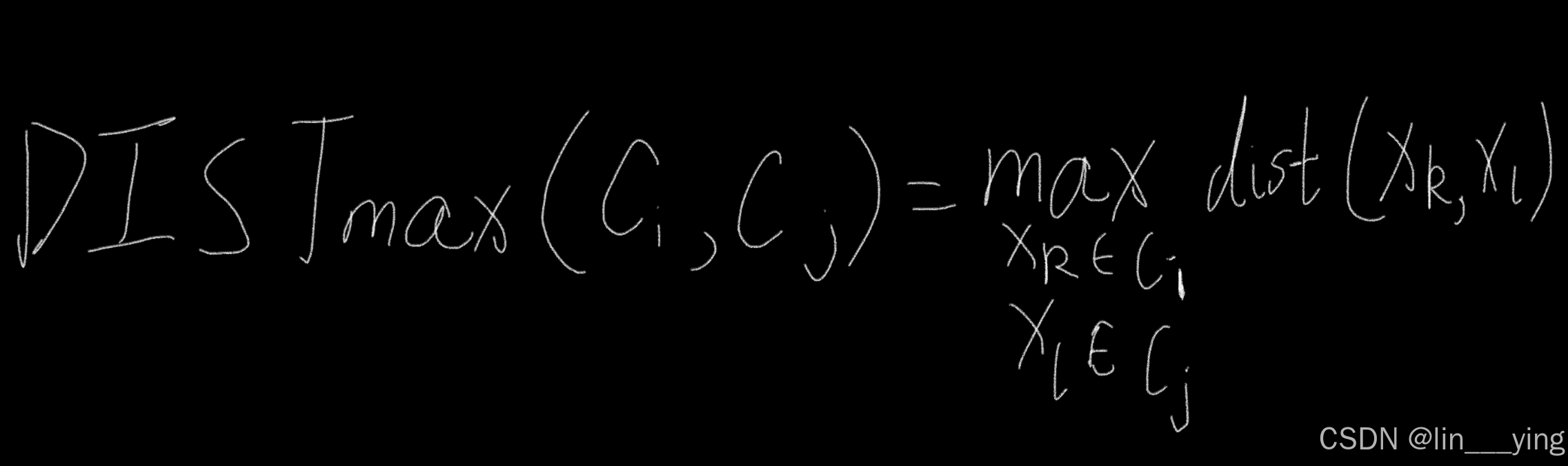
③簇平均距离
簇平均距离是两个簇成员之间距离的平均值
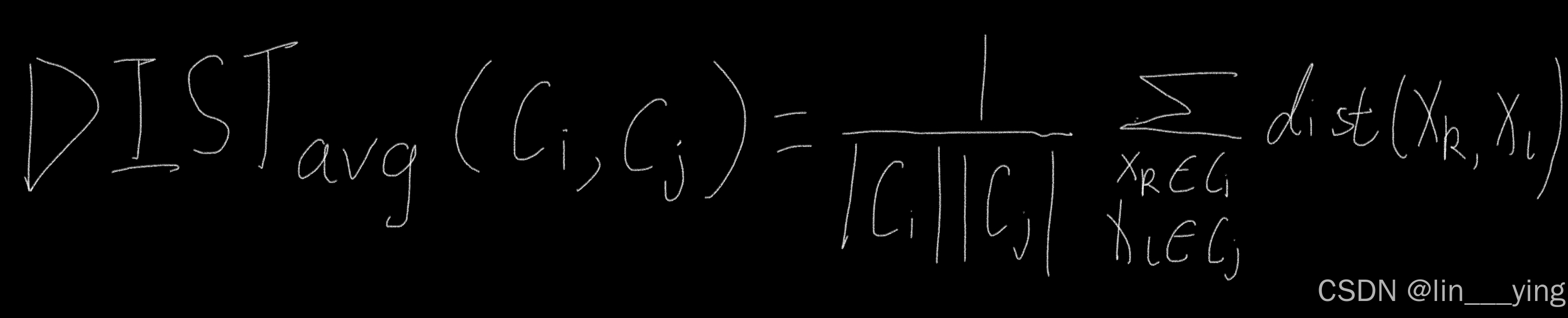
④簇中心距离
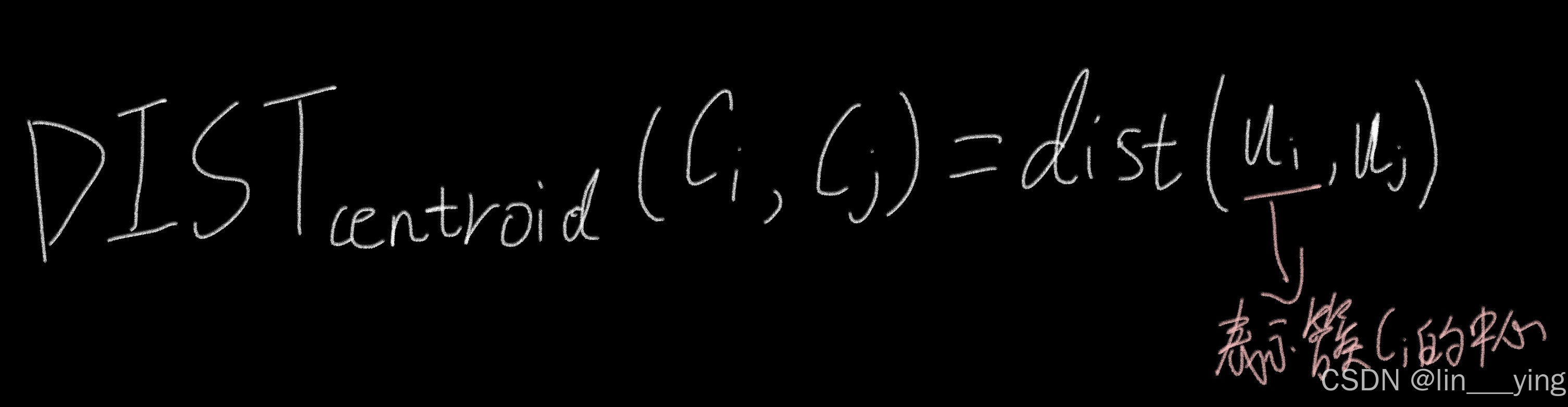
(2).算法流程
AGNES(Agglomerative Nesting)是一种自底向上的层次聚类算法,其核心流程是从每个样本为单个簇开始,不断合并距离最近的簇,直到满足停止条件(如达到预设簇数)
1 .初始化簇
将数据集中的每个样本都视为一个独立的簇,即初始簇的数量等于样本数量 n ,记为 {C1,C2,...,Cn } 。
2.计算簇间距离矩阵
根据选择的簇间距离度量(如单链接、全链接、平均链接、重心距离等),计算所有簇对之间的距离,生成一个 n × n 的距离矩阵。
3.合并距离最近的两个簇
在距离矩阵中找到距离最小的两个簇 Ci 和 Cj ,将它们合并为一个新的簇 Cnew = Ci ∪ Cj 。
4:更新簇集合和距离矩阵
从簇集合中移除 Ci 和 Cj ,并将新簇 Cnew 加入簇集合。
重新计算新簇与所有剩余簇之间的距离,更新距离矩阵。
5.重复合并过程
重复步骤3和步骤4,直到满足停止条件(常见条件:簇的数量达到预设值、簇间最小距离超过某个阈值、所有样本合并为一个簇)。