标签:#overlay #展示脑区 #高亮指定脑区 #脑区展示
目标:利用matlab、DPABI、mricron等工具展示选定脑区
方法一.matlab运行此程序
matlab
% 将特定脑区用颜色标注出来
% 选择D:\Software\Matlab\toolbox\DPABI_V2.2_161201\Templates 里面的aal文件
% 通过改变标签代码可改变颜色 或者直接替换颜色
function color
% 选择载入的图谱
% file = spm_select(inf,'image','select the files');
file = spm_select(1,'image','select the atlas');
V = spm_vol(file(1,:));
% 读取载入的图谱
img = spm_read_vols(V);
% 需要展示的脑区标签编号
img_label = [73 74 75 76 77 78];
% 将图谱中需要展示的脑区保留,其他脑区置0
img_label_select = ismember(img,img_label);
not_label = ~img_label_select;
img(not_label)=0;
% 二值化显示
% img(img~=0)=1;
% 更改标签编号以调整显示颜色
% img(img==73)=33;
% img(img==74)=34;
% 导出修改好的图像
V.fname = 'aalnew.nii';
spm_write_vol(V, img);载入脑图谱,img_label换成自己所需的脑区编号。
方法二.利用mricro的overlay功能进行叠加展示。
需要先将原始图像底版reslice 成与图谱相同大小。
可通过更改标签编号以调整显示颜色。
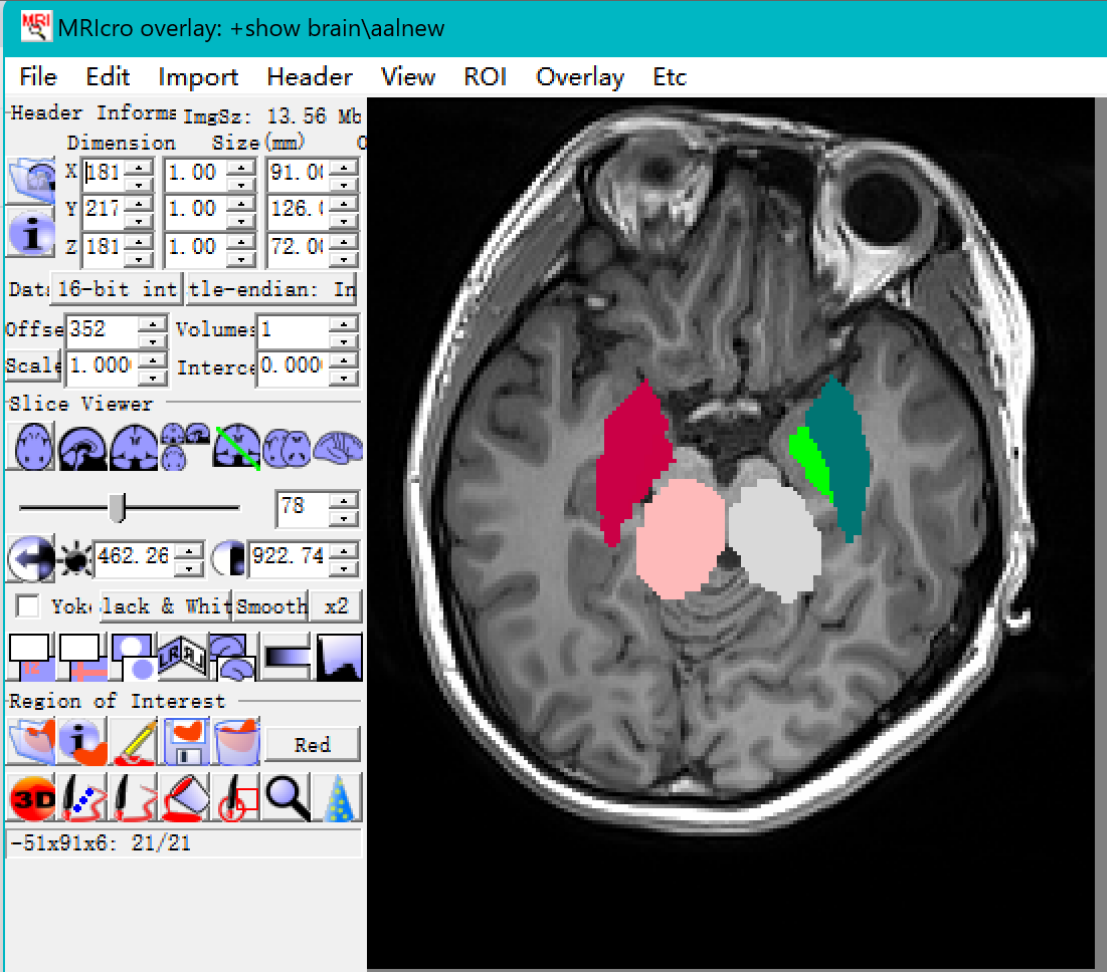
其他方法:
(1).利用dpabi/restplus的image calculator ,输入公式
matlab
(i1==37)|(i1==38) 37,38是海马脑区的标签。
|是matlab逻辑运算符,代表或运算。 (&与,|或,~非)
i1== 19|i1==20 %%可以不加括号,等价。
(2).打开mricron-win,利用overlay进行叠加显示。
只有一个脑区时,也可以用mricro叠加显示。
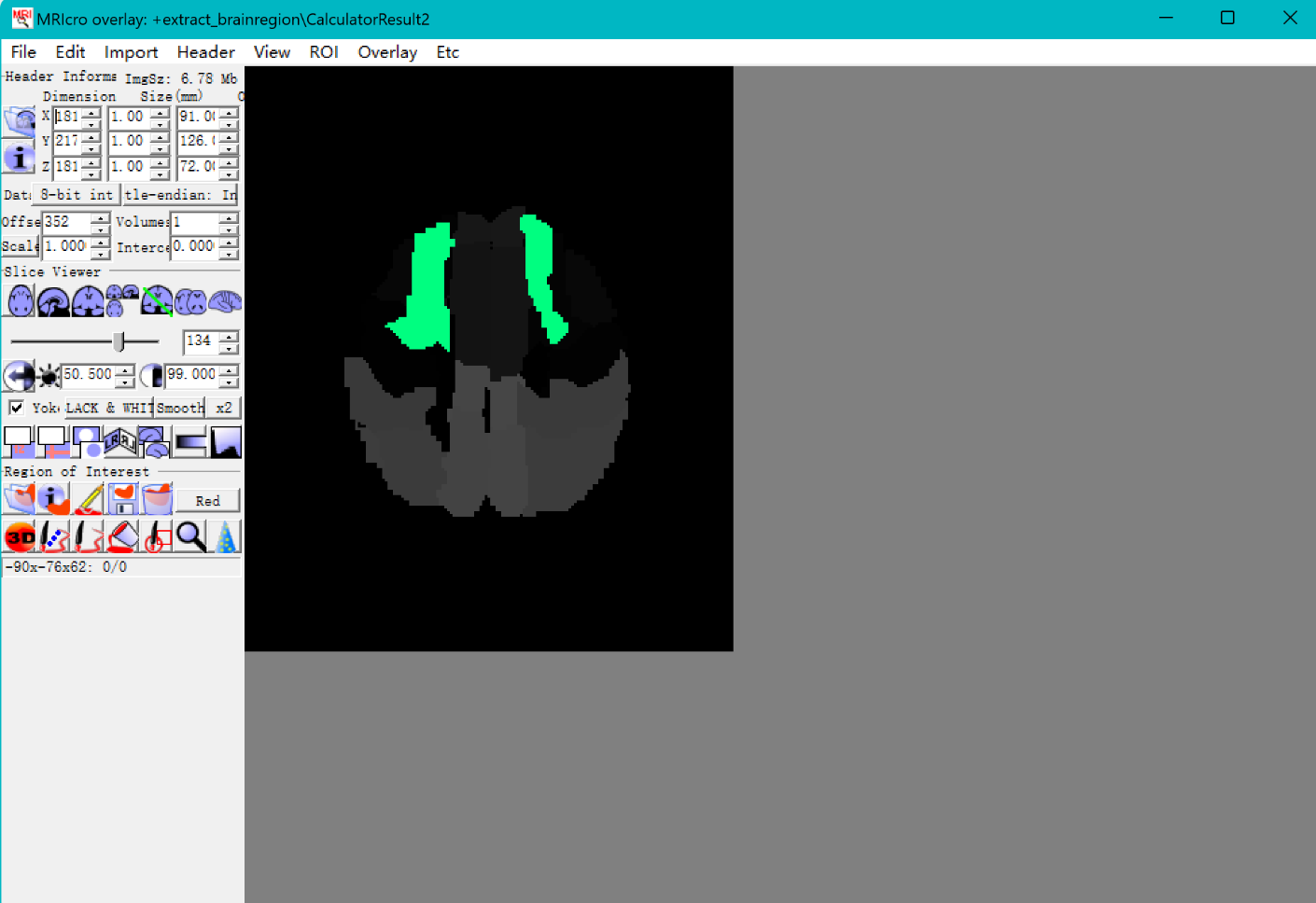
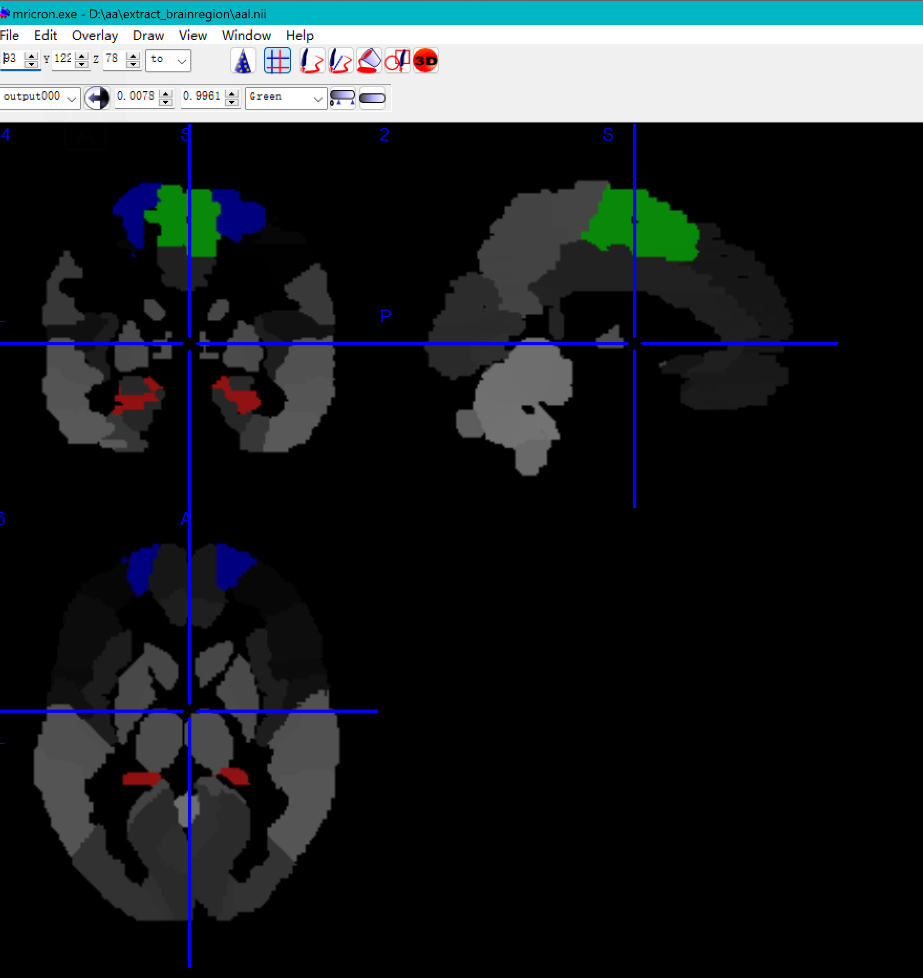
多个脑区时,先分别保存,再一个个的叠加。
改变colormap来改变色彩模式。
设置透明度来高亮overlay的脑区。