第一章:简介
现有的基于深度学习的RNA二级结构预测方法通常与动态规划或热力学方法相结合,以提高预测精度。LSTM[15]或Transformer[16]等结构被用于捕获核苷酸之间的远程相互作用。然而,这些现有的深度学习方法仍然面临着一些挑战:首先,LSTM和Transformer模块通常都涉及大量的模型参数,导致计算成本高,效率低,严重影响模型的性能。其次,当处理像lncRNAs这样长度通常超过200个核苷酸且结构复杂的套结构(如假结)方面表现不足。最后,由于不同家族的RNA序列具有保守性和同源性,传长RNA序列时,传统方法难以准确捕获核苷酸之间的远程相互作用,并且在预测RNA序列中的非嵌统方法对跨家族RNA二级结构预测缺乏足够的泛化能力,无法有效处理不同家族RNA结构的复杂性和多样性。
为了解决上述问题,本文提出了基于状态空间模型的端到端RNA二级结构预测模型SFMFold。该模型包括RNA序列表示学习和RNA结构表示学习两种并行结构,分别将RNA的序列信息和结构信息作为模型的输入。通过将输入RNA序列转换为16通道2D"图像",而不仅仅依赖于核苷酸序列,我们使模型能够明确地考虑所有潜在的碱基对和远程相互作用,并且我们使用选择性融合曼巴模块(SFMamba)进行RNA序列表示学习。它允许模型灵活地选择基配对特征信息,以优化预测性能。为了模拟RNA分子的结构,我们通过使用图神经网络将每个RNA分子表示为图来获得图级表示。最后,采用多模态对比学习方法对学习到的两种模态特征进行合并对齐,并使用多模态融合曼巴模块(MFMamba)对两种特征进行深度融合,最终得到RNA分子的最终预测图。
综上所述,我们提出的模型的贡献如下:
- 在RNA序列表示学习过程中,设计SFMamba模块捕获RNA序列中的远程依赖关系,根据碱基配对特征动态调整局部特征和全局特征的融合策略。
- 在RNA结构表示学习过程中,利用GCN获取RNA分子的图级表示,捕获RNA结构中的拓扑性质和碱基配对信息。
- 采用多模态对比学习框架整合序列特征和结构特征,使同一RNA分子在不同模态下的特征距离最小,使不同RNA分子之间的特征距离最大,提高了RNA二级结构预测的准确性。
- 设计了MFMamba模块,深度融合了两种不同模式的特征,能够捕捉序列和结构特征之间的复杂关系,从而增强了模型的泛化能力,提高了模型在跨家族RNA二级结构预测任务中的性能。
第二章:相关工作
第三章:材料和方法
3.2.模型架构
所提出的RNA二级结构预测模型的整体结构如图2所示。该模型由RNA序列表示学习、RNA结构表示学习和多模态对比学习三部分组成。首先,我们使用选择性融合曼巴模块(SFMamba)从输入RNA序列图中提取序列特征,以获得RNA序列之间的长距离相互作用。其次,我们使用图卷积网络来模拟RNA的结构特征。最后,采用多模态对比学习方法,最大化序列特征和结构特征之间的一致性,对齐多模态信息,提高模型的泛化能力。我们使用多模态融合曼巴模块(MFMamba)来融合这两个特征并得出最终的预测图。
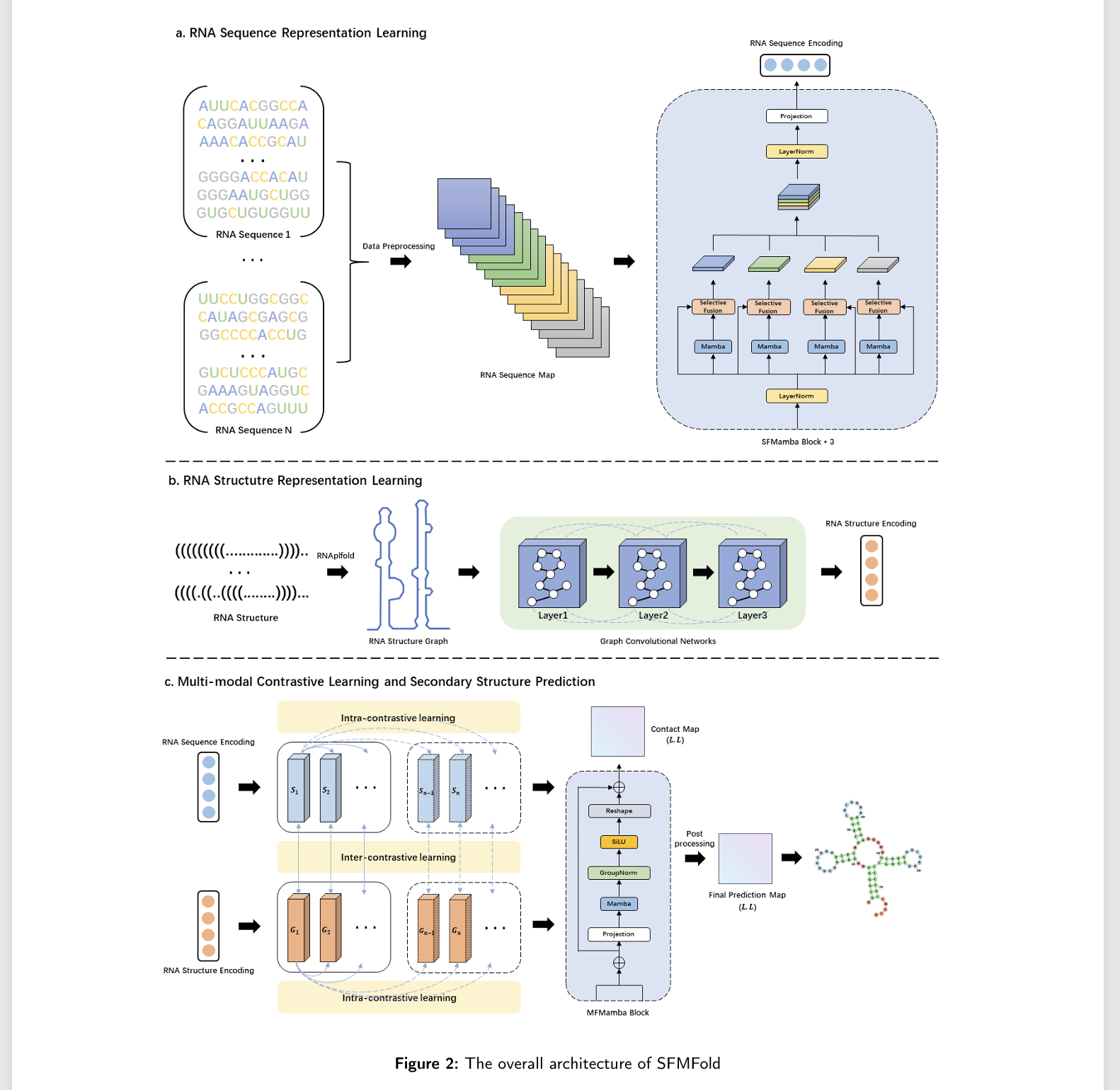
3.3.RNA序列表示学习:
RNA二级结构预测是对给定RNA序列的基本配对模式进行预测。传统的方法大多将RNA序列𝑆= (s1, s2,...s𝐿),s𝐿∈{mdis,𝑈,<e:1>,𝐺}直接作为模型的输入,在处理RNA长序列时难以捕捉序列中的长距离依赖关系。UFold介绍了一种将RNA序列转化为"图像"的新方法。与UFold一样,我们表示每个核苷酸由一个炎热的编码向量,𝑆转换为一个𝐿×4二进制矩阵𝑋∈{0,1}𝐿×4,然后执行克罗内克积𝑋获得一𝐿×𝐿×16张量,表示𝐾∈{0,1}𝐿×𝐿×16。通过将RNA序列表示为16通道图像来考虑每个碱基对(包括规范的和非规范的),其中每个通道表示一个碱基对,如图3所示。它能够模拟RNA序列中核苷酸之间的各种远距离相互作用,作为图像中的局部模式。
考虑到局部配对的细节特征和全局配对的宏观特征,我们使用SFMamba Block对RNA序列信息进行处理。数据预处理后的特征图谱根据碱基配对分为四组,分别输入到曼巴分支中,曼巴分支以其独特的机制捕获RNA序列中的长距离依赖关系。Mamba模块的具体结构如图4所示,主要包含两个分支结构。第一个分支首先通过Linear层将输入数据的各个特征映射到输出数据对应的特征上,通过卷积运算对序列数据进行处理,并应用SiLU激活函数增强特征表示。然后将这些处理过的特征输入到选择性状态空间模型(SSM)中,这是Mamba模块的核心。它类似于RNN中的递归结构,可以有效地对长序列进行建模,并且在序列长度上呈线性扩展。这种选择性机制允许模型根据当前输入有选择地传播或忘记信息,从而提高模型处理RNA序列等密集数据的性能。
第二分支对输入特征进行线性投影,并由SiLU激活函数处理;该分支不经过SSM层,其主要作用是增强特征表示。最后,对两个分支的输出特征进行合并,合并后的特征包含了更丰富的信息,同时保留了各分支特征的相对重要性。
我们使用选择性融合模块将局部特征与曼巴模块处理后得到的全局特征进行融合。该模块根据局部特征和全局特征的重要程度分别赋予权重,保证了模型在提取RNA序列特征时既考虑了局部配对信息,又考虑了全局配对信息。首先对特征进行维数变换,得到局部特征𝐹1∈算子𝐿×𝐿×4和全局特征𝐹2∈算子𝐿×𝐿×4,然后将它们连接起来,得到特征<s:1>𝑐∈算子𝐿×𝐿×8。随后,通过全连通层进一步对其进行处理,得到局部特征的权值为时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延时延。这些权重分别乘以𝐹1和𝐹2,然后通过元素加法融合。SFMamba模块根据输入特征的特征动态调整权值,使模型能够自适应选择最有利于当前任务的特征信息,更灵活地处理不同长度的RNA序列。
3.4.RNA结构表征学习
RNA序列的边缘连接RNA分子内的相邻节点,有效地表示核苷酸顺序并捕获RNA分子初级结构的线性排列;这一序列信息对于理解RNA分子的初级结构至关重要。大量研究表明,RNA分子的二级拓扑结构显著影响其功能。因此,除了序列信息外,我们还对RNA的二级结构特征进行了建模。RNA二级结构可以用图形形式表示,其中顶点表示单个核苷酸,边缘表示它们的相互作用。图卷积网络(GCN)擅长封装核苷酸的局部环境并在整个序列中传播信息,这是理解RNA二级结构所必需的过程。它通过特征嵌入和邻域聚合来捕获RNA二级结构的独特模式。这些网络的多层卷积架构有助于同时学习局部和全局上下文,从而提高模型对RNA分子结构的预测精度。GCN作为破译RNA序列拓扑性质和碱基配对的有力工具,已广泛应用于各种分类和预测任务中。
一个RNA分子可以表示为一个图形𝐺= (s, s),其中,s表示RNA分子内核苷酸的节点集,s表示这些节点之间各种相互作用的边缘集。RNA图中的每个核苷酸节点u与一个特征向量<s:1>𝑢∈𝑅𝑑相连,该特征向量包含当前和邻近的核苷酸类别、是否属于形成伪结的节点、一组物理或化学性质等相关信息。RNA图能够保留对理解RNA分子的结构和功能至关重要的细粒度信息。对于图中的每个核苷酸𝑢 ̄,我们初始化它的表示为 ̄0 ̄。然后,我们使用𝑘layer GCN逐层计算和更新每个节点表示。更新后的隐藏状态ℎ𝑘1𝑖节点𝑢𝑖层𝑘1计算如Eq.1所示,𝜎激活函数,𝑁(𝑢𝑖)是一组相邻节点𝑢𝑖,𝑐𝑖𝑗节点之间边的重量𝑢𝑖和其他相邻节点𝑢𝑗,和𝑊𝑘𝑘矩阵可学的重量是在层。在计算每个节点表示的最后一层后,我们通过读出操作获得RNA的图级表示Gn,该操作捕获了RNA的结构信息,如图1所示。其中Readout(⋅)表示将RNA图中每个节点的表示转换为一个全面的𝑑-dimensional表示的操作,从而捕获RNA图的整体结构。
3.5. 多模态对比学习策略
我们使用对比学习作为RNA的两种不同模式(序列特征和结构特征)之间的桥梁,捕捉它们的一致性,以减少模式之间的异质差异。通过最小化相同RNA在嵌入空间中不同形态之间的差异和最大化不同RNA在嵌入空间中的差异来改进特征融合。
我们使用对比学习作为RNA的两种不同模式(序列特征和结构特征)之间的桥梁,捕捉它们的一致性,以减少模式之间的异质差异。通过最小化相同RNA在嵌入空间中不同形态之间的差异和最大化不同RNA在嵌入空间中的差异来改进特征融合。其中𝐿𝑆和𝐿𝐺分别表示序列特征的对比学习损失函数和结构特征的对比学习损失函数,<s:1>(2)表示RNAsamples的总数,𝑆和𝐺分别表示序列和结构特征向量,它们是模型从输入数据中学习到的表示。𝛿是对比学习中用于控制softmax函数的超参数。𝛿值越小,softmax函数的输出越清晰,越倾向于选择正样本。较大的𝛿值将使输出更平滑,减少对正样本的偏好。
其中𝐿𝑆和𝐿𝐺分别表示序列特征的对比学习损失函数和结构特征的对比学习损失函数,<s:1>(2)表示RNAsamples的总数,𝑆和𝐺分别表示序列和结构特征向量,它们是模型从输入数据中学习到的表示。𝛿是对比学习中用于控制softmax函数的超参数。𝛿值越小,softmax函数的输出越清晰,越倾向于选择正样本。较大的𝛿值将使输出更平滑,减少对正样本的偏好。
在将对比学习应用于序列和结构表征之后,我们采用MFMamba Block将这两种表征融合,将序列和结构特征投影到一个共享的潜在空间中。网络最终输出一个𝐿×𝐿矩阵,然后乘以它的转置,使对称矩阵作为接触评分矩阵𝑈。我们使用的预测损失函数是通过随机梯度下降训练的二元交叉熵损失,以最小化接触分数矩阵𝑈和真配对矩阵之间的损失。模型的最终损失是这两个损失的加权和,其中,ρ n = 1, ρ n和ρ n是调节对比学习损失的贡献及其对整个模型的贡献的超参数。
3.6. 后处理
为了确保输出满足RNA配对约束,我们添加了一个后处理网络来过滤非标准碱基对,从而得到最终的RNA二级结构矩阵。后处理算法的具体实现见算法1。后处理考虑到RNA二级结构的三个硬约束: