
小编荐语:这个数据库厉害了! 基因预测模型终于有了"统一标尺"!不仅能给全基因组的错义突变打分,还特别擅长在没做trio测序的情况下揪出致病变异------对临床医生来说简直是省钱又省力的神器。
模型简介
错义变异因其微妙且依赖上下文的影响,在遗传解释中仍是挑战。尽管当前预测模型在已知疾病基因中表现良好,但其评分未在蛋白质组中校准,限制了普遍性。为弥补这一知识空白,研究人员开发了popEVE------一种结合进化和人类群体数据的深度生成模型,以估计全蛋白质组范围内变异的有害性。popEVE在不高估有害变异负担的情况下实现了最先进的性能,识别出严重发育障碍队列中442个基因的变异,其中包括123个新候选基因。这些基因在功能上与已知疾病基因相似,其变异通常定位于关键区域。令人惊讶的是,popEVE能够利用子外显子优先识别可能的因果变异,即使不进行父母测序也能实现诊断。研究为罕见病变异的解释提供了可推广的框架,特别是在单例病例中,并展示了校准的进化知情评分模型在临床基因组学中的实用性。
数据库
popEVE是一个用于预测变异严重程度和致病性的计算模型。它结合了物种间的变异和人类群体内部的变异,使整个蛋白质组的分数可比,同时最大限度地减少了种群偏差。虽然多个模型提供基因组尺度预测,popEVE是首个专门设计用于校准分数以实现跨基因可比的模型。
一个基因的测试
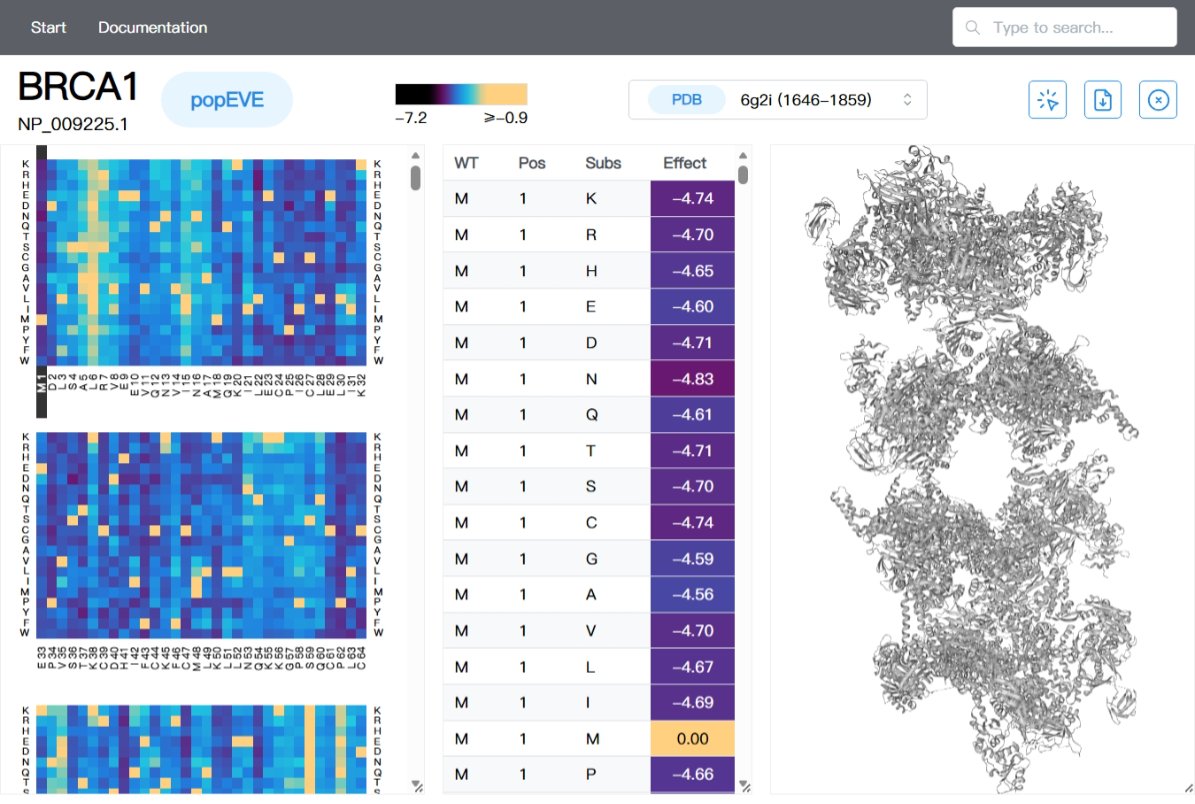
选取了个最经典的基因进行了搜索,看了下结果,左边是热图,中间是变异信息,右边是三维结构。
BRCA1 蛋白(NP_009225.1) 全部可能错义突变进行功能影响评分(Effect score) 的可视化结果,深蓝色:强负效应(deleterious,高危),黄色:接近 0(接近 wild type,无显著影响),越蓝表示突变越可能影响蛋白功能。
批量下载

数据库提供供下载 (bulk download) 的数据资源,包括:
- 按 RefSeq 转录本 ID (transcript ID) 排列的 tab-separated (TSV) score 文件 (即 popEVE 对所有 /大量蛋白及其可能突变所打分的表格)
- 按 GRCh38 染色体 (chromosome) 列出的 TSV 文件。
- 完整的 GRCh38 版本 (VCF 格式) ------ 带 popEVE 分数 (VCF with popEVE scores) 。也就是说你可以把整个基因组 /外显子组 (exome) 的变异数据进行注释 /打分。
参考: