关于RNA_seq和Ribo_seq技术的对比和BAM生成
- [1. RNA_seq和Ribo_seq技术基础概念解析](#1. RNA_seq和Ribo_seq技术基础概念解析)
-
- [1.1 Riboseq和RNAseq都是什么](#1.1 Riboseq和RNAseq都是什么)
- [1.2 什么是最长CDS,有什么用](#1.2 什么是最长CDS,有什么用)
- [1.3 什么是可变剪切,为什么要分析密码子使用偏好性](#1.3 什么是可变剪切,为什么要分析密码子使用偏好性)
-
- [1. 什么是可变剪切?](#1. 什么是可变剪切?)
- [2. 为什么要分析密码子使用偏好性?](#2. 为什么要分析密码子使用偏好性?)
- [2. RNA-seq与Ribo-seq流程差异解析](#2. RNA-seq与Ribo-seq流程差异解析)
-
- [2.1 解释一下相同的FASTQ,如何获得RNAseq的bam和Riboseq的bam,流程是什么](#2.1 解释一下相同的FASTQ,如何获得RNAseq的bam和Riboseq的bam,流程是什么)
-
- [🔍 理解流程的关键差异](#🔍 理解流程的关键差异)
- [📝 核心操作命令举例](#📝 核心操作命令举例)
- [💎 重要补充与提醒](#💎 重要补充与提醒)
- [2.2 这两者的BAM有什么区别,什么是参考基因组,什么是最长CDS,如何生成的bam](#2.2 这两者的BAM有什么区别,什么是参考基因组,什么是最长CDS,如何生成的bam)
- [🧬 参考基因组 (Reference Genome)](#🧬 参考基因组 (Reference Genome))
- [📊 RNA-seq与Ribo-seq BAM的区别](#📊 RNA-seq与Ribo-seq BAM的区别)
- [🧬 最长CDS (Longest CDS)](#🧬 最长CDS (Longest CDS))
- [🛠️ BAM文件的生成过程](#🛠️ BAM文件的生成过程)
-
- 核心步骤:
- [🔍 技术细节:BAM文件包含什么?](#🔍 技术细节:BAM文件包含什么?)
- [🎯 实际分析中的工作流程](#🎯 实际分析中的工作流程)
-
- [RNA-seq BAM生成:](#RNA-seq BAM生成:)
- [Ribo-seq BAM生成:](#Ribo-seq BAM生成:)
- [💡 关键理解点](#💡 关键理解点)
- [2.3 最长CDS有什么用,也可以用来生成BAM吗](#2.3 最长CDS有什么用,也可以用来生成BAM吗)
- [🎯 最长CDS的用途](#🎯 最长CDS的用途)
-
- 主要用途:
-
- [1. **读数定量 (Read Counting)**](#1. 读数定量 (Read Counting))
- [2. **翻译效率分析 (Ribo-seq特有)**](#2. 翻译效率分析 (Ribo-seq特有))
- [3. **核糖体足迹分析**](#3. 核糖体足迹分析)
- [🔄 生成BAM vs 使用最长CDS分析BAM](#🔄 生成BAM vs 使用最长CDS分析BAM)
- [🛠️ 如何生成最长CDS注释](#🛠️ 如何生成最长CDS注释)
- [💡 实际应用场景](#💡 实际应用场景)
- [🎯 为什么选择最长CDS而不是其他区域?](#🎯 为什么选择最长CDS而不是其他区域?)
- [❗ 重要提醒](#❗ 重要提醒)
- [📝 总结](#📝 总结)
- [2.4 那注释文件有什么用,有了参考基因序列为什么还要注释文件,只有用最长CDS才能获得基因表达矩阵吗](#2.4 那注释文件有什么用,有了参考基因序列为什么还要注释文件,只有用最长CDS才能获得基因表达矩阵吗)
- [🧩 注释文件的作用](#🧩 注释文件的作用)
-
- 具体作用:
-
- [1. **基因结构定义**](#1. 基因结构定义)
- [2. **指导比对过程**](#2. 指导比对过程)
- [3. **读数定量基础**](#3. 读数定量基础)
- [🎯 为什么两者都需要?](#🎯 为什么两者都需要?)
- [📊 获得基因表达矩阵的多种方法](#📊 获得基因表达矩阵的多种方法)
- [🎯 不同方法的适用场景](#🎯 不同方法的适用场景)
- [💡 实际工作流示例](#💡 实际工作流示例)
- [❓ 如何选择合适的方法?](#❓ 如何选择合适的方法?)
- [📝 总结](#📝 总结)
- [2.5 不同的物种有不同的参考基因组,注释文件吗,最长CDS是注释文件的一部分吗](#2.5 不同的物种有不同的参考基因组,注释文件吗,最长CDS是注释文件的一部分吗)
- [🧩 最长CDS与注释文件的关系](#🧩 最长CDS与注释文件的关系)
- [🛠️ 从原始注释生成最长CDS的实践](#🛠️ 从原始注释生成最长CDS的实践)
-
- 方法1:使用AGAT工具包
- [方法2:使用gffread + 自定义处理](#方法2:使用gffread + 自定义处理)
- 方法3:在R中处理
- [📁 实际项目中的文件组织](#📁 实际项目中的文件组织)
- [🔍 如何获取不同物种的参考数据](#🔍 如何获取不同物种的参考数据)
- [💡 重要注意事项](#💡 重要注意事项)
-
- [1. **版本一致性**](#1. 版本一致性)
- [2. **注释文件格式**](#2. 注释文件格式)
- [3. **什么时候需要自己生成最长CDS?**](#3. 什么时候需要自己生成最长CDS?)
- [📝 总结](#📝 总结)
- [2.6 总结以上所有提问,系统详细地总结回答给我](#2.6 总结以上所有提问,系统详细地总结回答给我)
-
- [🧬 核心概念解析](#🧬 核心概念解析)
- [1. **参考基因组 (Reference Genome)**](#1. 参考基因组 (Reference Genome))
- [2. **注释文件 (Annotation File)**](#2. 注释文件 (Annotation File))
- [3. **最长CDS (Longest CDS)**](#3. 最长CDS (Longest CDS))
- [🔄 从FASTQ到BAM:完整流程](#🔄 从FASTQ到BAM:完整流程)
- RNA-seq与Ribo-seq流程对比
- 详细命令示例
-
- [RNA-seq BAM生成:](#RNA-seq BAM生成:)
- [Ribo-seq BAM生成:](#Ribo-seq BAM生成:)
- [📊 BAM文件的本质与区别](#📊 BAM文件的本质与区别)
- BAM文件包含内容:
- [RNA-seq vs Ribo-seq BAM特征:](#RNA-seq vs Ribo-seq BAM特征:)
- [🎯 基因表达矩阵获取方法](#🎯 基因表达矩阵获取方法)
- 多种定量策略:
-
- [1. **基因水平定量(最常用)**](#1. 基因水平定量(最常用))
- [2. **转录本水平定量**](#2. 转录本水平定量)
- [3. **最长CDS定量(Ribo-seq专用)**](#3. 最长CDS定量(Ribo-seq专用))
- 方法选择指南:
- [🌍 多物种分析注意事项](#🌍 多物种分析注意事项)
- 物种特异性资源:
- 版本一致性原则:
- [🛠️ 最长CDS的生成与应用](#🛠️ 最长CDS的生成与应用)
- 生成流程:
- 生成命令示例:
- [💡 关键理解点总结](#💡 关键理解点总结)
- [1. **参考基因组 vs 注释文件**](#1. 参考基因组 vs 注释文件)
- [2. **BAM文件的本质**](#2. BAM文件的本质)
- [3. **最长CDS的正确理解**](#3. 最长CDS的正确理解)
- [4. **流程选择原则**](#4. 流程选择原则)
- [🚀 实践建议](#🚀 实践建议)
- [3. RNA-seq与Ribo-seq比对差异分析](#3. RNA-seq与Ribo-seq比对差异分析)
-
- [3.1 相同原始测序fastq及参考基因组,ribo和RNAseq的hisat2是否一样,是否只是生成的bam不一样,如果bam不一样,为什么不一样](#3.1 相同原始测序fastq及参考基因组,ribo和RNAseq的hisat2是否一样,是否只是生成的bam不一样,如果bam不一样,为什么不一样)
- [3.2 HISAT2到底是什么,生成的hisat2文件是什么,两者hisat2文件内容是否相同](#3.2 HISAT2到底是什么,生成的hisat2文件是什么,两者hisat2文件内容是否相同)
-
- [1. HISAT2 到底是什么?](#1. HISAT2 到底是什么?)
- [2. 生成的 "hisat2文件" 是什么?](#2. 生成的 “hisat2文件” 是什么?)
-
- [A. HISAT2 索引文件](#A. HISAT2 索引文件)
- [B. HISAT2 比对输出文件](#B. HISAT2 比对输出文件)
- [3. 两者 "hisat2文件" 内容是否相同?](#3. 两者 “hisat2文件” 内容是否相同?)
-
- [情况一:HISAT2 索引文件](#情况一:HISAT2 索引文件)
- [情况二:HISAT2 比对输出文件(SAM/BAM)](#情况二:HISAT2 比对输出文件(SAM/BAM))
- 总结
- [3.3 也就是RNAseq和riboseq的原始数据就不同,故而使用同样的hisat2工具,用同一参考基因组进行比对后得到的hisat2文件的内容也不同,且后续生成的bam也不同,是吗,如有不对,请指出并纠正](#3.3 也就是RNAseq和riboseq的原始数据就不同,故而使用同样的hisat2工具,用同一参考基因组进行比对后得到的hisat2文件的内容也不同,且后续生成的bam也不同,是吗,如有不对,请指出并纠正)
-
- [1. 关于"原始数据就不同"](#1. 关于“原始数据就不同”)
- [2. 关于"使用同样的hisat2工具,用同一参考基因组"](#2. 关于“使用同样的hisat2工具,用同一参考基因组”)
- [3. 关于"得到的hisat2文件的内容也不同"](#3. 关于“得到的hisat2文件的内容也不同”)
- [4. 关于"后续生成的bam也不同"](#4. 关于“后续生成的bam也不同”)
- 纠正后的完整、精确表述
- [3.4 "相同的参考基因组索引文件"参考基因组的索引文件是怎样生成的,什么情况下参考基因组的索引文件会相同](#3.4 ”相同的参考基因组索引文件“参考基因组的索引文件是怎样生成的,什么情况下参考基因组的索引文件会相同)
-
- 参考基因组索引文件是怎样生成的?
- 什么情况下参考基因组的索引文件会相同?
-
- [1. 完全相同的参考基因组FASTA文件](#1. 完全相同的参考基因组FASTA文件)
- [2. 完全相同的HISAT2软件版本](#2. 完全相同的HISAT2软件版本)
- [3. 完全相同的构建参数](#3. 完全相同的构建参数)
- 在实际分析中的意义
- [3.5 总结以上所有提问,系统详细地总结回答给我](#3.5 总结以上所有提问,系统详细地总结回答给我)
1. RNA_seq和Ribo_seq技术基础概念解析
1.1 Riboseq和RNAseq都是什么
在本文中,Ribo-seq 和 RNA-seq 是两种高通量测序技术,用于从不同层面研究基因的表达和调控:
RNA-seq(RNA测序)
- 全称:RNA Sequencing
- 作用 :用于分析细胞或组织中所有转录本(mRNA)的表达水平和序列信息。
- 原理:通过高通量测技术对RNA进行测序,可以定量检测哪些基因被转录、转录的量有多少。
Ribo-seq(核糖体分析)
- 全称:Ribosome Profiling
- 作用 :用于捕捉正在被翻译的mRNA片段 ,即核糖体保护的mRNA片段,从而反映蛋白质翻译的活跃程度。
- 原理:通过核酸酶消化未被核糖体保护的RNA,保留并测序那些被核糖体保护的片段,从而揭示翻译的位点和效率。
两者的区别与联系:
- RNA-seq 告诉你"哪些基因被转录";
- Ribo-seq 告诉你"哪些转录本正在被翻译成蛋白质";
- 联合分析 (如本文)可以揭示翻译效率(Ribo-seq/RNA-seq比值),判断基因在转录后是否受到调控。
1.2 什么是最长CDS,有什么用
好的,这是一个非常重要的生物信息学概念,尤其在处理转录组数据时。
1. 什么是CDS?
- CDS 的全称是 Coding Sequence ,即 编码序列。
- 它指的是基因的DNA或RNA序列中,从起始密码子(通常是AUG) 到终止密码子(UAA, UAG, UGA) 之间的那一段序列。
- 这段序列直接决定了蛋白质的氨基酸序列。
需要注意的是:CDS不等于整个mRNA。mRNA还包括5' 和3' 非翻译区,这些区域不编码蛋白质,但对翻译的调控、稳定性和定位至关重要。
2. 什么是最长CDS?
- 一个基因可以通过可变剪接 产生多个不同的转录本(mRNA异构体)。
- 这些不同的转录本有着不同长度的CDS,因此会编码不同长度(甚至不同功能)的蛋白质。
- 最长CDS策略 指的是:从一个基因的所有转录本中,选出那个拥有最长编码序列的转录本,作为该基因的代表,用于后续的分析。
3. 为什么要使用最长CDS?(有什么用)
在像本文这样的高通量测序数据分析中,使用最长CDS主要有以下几个目的和优点:
主要优点:
-
标准化和简化分析
- 如果一个基因有10个转录本,分析时是把它当作10个独立的实体,还是1个基因?使用最长CDS可以将一个基因的所有表达信号"归因"于一个代表转录本,极大地简化了后续的差异表达、翻译效率等分析。
-
避免低估基因的表达水平
- 在RNA-seq或Ribo-seq中,一条测序读段可能匹配到某个基因的多个转录本。如果随意分配,可能会分散计数。
- 选择最长CDS作为唯一代表,可以将匹配到该基因任何区域的读段(只要在CDS区域内)统一计数到这个代表转录本上,从而更准确地估计该基因的整体表达水平。
-
特别适合Ribo-seq分析
- Ribo-seq分析的是翻译过程,其读段严格定位在CDS区域内。
- 使用最长CDS可以确保捕获到该基因在翻译过程中可能使用的所有密码子信息 ,这对于研究密码子使用偏好性分析至关重要。如果选择一个短的CDS,可能会丢失掉该基因一部分的密码子使用信息。
-
保证数据的可比性
- 确保每个基因在最终的表达矩阵中只出现一次,使得不同基因之间的表达量(如RPKM)具有可比性。
潜在缺点:
- 可能不反映真实的生物学情况:细胞在特定条件下可能并不表达最长的那个转录本,而是表达一个较短的、功能不同的转录本。最长CDS策略可能会掩盖这种特异性调控。
简而言之,最长CDS是一个在全局分析中为了操作简便和结果稳健而广泛采用的"约定俗成"的标准策略。
1.3 什么是可变剪切,为什么要分析密码子使用偏好性
当然!这两个概念是理解您提供的这篇论文深层机制的关键。我们来逐一详细解释。
1. 什么是可变剪切?
一句话概括: 可变剪切是指一个基因的初始RNA转录本,通过不同的剪接方式,产生多个信使RNA变体,从而从一个基因得到多种不同蛋白质的过程。
详细解释:
-
基因的结构 :一个典型的真核生物基因由外显子 和内含子 交替组成。
- 外显子:是最终会出现在成熟mRNA中的序列,包含了蛋白质的编码信息。
- 内含子:是间隔序列,在RNA加工过程中会被移除。
-
剪切过程:当基因被转录成前体mRNA后,细胞内的"剪切机器"会精确地识别并切除内含子,然后将外显子连接起来,形成成熟的mRNA。
-
"可变"之处 :可变剪切 的核心在于,这个"剪切和连接"的过程不是只有一种固定模式 。细胞可以根据发育阶段、组织类型或环境刺激,选择性地将不同的外显子组合在一起。
一个生动的比喻:
把一个基因想象成一个电影拍摄的原始素材库。
- 外显子 = 一个个独立的场景。
- 内含子 = 废弃的、不同角度的镜头或不连贯的片段。
- 可变剪切 = 剪辑师 从原始素材中挑选不同的场景,以不同的顺序进行拼接。
- 最终结果 = 通过不同的剪辑方式,可以从同一份原始素材中剪出影院版、导演剪辑版、电视预告片等不同版本的电影,它们讲述的故事侧重点可能完全不同。
生物学意义:
可变剪切极大地增加了生物体的蛋白质多样性。人类基因数量只有约2万个,但通过可变剪切可以产生的蛋白质数量远超这个数字。它是细胞分化、应激响应和疾病发生等重要生命过程的关键调控机制。
在您论文中的体现:
论文中提到"选择最长CDS作为代表转录本",正是因为一个基因存在多个由可变剪切产生的转录本,为了分析方便,他们选择了编码序列最长的那个版本来代表该基因。
2. 为什么要分析密码子使用偏好性?
一句话概括: 分析密码子使用偏好性可以帮助我们理解基因翻译的效率和调控,并能揭示细胞在特定状态下的适应策略。
详细解释:
-
密码子与简并性:
- 遗传密码有64种,但氨基酸只有20种。因此,大多数氨基酸都由多个 不同的密码子编码。这被称为遗传密码的简并性。
- 例如,编码亮氨酸的密码子有6个:UUA, UUG, CUU, CUC, CUA, CUG。
-
密码子使用偏好:
- 尽管多个密码子编码同一种氨基酸,但生物体(从细菌到人类)并非均等地使用它们。它们会更频繁地使用其中某几个,而较少使用另外几个。这种非随机使用的现象就是密码子使用偏好。
-
为什么会产生偏好?(分析的原因所在)
-
a. 翻译效率
- 细胞内有丰度不同的tRNA。那些被高频使用的密码子,通常对应着细胞内含量更丰富的tRNA。
- 使用偏好密码子时,核糖体能找到匹配的tRNA更快,翻译速度就快,蛋白质合成效率高。
- 使用稀有密码子时,核糖体需要等待稀有的tRNA,容易造成翻译暂停甚至中止,效率低下。
-
b. 翻译准确性
- 偏好密码子通常与反密码子的配对更准确,有助于减少翻译错误,保证蛋白质的正确合成。
-
c. 调控蛋白质折叠
- 在mRNA上某些关键位置使用稀有密码子,可以故意让核糖体在此处"暂停"一下,这为新生肽链的正确折叠赢得了时间,从而影响最终蛋白质的三维结构和功能。
-
d. 反映进化适应与环境压力
- 密码子使用偏好与物种的GC含量密切相关。偏好使用以G或C结尾的密码子(GC3偏好)是基因组GC含量高的物种的常见特征。
- 当细胞处于应激状态时,可能会通过改变密码子使用模式来优先保证某些关键蛋白(如免疫蛋白)的高效合成。
-
总结:
分析密码子使用偏好就像是分析细胞在合成蛋白质时使用的"词汇偏好"。通过分析这种偏好,我们可以推断出细胞当前的"工作状态"(是高速合成还是精细调控),以及它如何适应内外环境的变化(如应激、菌群改变)。
2. RNA-seq与Ribo-seq流程差异解析
2.1 解释一下相同的FASTQ,如何获得RNAseq的bam和Riboseq的bam,流程是什么
虽然RNA-seq和Ribo-seq分析都可能从FASTQ文件开始,但它们要回答的科学问题不同,这导致从样品制备、建库到计算分析都存在显著差异。主要的区别在于:RNA-seq旨在分析转录本的组成和丰度,而Ribo-seq旨在捕获正在翻译的核糖体位置。
下面这张流程图直观地展示了两条流程的关键步骤与核心差异:
Ribo-seq 流程 RNA-seq 流程 下游分析 RNA-seq
转录本定量
差异表达分析 Ribo-seq
核糖体密度分析
翻译效率计算 读长裁剪
固定长度
eg. 28-30 nt 去除rRNA reads
Bowtie 核糖体位置比对
STAR
通常不许跨内含子 全转录组比对
STAR/HISAT2
允许跨内含子 去除rRNA reads
Bowtie 相同的FASTQ文件 原始数据质控
FastQC 适配器与质量修剪
fastp/Trimmomatic 流程分支 生成排序索引的BAM文件
🔍 理解流程的关键差异
为什么流程图中的步骤有所不同?这主要是由两种技术的本质目标决定的。
-
rRNA去除的必要性 :在生物体内,rRNA(核糖体RNA) 在总RNA中占了绝大多数。无论是RNA-seq还是Ribo-seq,实验本身的目标都是研究占比较少但功能重要的mRNA(信使RNA)。因此,在数据分析中,通常第一步就是用Bowtie等工具将rRNA的读数过滤掉,以减少无用数据对后续分析的干扰。
-
读长裁剪的原因 :Ribo-seq的建库过程会保留受到核糖体保护的片段,其长度分布集中在特定的尺寸(例如,用于监测翻译的核糖体足迹通常约28-30个核苷酸)。将这个长度统一,可以确保在后续分析中,所有读数都精确对应一个核糖体所覆盖的区域,从而更精确地定位翻译位点。RNA-seq则没有这个必要,因为它需要反映整个转录本的长度信息。
-
比对策略的根本不同:这是两者最核心的差异。
- RNA-seq 的读数可能来自转录本的任何部分,并且其比对工具(如STAR、HISAT2)被设计为能够识别跨内含子的剪接信号。例如,STAR是ENCODE计划的御用比对软件,能高效处理跨内含子的读数。
- Ribo-seq 的读数代表核糖体正在翻译的精确位置。为了准确统计每个位置的核糖体密度,通常要求读数是无剪接的连续比对。因此,在比对时常常会关闭剪接比对功能,或者使用专门为此优化的参数。
📝 核心操作命令举例
以下是一些在流程中可能用到的核心命令示例,帮助你具体理解:
-
质控与修剪
- 使用
fastqc进行原始数据质控。 - 使用
fastp进行适配器修剪和质量控制。
- 使用
-
去除rRNA读数
使用
bowtie将测序读数比对到rRNA参考序列上,并分离出未比对上的读数(即非rRNA读数)用于后续分析。例如:bashbowtie -n 0 -a --best --strata -S -p 4 --un *.rm_rRNA.fq rRNA_index -1 *.clean.1.fastq -2 *.clean.2.fastq *.aligned_rRNA.sam -
Ribo-seq读长裁剪
使用诸如
fastx_trimmer之类的工具将读数修剪至固定长度。bashfastx_trimmer -l 28 -i input.rm_rRNA.fq -o output.trimmed.fq -
序列比对
-
RNA-seq比对(以STAR为例) :使用STAR进行基因组比对,允许发现剪接位点。
bashSTAR --genomeDir /path/to/genome_index --readFilesIn *.rm_rRNA.fq --runThreadN 4 --outSAMtype BAM Unsorted --outFileNamePrefix rnaseq. -
Ribo-seq比对(以STAR为例) :通常会调整参数,例如禁用某些典型的RNA-seq功能,以优化对于短读数的比对。
bashSTAR --genomeDir /path/to/genome_index --readFilesIn *.trimmed.fq --runThreadN 4 --outSAMtype BAM Unsorted --alignEndsType EndToEnd --seedSearchStartLmax 15 --outFilterScoreMinOverLread 0 --outFilterMatchNminOverLread 0 --outFileNamePrefix riboseq.注意:
--alignEndsType EndToEnd参数要求读数末端完全匹配,更适合Ribo-seq的短读数。
-
-
生成BAM文件
使用
samtools对STAR输出的未排序BAM文件进行排序和索引,得到最终的BAM文件。bashsamtools sort -@ 4 -o sorted.bam Aligned.out.bam samtools index sorted.bam
💎 重要补充与提醒
- 质量控制贯穿始终:在每个主要步骤前后(如原始数据、修剪后、比对后)都建议进行质控,确保步骤有效且未引入偏差。
- 参数选择是关键 :以上命令仅为示例。实际分析中,参考基因组版本、注释文件、软件参数(特别是Ribo-seq的比对参数) 都需要根据具体的实验设计和物种进行调整。强烈建议查阅所选工具的官方文档。
- 从BAM到科学发现 :得到BAM文件只是上游分析的第一步。下游分析才是精髓:
- RNA-seq的BAM 文件会用于进行转录本定量(如使用featureCounts、HTSeq),进而做差异基因表达分析(如DESeq2、edgeR)。
- Ribo-seq的BAM 文件会用于计算核糖体密度 ,分析翻译效率(例如与匹配的RNA-seq数据联合使用),并寻找开放阅读框(ORF)。
2.2 这两者的BAM有什么区别,什么是参考基因组,什么是最长CDS,如何生成的bam
很好的问题!这些是理解RNA-seq和Ribo-seq分析的核心概念。让我逐一详细解释:
🧬 参考基因组 (Reference Genome)
参考基因组是一个物种的"标准"DNA序列模板,就像一本参考书或地图。
- 是什么:某个物种的代表性基因组DNA序列,包含了所有染色体(和线粒体等)的序列
- 如何获得:通常来自权威数据库(如人类的GRCh38、小鼠的GRCm39)
- 作用:作为"地图",让测序读数能够找到自己在基因组上的正确位置
- 包含内容 :
- 序列本身(ATCG碱基序列)
- 注释信息:基因位置、外显子/内含子边界、转录方向等
示例:就像你有一张城市地图(参考基因组),测序读数就像地址片段,你需要在地图上找到每个地址的确切位置。
📊 RNA-seq与Ribo-seq BAM的区别
虽然都是BAM文件,但它们包含的信息和特征截然不同:
| 特征 | RNA-seq BAM | Ribo-seq BAM |
|---|---|---|
| 读数分布 | 均匀覆盖整个转录本 | 集中在CDS区域,呈三碱基周期性 |
| 剪接情况 | 大量跨内含子的剪接读数 | 主要是外显子内部的连续读数 |
| 读长特征 | 长度多样(通常75-150bp) | 固定短长度(通常28-30bp) |
| 信息内容 | 转录本结构和丰度 | 核糖体位置和翻译活性 |
可视化差异:
- RNA-seq:像均匀照亮整个转录本的灯光
- Ribo-seq:像只在翻译区域闪烁的聚光灯,且每3个碱基一个亮点
🧬 最长CDS (Longest CDS)
CDS = 编码序列 (Coding Sequence),即蛋白质编码区域
最长CDS指的是:当一个基因有多个转录本亚型时,选择编码蛋白最长的那个CDS区域。
为什么需要最长CDS?
- 基因通常有多个转录本变体
- 不同变体的CDS长度可能不同
- 为了标准化分析,常选择最长的CDS作为该基因的代表
如何生成最长CDS注释?
bash
# 从GTF/GFF注释文件提取最长转录本
# 使用工具如:
# 1. gffread (来自 cufflinks)
gffread -E input.gff -T -o output.gtf
# 2. 自定义脚本过滤
# 或使用R/Bioconductor包如GenomicFeatures🛠️ BAM文件的生成过程
BAM文件的生成本质上是将测序读数映射到参考基因组的过程:
核心步骤:
-
准备参考索引(一次性工作)
bash# 使用STAR建立基因组索引 STAR --runMode genomeGenerate \ --genomeDir /path/to/genome_index \ --genomeFastaFiles genome.fa \ --sjdbGTFfile annotation.gtf \ --runThreadN 10 -
序列比对(核心步骤)
bash# 将FASTQ读数比对到基因组 STAR --genomeDir /path/to/genome_index \ --readFilesIn reads.fastq \ --outSAMtype BAM Unsorted \ --runThreadN 4 -
后处理
bash# 排序BAM文件 samtools sort -@ 4 -o sorted.bam Aligned.out.bam # 建立索引 samtools index sorted.bam
🔍 技术细节:BAM文件包含什么?
BAM是SAM的二进制格式,每行代表一个读数的比对信息:
- QNAME:读数名称
- FLAG:比对状态位标识
- RNAME:参考序列名称(染色体)
- POS:比对起始位置
- CIGAR:比对详情(匹配、缺失、插入等)
- SEQ:读数序列
- QUAL:测序质量值
🎯 实际分析中的工作流程
RNA-seq BAM生成:
bash
# 1. 质控
fastqc *.fastq
# 2. 修剪
trim_galore --quality 20 --length 25 input.fastq
# 3. 比对
STAR --genomeDir genome_index \
--readFilesIn input_trimmed.fq \
--outSAMtype BAM SortedByCoordinate
# 4. 得到: Aligned.sortedByCoord.out.bamRibo-seq BAM生成:
bash
# 1-2. 质控和修剪(同RNA-seq)
# 3. 去除rRNA污染
bowtie -x rRNA_index input_trimmed.fq --un no_rRNA.fq
# 4. 读长筛选(选择28-30nt的读数)
awk 'length(seq)>=28 && length(seq)<=30' no_rRNA.fq > filtered.fq
# 5. 比对(特殊参数)
STAR --genomeDir genome_index \
--readFilesIn filtered.fq \
--alignEndsType EndToEnd \
--outFilterScoreMinOverLread 0 \
--outSAMtype BAM SortedByCoordinate💡 关键理解点
- 参考基因组是静态的"地图",而BAM文件是动态的"定位结果"
- 最长CDS是分析约定,确保不同基因间的可比性
- BAM文件不仅包含位置信息,还包含比对质量、序列特征等丰富信息
- RNA-seq和Ribo-seq BAM的根本区别源于它们要回答的生物学问题不同
理解这些基础概念后,你就能更好地解读BAM文件中的信息,并选择正确的下游分析方法了。
2.3 最长CDS有什么用,也可以用来生成BAM吗
很好的问题!你触及了生物信息学分析中一个重要的概念。让我详细解释最长CDS的作用和它与BAM文件的关系。
🎯 最长CDS的用途
最长CDS本身不能用来生成BAM文件 ,但它是在生成BAM文件之后进行下游分析的关键工具。
主要用途:
1. 读数定量 (Read Counting)
bash
# 使用featureCounts基于最长CDS注释对BAM文件进行定量
featureCounts -T 8 -t CDS -g gene_id -a longest_cds.gtf -o counts.txt *.bam
# 或使用HTSeq
htseq-count -t CDS -i gene_id -f bam aligned.bam longest_cds.gtf > counts.txt2. 翻译效率分析 (Ribo-seq特有)
r
# 在R中计算翻译效率
rna_counts <- read.table("rna_counts.txt")
ribo_counts <- read.table("ribo_counts.txt")
# 使用最长CDS区域内的读数计算TE
translation_efficiency <- ribo_counts / (rna_counts + 1)3. 核糖体足迹分析
- 分析核糖体在CDS区域的三碱基周期性
- 识别翻译起始位点
- 检测移码或翻译暂停位点
🔄 生成BAM vs 使用最长CDS分析BAM
这是一个重要的概念区分:
FASTQ文件 比对到参考基因组 生成BAM文件 基因注释文件 提取最长CDS 最长CDS注释 BAM文件分析 获得基因表达矩阵
关键区别:
- 生成BAM:需要完整的参考基因组序列(FASTA)
- 分析BAM:需要基因注释信息(GTF),其中可能包含最长CDS定义
🛠️ 如何生成最长CDS注释
方法1:使用gffread
bash
# 从原始GTF提取最长转录本
gffread -E input.gtf --keep-exon-attrs -T -o temp.gtf
# 然后使用脚本过滤得到每个基因最长的CDS
# 或者使用专门的工具方法2:使用R/Bioconductor
r
library(GenomicFeatures)
library(dplyr)
# 创建转录本数据库
txdb <- makeTxDbFromGFF("annotation.gtf")
# 获取所有CDS
cds <- cdsBy(txdb, by="tx")
# 计算每个转录本CDS长度
cds_lengths <- sum(width(cds))
# 找到每个基因的最长CDS转录本
longest_tx <- cds_lengths %>%
as.data.frame() %>%
group_by(gene_id) %>%
slice_max(order_by = length) %>%
pull(tx_name)
# 导出最长CDS的GTF
export(longest_tx, "longest_cds.gtf")方法3:使用专门的工具
bash
# 使用AGAT工具包
agat_sp_keep_longest_isoform.pl -gff input.gff -o longest_isoforms.gff
# 或使用CPAT等工具💡 实际应用场景
场景1:RNA-seq差异表达分析
bash
# 1. 生成BAM文件(使用完整基因组)
STAR --genomeDir genome_index --readFilesIn RNAseq.fastq
# 2. 使用最长CDS进行定量
featureCounts -a longest_cds.gtf -o rna_counts.txt RNAseq.bam
# 3. 在R中进行差异分析
# → 得到每个基因的表达水平场景2:Ribo-seq翻译效率分析
bash
# 1. 分别生成RNA-seq和Ribo-seq的BAM
STAR --genomeDir genome_index --readFilesIn RNAseq.fastq
STAR --genomeDir genome_index --readFilesIn Riboseq.fastq
# 2. 使用相同的最长CDS注释分别定量
featureCounts -a longest_cds.gtf -o rna_counts.txt RNAseq.bam
featureCounts -a longest_cds.gtf -o ribo_counts.txt Riboseq.bam
# 3. 计算翻译效率
# → 识别高效翻译的基因🎯 为什么选择最长CDS而不是其他区域?
优势:
- 标准化比较:确保每个基因使用相同的区域进行比较
- 生物学意义:CDS区域直接对应蛋白质产物
- 技术考量:避免UTR区域在Ribo-seq中的复杂信号
替代方案:
- 全部转录本:考虑所有异构体,但分析复杂
- 全部外显子:包含UTR,但UTR在翻译调控中信号复杂
❗ 重要提醒
- 时序关系:先有BAM,后用最长CDS分析BAM
- 一致性 :RNA-seq和Ribo-seq要使用相同的最长CDS定义
- 质量控制:确保CDS注释质量,错误的注释会导致错误的结果
- 物种特异性:不同物种可能需要不同的CDS定义策略
📝 总结
- 最长CDS不是用来生成BAM的 ,而是用来解释BAM文件的
- 它是一个分析约定,确保基因间定量结果的可比性
- 在Ribo-seq分析中尤其重要,因为核糖体主要分布在CDS区域
- 生成最长CDS需要从原始注释文件出发,通过生物信息学方法提取
理解这个区别很重要:参考基因组帮你定位读数 ,而最长CDS帮你解释这些读数的生物学意义。
2.4 那注释文件有什么用,有了参考基因序列为什么还要注释文件,只有用最长CDS才能获得基因表达矩阵吗
非常好的问题!这触及了基因组注释的核心概念。让我详细解释注释文件的作用以及基因表达矩阵的多种获取方式。
🧩 注释文件的作用
注释文件回答了"参考基因组上有什么"的问题,就像给一张空白地图添加地标和说明:
| 参考基因组 (FASTA) | 注释文件 (GTF/GFF) |
|---|---|
| 只有ATCG序列 | 标记基因位置、结构、功能 |
| 像一本没有标点的小说 | 像加了章节标题和段落标记的小说 |
| 知道序列但不知道功能 | 知道哪里是基因、外显子、启动子等 |
具体作用:
1. 基因结构定义
gtf
# GTF文件示例行
chr1 ensembl gene 1000 5000 . + . gene_id "GENE1"
chr1 ensembl transcript 1000 5000 . + . transcript_id "T1"
chr1 ensembl exon 1000 2000 . + . exon_id "E1"
chr1 ensembl CDS 1500 2000 . + 0 gene_id "GENE1"2. 指导比对过程
bash
# STAR使用注释文件改进剪接位点检测
STAR --genomeDir index \
--sjdbGTFfile annotation.gtf \ # 这里使用注释!
--readFilesIn reads.fastq3. 读数定量基础
bash
# featureCounts需要注释文件知道哪里计数
featureCounts -a annotation.gtf \ # 这里使用注释!
-t exon \ # 计数什么特征
-g gene_id \ # 按什么分组
-o counts.txt *.bam🎯 为什么两者都需要?
类比:参考基因组就像一张空白世界地图,注释文件就像在地图上标记国家、城市、河流的标签。
bash
# 没有注释文件 → 知道序列但不知道基因
ATCGATCGATCGATCG... (数百万行)
# 有注释文件 → 知道功能元素
chr1:1000-5000 → 基因GENE1, 蛋白质编码
chr1:1500-2000 → 外显子1, CDS区域📊 获得基因表达矩阵的多种方法
不,最长CDS不是唯一方法! 实际上有多个层次的定量策略:
方法1:基于基因水平 (最常用)
bash
# 计数所有外显子区域(包括UTR)
featureCounts -a annotation.gtf -t exon -g gene_id -o gene_counts.txt *.bam
# 或者使用HTSeq
htseq-count -f bam -t exon -i gene_id aligned.bam annotation.gtf方法2:基于转录本水平
bash
# 使用Salmon或Kallisto进行转录本定量
salmon quant -i transcriptome_index -l A -1 read1.fq -2 read2.fq -o quants
# 然后聚合到基因水平
tximport --type salmon --countsFromAbundance lengthScaledTPM salmon_quants/ gene_counts.txt方法3:基于外显子水平
bash
# 计数每个外显子
featureCounts -a annotation.gtf -t exon -g exon_id -o exon_counts.txt *.bam方法4:基于最长CDS (Ribo-seq专用)
bash
# 只计数CDS区域,且每个基因选最长转录本
featureCounts -a longest_cds.gtf -t CDS -g gene_id -o cds_counts.txt *.bam🎯 不同方法的适用场景
| 方法 | 适用场景 | 优点 | 缺点 |
|---|---|---|---|
| 基因水平 | 大部分RNA-seq分析 | 简单、稳定 | 忽略异构体差异 |
| 转录本水平 | 异构体差异研究 | 分辨不同转录本 | 计算复杂、准确性依赖注释质量 |
| 外显子水平 | 外显子使用差异分析 | 检测可变剪接 | 数据维度高 |
| 最长CDS | Ribo-seq翻译效率 | 标准化、避免UTR影响 | 丢失异构体信息 |
💡 实际工作流示例
RNA-seq标准分析(最常用):
bash
# 1. 比对
STAR --genomeDir genome_index --sjdbGTFfile annotation.gtf --readFilesIn RNAseq.fq
# 2. 基因水平定量(使用所有外显子)
featureCounts -a annotation.gtf -t exon -g gene_id -o counts.txt Aligned.sortedByCoord.out.bam
# 3. 得到表达矩阵
# gene1 count1
# gene2 count2
# ... ...Ribo-seq翻译效率分析:
bash
# 1. 分别比对RNA-seq和Ribo-seq
STAR ... --readFilesIn RNAseq.fq # → RNAseq.bam
STAR ... --readFilesIn Riboseq.fq # → Riboseq.bam
# 2. 使用相同的最长CDS注释分别定量
featureCounts -a longest_cds.gtf -t CDS -g gene_id -o rna_counts.txt RNAseq.bam
featureCounts -a longest_cds.gtf -t CDS -g gene_id -o ribo_counts.txt Riboseq.bam
# 3. 计算翻译效率
TE = ribo_counts / (rna_counts + 1)❓ 如何选择合适的方法?
考虑因素:
-
科学问题:
- 差异基因表达 → 基因水平
- 异构体差异 → 转录本水平
- 翻译效率 → 最长CDS
-
数据类型:
- RNA-seq → 基因/转录本水平
- Ribo-seq → 最长CDS
-
注释质量:
- 注释完整 → 可用转录本水平
- 注释一般 → 用基因水平更稳定
📝 总结
- 注释文件是理解基因组功能的"说明书",没有它,我们不知道序列的生物学意义
- 参考基因组+注释文件才构成完整的分析基础
- 最长CDS只是多种定量策略之一,不是唯一方法
- 基因水平定量(基于所有外显子) 是最常用、最稳定的方法
- 选择方法要基于科学问题、数据类型和注释质量
对于大部分RNA-seq分析,从基因水平开始是最安全的选择。只有在特定问题(如翻译效率分析)时,才需要考虑最长CDS策略。
2.5 不同的物种有不同的参考基因组,注释文件吗,最长CDS是注释文件的一部分吗
非常准确的问题!这些都是生物信息学分析中的关键概念。让我逐一详细解释:
🌍 不同物种的参考基因组和注释文件
是的,不同物种有完全不同的参考基因组和注释文件,就像不同国家有不同的地图和地名词典。
实际情况:
| 物种 | 参考基因组示例 | 注释文件来源 | 特点 |
|---|---|---|---|
| 人类 | GRCh38 (hg38), GRCh37 (hg19) | Ensembl, GENCODE, RefSeq | 最完善,多个版本 |
| 小鼠 | GRCm39 (mm39), GRCm38 (mm10) | Ensembl, MGI | 哺乳动物模式生物 |
| 果蝇 | BDGP6 (dm6) | FlyBase | 经典遗传学模型 |
| 拟南芥 | TAIR10 | TAIR | 植物模式生物 |
| 水稻 | IRGSP-1.0 | RAP-DB, MSU-RGAP | 重要农作物 |
版本管理很重要:
bash
# 错误:版本不匹配!
# 使用hg19基因组 + hg38注释 → 完全错误!
# 正确:版本一致
# 使用GRCh38基因组 + Ensembl 109注释 (基于GRCh38)🧩 最长CDS与注释文件的关系
最长CDS是注释文件的派生品,不是原始注释的一部分。
关系图解:
原始注释文件 (annotation.gtf)
↓ (包含所有转录本信息)
所有基因的所有转录本
↓ (生物信息学处理)
提取每个基因的最长CDS转录本
↓
最长CDS注释文件 (longest_cds.gtf)具体来说:
- 原始注释:包含基因A的3个转录本(T1, T2, T3),各有不同CDS长度
- 最长CDS:只保留基因A的T1(假设T1的CDS最长)
🛠️ 从原始注释生成最长CDS的实践
方法1:使用AGAT工具包
bash
# 安装AGAT
conda install -c bioconda agat
# 提取最长异构体
agat_sp_keep_longest_isoform.pl -gff input.gff -o longest_isoforms.gff
# 然后从最长异构体中提取CDS
agat_sp_extract_sequences.pl -gff longest_isoforms.gff -f genome.fa -t cds -o longest_cds.fasta方法2:使用gffread + 自定义处理
bash
# 提取所有CDS
gffread -E input.gff -g genome.fa -x all_cds.fasta
# 然后需要自定义脚本选择每个基因最长的CDS
python filter_longest_cds.py all_cds.fasta > longest_cds.fasta方法3:在R中处理
r
library(GenomicFeatures)
library(Biostrings)
# 创建转录本数据库
txdb <- makeTxDbFromGFF("input.gff")
# 获取CDS按基因分组
cds_by_gene <- cdsBy(txdb, by = "gene")
# 计算每个基因的CDS总长度
cds_lengths <- sum(width(cds_by_gene))
# 找到每个基因最长的CDS
longest_cds <- cds_by_gene[cds_lengths == max(cds_lengths), ]
# 导出为GTF
export(longest_cds, "longest_cds.gtf")📁 实际项目中的文件组织
典型分析项目的文件结构:
project/
├── reference/
│ ├── genome/
│ │ ├── GRCh38.fa # 参考基因组序列
│ │ └── GRCh38.fa.fai # 基因组索引
│ ├── annotations/
│ │ ├── original/
│ │ │ └── Homo_sapiens.GRCh38.109.gtf # 原始注释
│ │ └── processed/
│ │ ├── longest_cds.gtf # 生成的最长CDS
│ │ └── gene_level.gtf # 基因水平注释
│ └── indexes/
│ ├── star_index/ # STAR索引
│ └── bowtie2_index/ # Bowtie2索引
├── data/
│ ├── RNAseq/
│ │ └── sample1.fastq
│ └── Riboseq/
│ └── sample1.fastq
└── results/
├── bam_files/
└── count_tables/🔍 如何获取不同物种的参考数据
常用数据库:
bash
# 1. Ensembl (最常用)
# 人类: ftp.ensembl.org/pub/release-109/fasta/homo_sapiens/dna/
# 小鼠: ftp.ensembl.org/pub/release-109/fasta/mus_musculus/dna/
# 2. NCBI RefSeq
# https://www.ncbi.nlm.nih.gov/assembly/
# 3. UCSC Genome Browser
# https://hgdownload.soe.ucsc.edu/downloads.html
# 4. 物种特定数据库
# 植物: Phytozome, Gramene
# 微生物: NCBI, EBI下载示例:
bash
# 下载人类GRCh38基因组
wget ftp.ensembl.org/pub/release-109/fasta/homo_sapiens/dna/Homo_sapiens.GRCh38.dna.primary_assembly.fa.gz
# 下载对应注释
wget ftp.ensembl.org/pub/release-109/gtf/homo_sapiens/Homo_sapiens.GRCh38.109.gtf.gz
# 解压
gunzip *.gz💡 重要注意事项
1. 版本一致性
bash
# 错误示例
genome: GRCh38 (Ensembl release 105)
annotation: GRCh37 (Ensembl release 87) # 不匹配!
# 正确示例
genome: GRCh38 (Ensembl release 109)
annotation: Homo_sapiens.GRCh38.109.gtf # 匹配!2. 注释文件格式
bash
# GTF (Gene Transfer Format) - 更常见
chr1 ensembl gene 11869 14409 . + . gene_id "ENSG00000223972"; gene_name "DDX11L1";
# GFF (General Feature Format)
chr1 ensembl gene 11869 14409 . + . ID=gene:ENSG00000223972;Name=DDX11L1
# 两者可互相转换3. 什么时候需要自己生成最长CDS?
- Ribo-seq翻译效率分析:必须使用
- 跨物种比较:需要统一标准
- 注释文件质量差:需要手动整理
- 特定研究问题:如只关注主要蛋白产物
📝 总结
- 不同物种有不同的参考基因组和注释文件,就像不同语言有不同的字典
- 最长CDS不是原始注释的一部分,而是通过生物信息学处理从原始注释中派生出来的
- 生成最长CDS是一个预处理步骤,用于特定分析目的
- 版本一致性至关重要 - 基因组、注释文件、分析工具都要匹配
对于大部分标准RNA-seq分析,你不需要自己生成最长CDS,直接使用原始注释文件进行基因水平定量即可。只有在进行Ribo-seq翻译效率分析或特定比较研究时,才需要这个额外的处理步骤。
2.6 总结以上所有提问,系统详细地总结回答给我
🧬 核心概念解析
1. 参考基因组 (Reference Genome)
- 是什么:物种的标准DNA序列模板,包含所有染色体的ATCG序列
- 作用:作为"地图"让测序读数找到自己的位置
- 来源:权威数据库(Ensembl、NCBI、UCSC)
- 示例:人类GRCh38、小鼠GRCm39、果蝇BDGP6
2. 注释文件 (Annotation File)
-
是什么:描述参考基因组上功能元素的"说明书"
-
格式:GTF/GFF格式,包含基因位置、外显子、CDS等信息
-
作用:将序列坐标转化为生物学意义
-
内容 :
gtfchr1 ensembl gene 1000 5000 . + . gene_id "GENE1" chr1 ensembl CDS 1500 2000 . + 0 gene_id "GENE1"
3. 最长CDS (Longest CDS)
- 是什么:从多转录本基因中选择编码序列最长的转录本
- 地位:原始注释的派生品,非原始注释的一部分
- 生成方法:通过生物信息学工具从原始注释提取
- 用途:主要用于Ribo-seq翻译效率分析
🔄 从FASTQ到BAM:完整流程
RNA-seq与Ribo-seq流程对比
| 步骤 | RNA-seq | Ribo-seq |
|---|---|---|
| 原始质控 | FastQC | FastQC |
| 质量修剪 | Trimmomatic/fastp | Trimmomatic/fastp |
| 去污染 | 去除rRNA读数 | 去除rRNA读数 |
| 读长处理 | 保持原始长度 | 裁剪至固定长度(28-30nt) |
| 序列比对 | STAR/HISAT2(允许剪接) | STAR(禁止剪接,特殊参数) |
| 输出格式 | BAM文件 | BAM文件 |
详细命令示例
RNA-seq BAM生成:
bash
# 比对
STAR --genomeDir genome_index \
--sjdbGTFfile annotation.gtf \
--readFilesIn RNAseq.fastq \
--outSAMtype BAM SortedByCoordinate
# 排序索引
samtools sort -@ 4 -o RNAseq.sorted.bam Aligned.out.bam
samtools index RNAseq.sorted.bamRibo-seq BAM生成:
bash
# 读长筛选与比对
STAR --genomeDir genome_index \
--readFilesIn Riboseq_trimmed.fq \
--alignEndsType EndToEnd \
--outFilterScoreMinOverLread 0 \
--outSAMtype BAM SortedByCoordinate📊 BAM文件的本质与区别
BAM文件包含内容:
- 序列比对位置(染色体、起始位置)
- 比对质量信息(CIGAR字符串、MAPQ)
- 原始序列和质量值
- 比对标记(是否配对、是否跨内含子等)
RNA-seq vs Ribo-seq BAM特征:
| 特征 | RNA-seq BAM | Ribo-seq BAM |
|---|---|---|
| 读数分布 | 均匀覆盖转录本 | 集中在CDS区域 |
| 周期模式 | 无周期性 | 三碱基周期性 |
| 剪接情况 | 大量剪接读数 | 主要为连续比对 |
| 读长分布 | 多样(75-150bp) | 固定短长度(28-30bp) |
| 生物学意义 | 转录本丰度 | 核糖体位置 |
🎯 基因表达矩阵获取方法
多种定量策略:
1. 基因水平定量(最常用)
bash
featureCounts -a annotation.gtf -t exon -g gene_id -o counts.txt *.bam2. 转录本水平定量
bash
salmon quant -i transcriptome_index -l A -1 read1.fq -2 read2.fq -o quants3. 最长CDS定量(Ribo-seq专用)
bash
featureCounts -a longest_cds.gtf -t CDS -g gene_id -o ribo_counts.txt Riboseq.bam方法选择指南:
| 方法 | 适用场景 | 优点 | 缺点 |
|---|---|---|---|
| 基因水平 | 大部分RNA-seq | 稳定可靠 | 忽略异构体 |
| 转录本水平 | 异构体差异研究 | 分辨率高 | 计算复杂 |
| 最长CDS | 翻译效率分析 | 标准化好 | 丢失信息 |
🌍 多物种分析注意事项
物种特异性资源:
| 物种 | 基因组版本 | 注释来源 |
|---|---|---|
| 人类 | GRCh38/hg38 | Ensembl, GENCODE |
| 小鼠 | GRCm39/mm39 | Ensembl, MGI |
| 果蝇 | BDGP6/dm6 | FlyBase |
| 植物 | 物种特定 | Phytozome, TAIR |
版本一致性原则:
bash
# 错误:版本不匹配
基因组: GRCh38
注释文件: GRCh37版本 # 染色体坐标不匹配!
# 正确:版本完全一致
基因组: GRCh38 (Ensembl release 109)
注释文件: Homo_sapiens.GRCh38.109.gtf🛠️ 最长CDS的生成与应用
生成流程:
原始注释文件 (annotation.gtf)
↓ 提取所有转录本
多转录本基因集合
↓ 选择每个基因最长CDS
最长CDS注释 (longest_cds.gtf)
↓ 用于定量
基因表达矩阵生成命令示例:
bash
# 使用AGAT工具包
agat_sp_keep_longest_isoform.pl -gff input.gff -o longest_isoforms.gff
# 或使用R语言
library(GenomicFeatures)
txdb <- makeTxDbFromGFF("input.gtf")
longest_cds <- ... # 生物信息学处理💡 关键理解点总结
1. 参考基因组 vs 注释文件
- 参考基因组 = 空白地图(只有序列)
- 注释文件 = 地图上的标记(基因、外显子等功能元素)
- 两者必须版本匹配
2. BAM文件的本质
- BAM是比对结果的二进制格式
- 包含序列位置、质量、比对详情等信息
- RNA-seq和Ribo-seq BAM反映不同的生物学过程
3. 最长CDS的正确理解
- 是分析工具,非原始数据
- 主要用于标准化比较
- 在翻译效率分析中不可或缺
4. 流程选择原则
- 科学问题驱动:不同问题需要不同流程
- 数据特性决定:RNA-seq和Ribo-seq需要不同处理
- 质量控制贯穿:每个步骤都需要质控
🚀 实践建议
- 始终从权威数据库下载匹配的基因组和注释文件
- RNA-seq分析从基因水平定量开始
- Ribo-seq分析必须使用最长CDS策略
- 保持整个分析流程的版本一致性
- 在每个关键步骤进行质量控制
这个系统总结涵盖了从原始FASTQ到最终基因表达矩阵的完整知识体系,应该能够帮助你全面理解整个分析流程和各个组件之间的关系。
3. RNA-seq与Ribo-seq比对差异分析
3.1 相同原始测序fastq及参考基因组,ribo和RNAseq的hisat2是否一样,是否只是生成的bam不一样,如果bam不一样,为什么不一样
这是一个非常好的问题,它触及了RNA-seq和Ribo-seq(核糖体图谱分析)在数据分析核心环节的关键区别。
简单直接的回答是:不,不完全一样。虽然都使用HISAT2,但关键的比对参数和预处理步骤通常需要根据数据特性进行调整,而这直接导致了最终BAM文件内容的根本性不同。
下面我们来详细解释为什么。
核心区别:实验目的与数据特性
首先要理解RNA-seq和Ribo-seq的生物学目的:
- RNA-seq: 目的是捕获细胞中所有转录本(mRNA)的存在和丰度。它的读段是随机地从整个转录本长度上产生的。
- Ribo-seq: 目的是捕获正在被翻译的mRNA片段,即被核糖体保护的足迹。这些足迹非常短(约28-30核苷酸),并且精确地定位在翻译的起始密码子和终止密码子之间,具有明显的周期性(三核苷酸周期)。
这些不同的目的导致了数据特性的巨大差异,进而影响了比对策略。
详细对比:从输入到输出的差异
| 环节 | RNA-seq | Ribo-seq | 解释与原因 |
|---|---|---|---|
| 1. 输入FASTQ | 相同的原始测序数据(理论上) | 通常不同 | 虽然源头是同一批细胞,但建库方式不同。Ribo-seq的读长被严格限定在核糖体足迹长度,而RNA-seq读长分布更广。在分析时,通常会对Ribo-seq数据进行读长筛选,只保留特定长度(如28-30nt)的读段进行比对,以增强信噪比。 |
| 2. 参考基因组 | 相同 | 相同 | 两者都使用相同的参考基因组和注释文件,因为它们的读段都源于生物的基因组。 |
| 3. HISAT2 参数 | 通常使用默认或标准RNA-seq参数 例如:--rna-strandness |
需要特殊参数 关键:--no-spliced-alignment 或 --pen-noncansplice |
这是最核心的区别。 • RNA-seq :读段可能跨越外显子-外显子连接处,因此HISAT2的剪接比对 功能至关重要。 • Ribo-seq :核糖体保护的足迹非常短,且来自单个外显子(因为一个核糖体占据的位置远小于一个内含子)。如果允许剪接比对,大量短读段可能会被错误地 比对到跨越内含子的基因组区域,产生假阳性。因此,通常需要关闭或严格限制剪接比对 ,强制进行非剪接比对。 |
| 4. 预处理与比对策略 | 直接比对 | 偏移比对 | Ribo-seq分析中的一个关键步骤是确定核糖体保护足迹的精确位置。由于核糖体本身有大小,测序读段的5'端起始位置并不直接对应核糖体P位点(肽酰位点)的中心。因此,在比对后或比对前,需要对读段的起始位置进行一个固定长度的偏移(例如,向后偏移12nt或15nt),以精确定位密码子。这个偏移后的位置信息才会被用于后续分析。这在RNA-seq中是完全不需要的。 |
| 5. 输出BAM文件 | 非常不同 | 非常不同 | 由于上述所有区别,生成的BAM文件在内容上存在根本差异: • 比对位置分布 :RNA-seq读段均匀分布在转录本上;Ribo-seq读段密集堆积在蛋白质编码区 ,并且在起始和终止密码子处有显著富集。 • 剪接信息 :RNA-seq的BAM包含大量跨越内含子的比对(N在CIGAR字符串中);Ribo-seq的BAM几乎全是连续的比对(M在CIGAR字符串中)。 • 周期信号 :Ribo-seq的BAM文件,在正确的读长和偏移下,会显示出三核苷酸的周期性,即读段在三个阅读框中的一个有显著富集,这是其数据质量的重要指标。RNA-seq没有这种模式。 |
总结与比喻
您可以将这个过程想象成使用同一张城市地图(参考基因组)寻找两种不同的目标:
-
RNA-seq 像是在问:"这座城市里有哪些建筑(基因),它们大概有多大(表达量)?"
- 你派出的调查员(读段)可以乘坐直升机,从空中拍摄整个建筑群的全景,即使有墙壁(内含子)阻挡,他们也能看到建筑的整体结构。HISAT2就像是一个能理解"这个建筑由A、B、C三栋楼通过走廊连接而成"的智能地图分析师。
-
Ribo-seq 像是在问:"这座城市里,哪些建筑的窗户(密码子)后面此刻正有人(核糖体)在活动?"
- 你派出的调查员是地面特工,他们非常矮小(短读段),只能紧贴着每一扇窗户进行记录。他们无法从空中跨越整条街道(内含子)去观察另一栋楼。HISAT2在这里需要被设定为只寻找与单扇窗户精确匹配的位置,而不是去猜测它是否属于一个跨越街道的复杂结构。
结论:
- HISAT2工具相同,但参数策略不同 :对于Ribo-seq,通常需要禁用或严惩剪接比对(
--no-spliced-alignment)。 - BAM文件内容根本不同 :这不仅仅是"不一样",而是由于实验本质和数据分析目标不同导致的预期内的、有意义的差异。Ribo-seq的BAM文件包含了翻译的动态信息,而RNA-seq的BAM文件包含了转录本的静态结构和丰度信息。
因此,在分析Ribo-seq数据时,绝对不能简单套用RNA-seq的标准流程,必须根据其生物学和技术特点进行相应的调整。
3.2 HISAT2到底是什么,生成的hisat2文件是什么,两者hisat2文件内容是否相同
好的,我们来彻底厘清这几个概念。这是一个非常关键的区分,理解了它们,你就明白了比对的核心流程。
1. HISAT2 到底是什么?
HISAT2 是一个软件,一个工具包。 它主要包含两个核心功能:
- 构建索引 :为一个参考基因组(如 human GRCh38)建立一个高效的、可供快速搜索的数据结构。这个功能通过
hisat2-build命令实现。 - 进行比对 :将测序得到的短读段文件映射到预先构建好的基因组索引上,找到每个读段最可能来源的基因组位置。这个功能通过
hisat2命令实现。
简单比喻:
- 参考基因组 是一本厚厚的书(例如《莎士比亚全集》)。
hisat2-build就像是给这本书制作一个超详细的、数字化的索引目录 (比如"to be, or not to be" 出现在第X页第Y行)。这个目录就是 HISAT2索引文件。hisat2(比对命令) 就像是有一个超级搜索引擎 。你给它一堆碎纸片(FASTQ文件,即测序读段),它利用之前建好的索引目录,快速找出每一张碎纸片上的文字在这本厚书中的出处。- 最终生成的BAM/SAM文件 ,就是这个搜索引擎输出的比对结果报告,里面清晰地记录了"碎纸片A来自第X页第Y行"。
2. 生成的 "hisat2文件" 是什么?
这里需要精确一下术语。运行HISAT2流程,通常会生成两种性质完全不同的文件:
A. HISAT2 索引文件
-
这是什么? 这是通过
hisat2-build命令预先创建的、用于加速比对的数据库文件。它不是由你的测序数据生成的,而是由参考基因组FASTA文件生成的。 -
文件内容: 是一系列经过高度压缩和重排的基因组序列数据,通常以
.ht2或.ht2l作为后缀(例如genome.1.ht2,genome.2.ht2等)。你不能直接打开或阅读这些文件的内容,它们是给HISAT2软件内部使用的。 -
创建命令示例:
bashhisat2-build -p 4 reference_genome.fa genome # 这会生成一堆名为 genome.1.ht2, genome.2.ht2... 的文件
B. HISAT2 比对输出文件
- 这是什么? 这是运行
hisat2比对命令后产生的结果文件,它记录了你的测序读段 与参考基因组的匹配情况。 - 默认格式:SAM文件 。但通常会立即使用
samtools将其转换为更紧凑的 BAM文件。 - 文件内容: 这是一个结构化的文本(SAM)或二进制(BAM)文件,每一行代表一个读段的比对信息,包含至少11个必填字段。
- 关键字段包括:
QNAME: 读段的ID。FLAG: 比对的位标志(是否是pair-end,是否比对到正链/负链等)。RNAME: 比对到的参考基因组染色体/contig名称。POS: 比对上的最左端起始位置。CIGAR: 简要比对信息表达式 ,这是理解比对细节的关键 !它用字母和数字描述读段是如何匹配、插入、缺失或剪接的。M: 匹配或错配I: 相对于参考基因组的插入D: 相对于参考基因组的缺失N: 内含子跳过(这是RNA-seq特有的重要标志)
SEQ: 读段的原始序列。
- 关键字段包括:
3. 两者 "hisat2文件" 内容是否相同?
现在我们来回答你的核心问题,这需要分情况讨论:
情况一:HISAT2 索引文件
- 结论:完全相同。
- 原因: 无论是处理RNA-seq还是Ribo-seq数据,只要它们来自同一个物种 ,你就会使用同一个参考基因组 (例如,都是人类GRCh38)。因此,你为这个基因组构建的HISAT2索引文件(那一堆
.ht2文件)是完全一样的。你只需要构建一次,就可以用于该物种的所有比对任务。
情况二:HISAT2 比对输出文件(SAM/BAM)
- 结论:完全不同。 这正是你上一个问题的核心。
- 原因(结合文件内容解释):
-
CIGAR字符串的根本差异:
- RNA-seq BAM :由于读段可能跨越外显子-内含子边界,其CIGAR字符串中会大量出现
N(表示内含子)。例如:100M150N50M表示一个100bp的匹配,然后跳过了150bp的内含子,再匹配50bp。 - Ribo-seq BAM :由于使用了
--no-spliced-alignment参数,并且读段很短,它们几乎总是比对到一个连续的区域。其CIGAR字符串几乎全是30M(假设读长为30bp)这样的连续匹配,几乎没有N。
- RNA-seq BAM :由于读段可能跨越外显子-内含子边界,其CIGAR字符串中会大量出现
-
比对位置分布的差异:
- RNA-seq BAM:读段均匀分布在基因的整个转录本上,包括5'UTR, CDS, 3'UTR。
- Ribo-seq BAM :读段高度集中在CDS区域,并且在起始密码子和终止密码子处有显著的堆积。在基因组浏览器中查看时,两者的模式一眼就能区分。
-
FLAG字段的可能差异:
- 如果链特异性建库方式不同,或者数据处理流程对链的推断方式不同,代表正负链的FLAG位也会不同。
-
总结
| 文件类型 | 是否相同 (RNA-seq vs Ribo-seq) | 原因 |
|---|---|---|
| HISAT2 索引文件 (.ht2) | 相同 | 基于相同的参考基因组构建。 |
| HISAT2 比对输出文件 (BAM/SAM) | 完全不同 | 1. 比对参数不同 (Ribo-seq禁用剪接)。 2. CIGAR字符串不同 (RNA-seq有N,Ribo-seq没有)。 3. 基因组位置分布不同(RNA-seq均匀,Ribo-seq集中于CDS)。 |
所以,你的流程是:
- 用 相同的 HISAT2索引文件。
- 用 不同的 HISAT2比对参数。
- 最终生成 内容完全不同的 BAM文件,以反映其背后完全不同的生物学现象。
3.3 也就是RNAseq和riboseq的原始数据就不同,故而使用同样的hisat2工具,用同一参考基因组进行比对后得到的hisat2文件的内容也不同,且后续生成的bam也不同,是吗,如有不对,请指出并纠正
您的理解几乎完全正确,但有一个非常关键的点需要做更精确的纠正。
您的陈述是:
"RNAseq和riboseq的原始数据就不同,故而使用同样的hisat2工具,用同一参考基因组进行比对后得到的hisat2文件的内容也不同,且后续生成的bam也不同"
我们来逐一分析:
1. 关于"原始数据就不同"
您的这个观点是完全正确的。
"原始数据不同"是导致后续所有差异的根源。这种不同体现在:
- 片段来源:RNA-seq来自随机断裂的整个转录本;Ribo-seq来自被核糖体保护的特定短片段。
- 片段长度:RNA-seq读长分布较广;Ribo-seq读长集中在28-30个核苷酸。
- 序列特征:Ribo-seq读段具有三核苷酸周期性,而RNA-seq没有。
2. 关于"使用同样的hisat2工具,用同一参考基因组"
这也是正确的。
您使用同一个软件和同一套参考基因组索引。
3. 关于"得到的hisat2文件的内容也不同"
这里需要精确纠正:您可能混淆了"HISAT2文件"的类型。
在上一轮回答中我们解释了,运行HISAT2会涉及两种文件:
- 索引文件 :是
.ht2文件。 - 比对输出文件 :通常是 SAM/BAM文件。
纠正后的表述是:
- 索引文件是相同的。
- 比对输出文件是不同的。
所以,更准确的说法是:"...进行比对后得到的比对结果文件(SAM/BAM) 的内容也不同"。
4. 关于"后续生成的bam也不同"
这是正确的,并且是核心结论。
BAM文件就是比对输出的最终格式,它的不同是实验差异和不同比对参数共同作用下的必然结果。
纠正后的完整、精确表述
"RNA-seq和Ribo-seq的原始FASTQ数据因其建库原理不同而存在本质差异(长度、分布、特征)。因此,尽管使用相同的HISAT2工具和相同的参考基因组索引文件,但在比对时通常需要为Ribo-seq设置特殊参数(如禁用剪接比对)。这种 数据本身的差异与 比对参数的差异共同导致了两者最终输出的BAM文件在内容上存在根本不同(例如,CIGAR字符串中是否包含内含子跳过'N',以及读段在基因组上的分布模式)。"
用一个简单的流程图来总结:
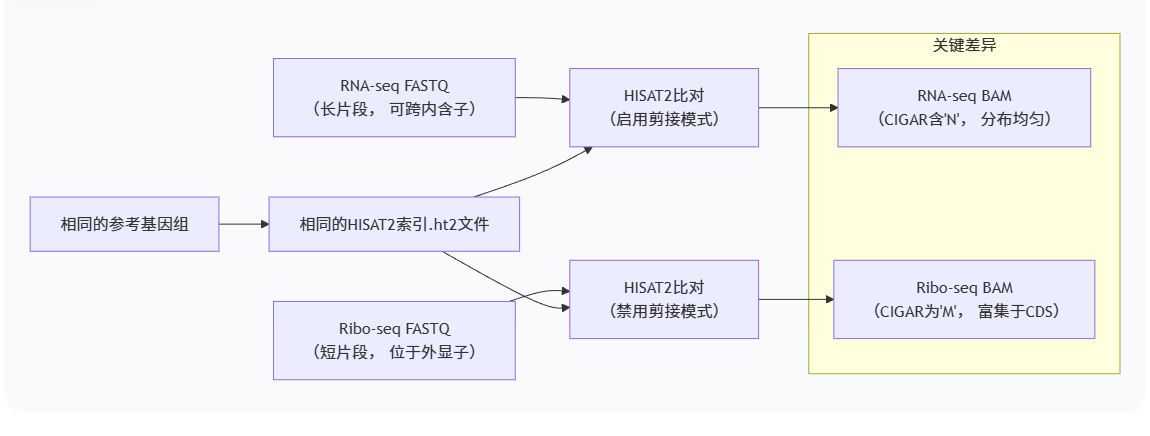
您的理解已经非常到位,只是在术语的精确性上稍有偏差。这个纠正非常重要,因为它能帮助您更清晰地理解生物信息学分析中每个文件的具体角色和生成流程。
3.4 "相同的参考基因组索引文件"参考基因组的索引文件是怎样生成的,什么情况下参考基因组的索引文件会相同
非常好,这个问题触及了生物信息学可重复性的核心。我们来详细拆解一下。
参考基因组索引文件是怎样生成的?
参考基因组的索引文件是通过HISAT2软件包中的 hisat2-build 命令生成的。这是一个预计算的过程,目的是将庞大的基因组序列转换为一个高效的、可供快速搜索的数据结构。
基本命令如下:
bash
hisat2-build -p <线程数> <输入参考基因组.fasta> <输出索引文件的基础名称>示例:
bash
hisat2-build -p 8 Homo_sapiens.GRCh38.dna.primary_assembly.fa grch38_index运行后会生成一系列文件:grch38_index.1.ht2, grch38_index.2.ht2, grch38_index.3.ht2 ... 这些文件共同构成了HISAT2索引。
生成过程中发生了什么?
- 读取FASTA :软件读取参考基因组的FASTA文件(如
Homo_sapiens.GRCh38.fa)。 - 构建全局FM-index :这是核心算法。它将整个基因组序列转换成一个后缀数组 和Burrows-Wheeler变换 的数据结构。这使得软件能够极其快速地在亿万个碱基中定位一个短字符串(你的测序读段)的可能位置。
- 构建局部索引:为了处理剪接比对,HISAT2还会构建全基因组和局部基因组区域的索引,以高效地找到跨越外显子-外显子边界的读段。
- 序列化保存 :将构建好的复杂数据结构序列化并保存到一系列
.ht2文件中。
什么情况下参考基因组的索引文件会相同?
这是一个关键问题,因为索引文件的"相同"是保证分析结果可比性和可重复性的绝对前提 。两个索引文件在以下条件全部满足时,才能被认为是相同的:
1. 完全相同的参考基因组FASTA文件
这是最根本的条件。即使基因组版本号相同(比如都是GRCh38),如果来源或构建方式不同,也可能导致文件内容不同。
- 来源 :必须来自同一数据库(如GENCODE, Ensembl, UCSC)的同一版本。
- 不相同示例 :
GRCh38.p14与GRCh38.p13是不同的;GENCODE v44与ENSEMBL release 110的GRCh38也可能有细微差别。
- 不相同示例 :
- 文件内容 :文件的MD5校验和必须完全一致。这意味着:
- 序列内容完全相同。
- 染色体命名规则完全相同(例如
chr1vs1)。 - 线粒体染色体命名完全相同(例如
chrMvsMT)。 - 包含的染色体/contig集合完全相同。
2. 完全相同的HISAT2软件版本
不同版本的HISAT2可能使用更新或略有不同的算法来构建索引。即使输入相同,用v2.1.0和v2.2.1构建的索引文件也可能不兼容。
- 必须使用相同的主版本号和次版本号。
3. 完全相同的构建参数
hisat2-build 命令有一些可选的参数,这些参数会改变索引的构建方式。
--large-index: 对于大型基因组(如人类、小鼠),如果默认索引超过4GB,需要使用此参数。用和不用此参数生成的索引是不同的。- 其他参数 :如
--bmax,--dcv等高级参数,如果指定了,必须完全一致。 - 最常见的情况是:都使用默认参数。
总结一下,确保索引相同的"黄金法则":
使用来自同一来源、MD5校验和完全一致的FASTA文件,在完全相同的HISAT2软件版本下,使用完全相同的命令行参数,重新运行
hisat2-build命令。
在实际分析中的意义
-
为什么强调索引要相同?
- 可重复性:你昨天跑的分析,今天换了一个索引,结果可能就有微妙的差异,这无法接受。
- 合作共享:当多个实验室合作时,必须统一使用完全相同的参考基因组和索引,否则结果无法直接比较。
- 流程自动化:在生信流程中,索引的生成是独立的、可复用的步骤。一旦生成,所有后续分析都应基于此索引。
-
给你的建议:
- 记录 :在你的实验记录或分析脚本中,明确记录你使用的参考基因组FASTA文件的确切来源、版本号和MD5值 ,以及构建索引的HISAT2版本和完整命令。
- 共享:在团队内部分享构建好的索引文件,而不是让每个人自己构建,以避免意外的不一致。
- 对于RNA-seq和Ribo-seq :只要你遵循了上述"黄金法则",那么你就可以放心地使用同一套索引文件来处理同一物种的RNA-seq和Ribo-seq数据。它们的不同将只体现在比对参数和最终的BAM文件内容上,而索引基础是统一和可靠的。
所以,回到你的工作流,正确的理解是:你为人类GRCh38构建一套HISAT2索引,然后用这套完全相同的索引,分别以不同的参数去比对RNA-seq和Ribo-seq的FASTQ数据,从而得到内容迥异的BAM文件。
3.5 总结以上所有提问,系统详细地总结回答给我
好的,让我们对以上所有讨论进行一次系统、详细的总结。这将帮助您彻底理解从原始数据到比对结果的整个流程,以及RNA-seq和Ribo-seq在其中的核心区别。
全面总结:RNA-seq与Ribo-seq的HISAT2比对分析
第一部分:核心概念定义
-
HISAT2是什么?
- 它是一个软件工具,主要用于将高通量测序产生的短读段快速、准确地比对到参考基因组上。
- 它包含两个主要功能:
hisat2-build: 为参考基因组构建索引。hisat2: 执行实际的比对任务。
-
"HISAT2文件"指什么?
- 这是一个需要精确的术语,通常指两种文件:
- 索引文件 : 由
hisat2-build生成的一系列以.ht2为后缀的文件。它是供软件快速搜索的数据库,用户不可读。 - 比对输出文件 : 由
hisat2比对命令生成的 SAM文件 或其二进制格式 BAM文件 。它记录了每个读段比对到基因组的详细信息,是分析的核心结果。
- 索引文件 : 由
- 这是一个需要精确的术语,通常指两种文件:
-
参考基因组索引文件如何生成?
- 命令 :
hisat2-build -p [线程数] [参考基因组.fasta] [输出索引基础名] - 过程 : 该命令读取参考基因组FASTA文件,通过复杂的算法(如BWT、FM-index)构建一个高效的数据结构,并保存为多个
.ht2文件。 - 索引文件相同的条件(黄金法则) :
- 完全相同的 参考基因组FASTA文件(来源、版本、MD5校验和均一致)。
- 完全相同的 HISAT2软件版本。
- 完全相同的
hisat2-build构建参数(通常使用默认参数即可)。
- 命令 :
第二部分:RNA-seq与Ribo-seq的流程对比与分析
下面的流程图和表格清晰地展示了两者的异同:
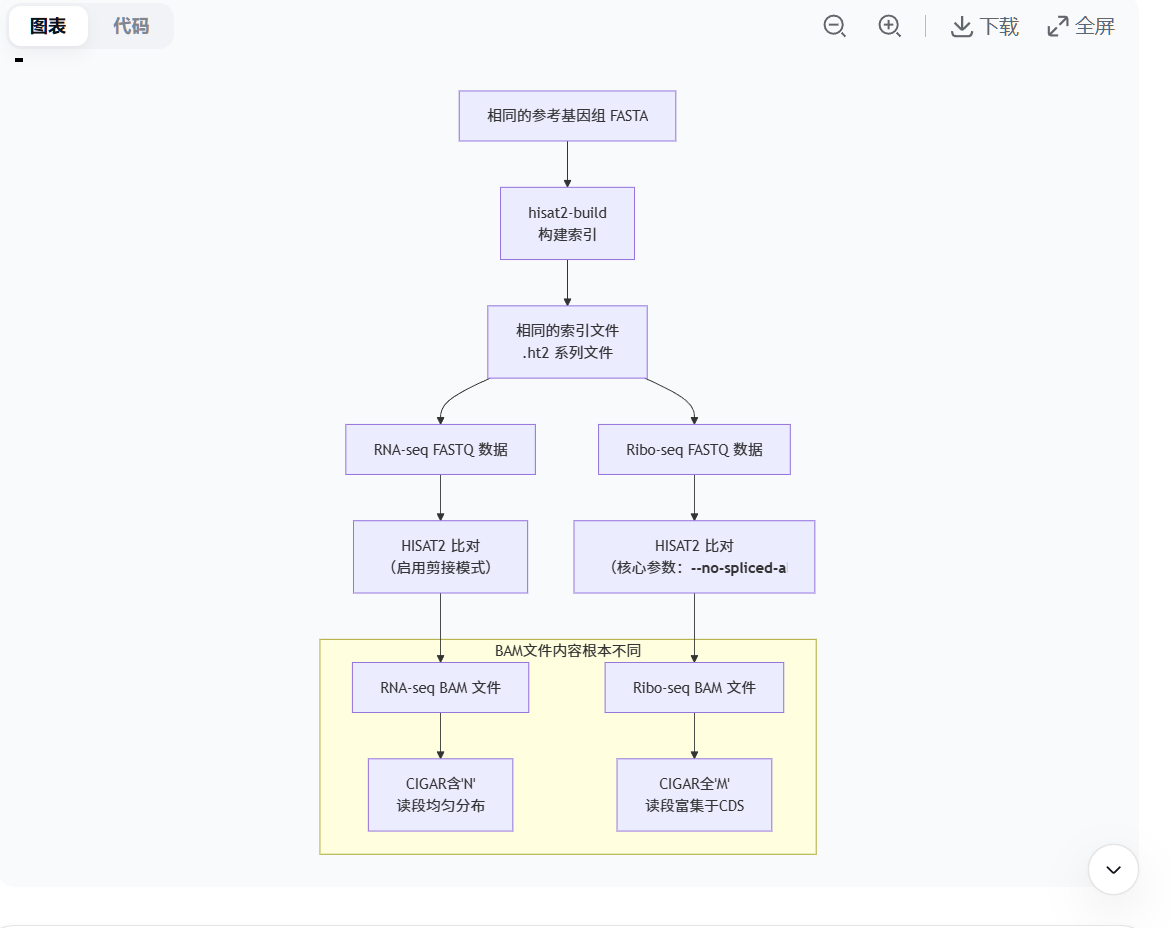
| 环节 | 相同点 | 不同点 | 原因与解释 |
|---|---|---|---|
| 1. 原始数据(FASTQ) | 都是测序产生的短读段序列 | 本质不同 | 实验目的不同 : • RNA-seq : 随机断裂的整个转录本,反映转录本的存在与丰度 。读长分布较广。 • Ribo-seq : 核糖体保护的特定短片段(~28-30nt),反映翻译的动态 。读长集中,具有三核苷酸周期性。 |
| 2. 参考基因组与索引 | 完全相同 | 无 | 因为分析的是同一物种,使用相同的参考基因组FASTA文件,按照"黄金法则"生成的索引文件自然是同一套。 |
| 3. HISAT2比对参数 | 使用同一个hisat2命令 |
关键参数不同 | 由数据特性决定 : • RNA-seq : 使用标准参数,特别是启用剪接比对 ,因为读段会跨越外显子-内含子边界。 • Ribo-seq : 必须使用 --no-spliced-alignment (或类似参数),因为短足迹来自单个外显子,强制进行连续比对以避免假阳性。 |
| 4. 输出BAM文件 | 都是标准的BAM格式 | 内容根本不同 | 这是不同流程的必然结果 : • RNA-seq BAM : - CIGAR字符串 : 包含大量 N(代表内含子跳过)。 - 基因组分布 : 读段相对均匀地分布在基因的整个长度上(5'UTR, CDS, 3'UTR)。 • Ribo-seq BAM : - CIGAR字符串 : 几乎全是 M(连续匹配),没有 N。 - 基因组分布 : 读段高度密集地堆积在蛋白质编码区,在起始和终止密码子处有显著富集,并呈现三核苷酸周期性。 |
第三部分:核心结论与误区澄清
- 原始数据是根源:RNA-seq和Ribo-seq的FASTQ数据从根源上就是不同的,这决定了后续所有分析策略的差异。
- 工具与基础相同 :两者使用相同的HISAT2工具 和完全相同的参考基因组索引文件。这是进行分析的公共基础。
- 比对策略是关键 :必须根据数据特性调整比对参数。对Ribo-seq禁用剪接比对是核心步骤,否则会产生大量错误结果。
- 结果文件是体现 :最终生成的BAM文件内容的差异(如CIGAR字符串、读段分布)不是错误,而是正确分析后对底层不同生物学过程的真实反映。RNA-seq展示了"哪些基因被转录",而Ribo-seq展示了"哪些mRNA正在被如何翻译"。
一句话总结:
您使用同一套HISAT2索引,但针对RNA-seq和Ribo-seq这两种生物学本质不同的实验数据,采用了不同的比对策略,从而生成了内容迥异但各自正确的BAM文件,以分别揭示转录组和翻译组的奥秘。