本文来源:k学长的深度学习宝库,点击查看源码&详细教程。深度学习,从入门到进阶,你想要的,都在这里。包含学习专栏、视频课程、论文源码、实战项目、云盘资源等。

1、研究背景与动机
医学图像分割的背景
医学图像分割(Medical Image Segmentation)是医疗 AI 领域的核心任务之一。它的目标是把 CT、MRI 等影像中的器官或病灶准确地勾画出来,为 疾病诊断、手术规划、放疗靶区划定 等提供关键支持。
传统方法:早期主要依赖人工描绘,费时费力且主观性强。
深度学习方法:自从卷积神经网络(CNN)发展起来之后,全卷积网络(FCN)和 U-Net 成为主流,极大提高了分割的自动化和准确性。
其中 U-Net(2015 年提出)凭借 编码器-解码器对称结构 和 跳跃连接(skip connection),在医学图像分割领域几乎成为标准方案。
CNN 的局限性
虽然 U-Net 及其变体表现优秀,但 CNN 有一个天然短板:
卷积核的感受野是有限的,属于 局部建模。
想要捕捉 全局依赖关系(long-range dependency) 时,需要多层堆叠,但这会带来计算开销大、效果受限的问题。
这意味着 CNN 在处理 形状差异大、结构复杂 的器官时,可能不能充分理解全局上下文信息,导致分割边界不够精细。
Transformer 的优势与不足
Transformer 在自然语言处理(NLP)中大获成功,它依靠 自注意力机制(Self-Attention),可以:
有效捕捉 全局上下文;
经过大规模预训练后,具有强大的迁移能力。
视觉领域的 ViT(Vision Transformer) 也证明了 Transformer 在图像分类中的潜力。但问题在于:
Transformer 会把图像切成 patch(小块),再当作序列输入,因此得到的特征分辨率低;
它对 细粒度的空间信息 处理不足,导致定位精度下降。 在医学图像分割中,这种缺陷尤其致命,因为需要非常精确的边界。
TransUNet 的提出动机
综上,研究者们意识到:
CNN → 适合局部建模(捕捉低层次细节)
Transformer → 适合全局建模(理解长程依赖)
于是 TransUNet 被提出,作为首个将 Transformer 与 U-Net 结合 的医学图像分割框架:
编码器:利用 CNN 特征图做 patch embedding,再送入 Transformer,融合全局上下文。
解码器:借助 U-Net 的跳跃连接,把高分辨率的 CNN 特征与 Transformer 编码特征结合,恢复细节,提升定位能力。
这样,TransUNet 同时解决了 CNN 缺乏全局信息 和 Transformer 缺乏局部细节 的问题。实验表明,它在多器官 CT 分割和心脏 MRI 分割中性能优于多种现有方法
2102.04306v1_translated
。
👉 总结一句话: TransUNet 的动机就是想把 CNN 的"看局部细节"能力和 Transformer 的"抓全局关系"能力结合起来,用于医学图像分割这种既要求全局理解又要求边界精细的任务。
2、核心创新点
CNN 与 Transformer 的混合编码器
TransUNet 并没有直接把原始图像分块输入 Transformer,而是:
先用 CNN(ResNet-50) 提取图像的低层特征;
再将这些特征划分为 patch,送入 Vision Transformer (ViT)。
这样做有两个好处:
CNN 先把局部边缘、纹理等低层特征提取出来,避免 Transformer 在缺少空间细节的情况下"摸不着边";
CNN 中间层的高分辨率特征图还能在解码阶段通过跳跃连接(skip connection)用来恢复精细结构。
创新点:通过混合式设计,把 CNN 的局部优势和 Transformer 的全局优势结合了起来
2102.04306v1_translated
。
U-Net 风格的跳跃连接(Skip Connections)
传统 Transformer 在下采样后得到的特征分辨率比较低,容易丢失细节。TransUNet 借鉴了 U-Net 的思想:
在不同分辨率层级,引入 跳跃连接,把 CNN 的高分辨率特征与 Transformer 输出的语义特征融合;
最终在解码器中逐步恢复分辨率,实现 精确定位 + 全局理解。
创新点:把经典 U-Net 的 skip connection 融合到 Transformer 架构里,让分割结果更细致,边界更清晰
2102.04306v1_translated
。
级联上采样解码器(CUP, Cascaded Upsampler)
TransUNet 没有直接用"粗暴上采样"来恢复图像,而是设计了 级联上采样器(CUP):
多个上采样模块层层递进,每个模块包括 上采样算子 + 卷积层 + ReLU 激活;
在不同分辨率尺度与编码器的 CNN 特征做融合。
创新点:CUP 能逐步细化特征图,还能借助 skip connection 把空间细节补回来,比单次大尺度上采样更稳定、更精确
2102.04306v1_translated
。
Transformer 作为医学图像分割的编码器
这是第一次把 Transformer 引入医学图像分割 任务:
Transformer 的自注意力机制提供了强大的 全局上下文建模能力;
通过与 CNN 融合,解决了纯 Transformer 分辨率不足、纯 CNN 全局信息不足 的缺陷。
创新点:在分割框架中引入 ViT 并与 U-Net 有机结合,为医学影像分割开辟了新思路
2102.04306v1_translated
。
实验验证与性能突破
TransUNet 在多个数据集(Synapse 多器官 CT、ACDC 心脏 MRI)上的实验表明:
相比 CNN-only(如 U-Net、AttnUNet),能捕捉更多全局语义,误分割更少;
相比 Transformer-only(如 ViT-CUP),能更好恢复边界细节。 最终在 DSC(Dice Score)和 Hausdorff Distance 等指标上均显著优于对比方法。
🔑 总结一下核心创新点:
CNN + Transformer 混合编码器 → 结合局部细节与全局上下文。
U-Net 式跳跃连接 → 弥补 Transformer 的定位能力不足。
级联上采样解码器(CUP) → 分层恢复分辨率,更精准。
首次探索 Transformer 在医学图像分割中的应用 → 推动了新的研究方向。
显著的实验效果 → 在多器官分割和心脏分割任务上均刷新了性能纪录。
3、模型的网络结构
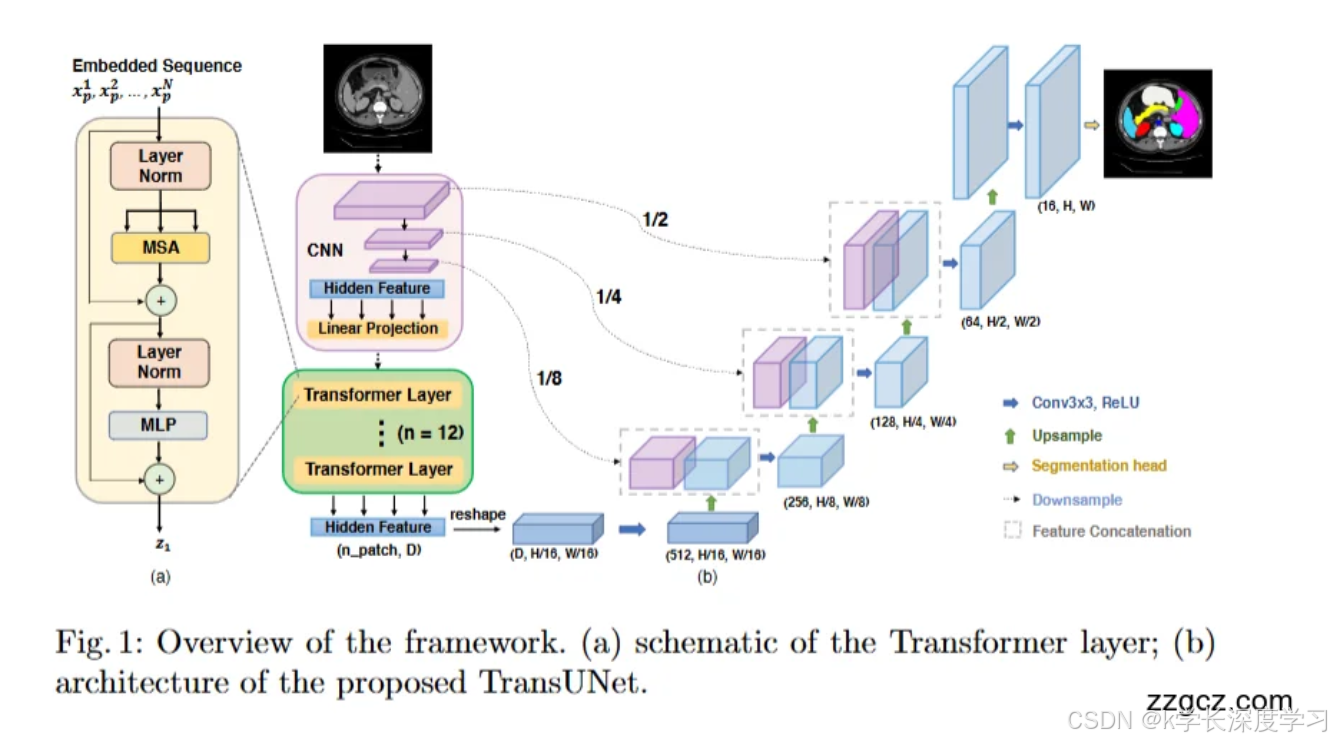
输入与特征提取(CNN Backbone)
输入:一张医学影像(如 CT、MRI 切片),尺寸为 H×W。
CNN Backbone(ResNet-50 等):首先对图像进行卷积特征提取,得到多层特征图:
1/2 1/4 1/8 分辨率的特征会被保留,作为后续 跳跃连接(skip connection)的输入
这些特征包含了 低层次的纹理、边缘和空间细节。
Transformer 编码器(全局上下文建模)
从 CNN 得到的最后一层特征图,会被划分为固定大小的 patch(小块),再 展平 成序列。
经过 线性投影(Linear Projection) 映射到 D 维嵌入空间,并加上 位置编码(Positional Embedding) 来保留位置信息。
这些序列会依次经过 12 层 Transformer Layer(Multi-head Self Attention + MLP + 残差连接 + LayerNorm)。
输出的结果是一个全局上下文特征序列,能够捕捉远距离依赖关系。
👉 图中左边 (a) 就是一个 Transformer 层的结构示意图。
特征重塑(Reshape)
Transformer 输出的序列 (npatch,D) 会被 重塑回空间特征图:
尺寸为 (D,H/16,W/16)。
注意:这里的空间分辨率被压缩了 16 倍,所以需要后续的解码器来逐步恢复。
解码器(CUP:级联上采样器)
解码器部分是 U-Net 风格:
使用 逐层上采样模块(每个模块包含:上采样算子 + Conv3×3 + ReLU);
在1/16 1/8 1/4 1/2 1/1 的过程中,逐步恢复空间分辨率;
每个阶段都会把 Transformer 的解码特征 与对应分辨率的 CNN 特征做拼接(Feature Concatenation);
最终得到与原图一样大小的特征图 (H,W)。
输出(Segmentation Head)
在最后一层,会接一个 卷积层 + 上采样,生成最终的 分割掩码(Segmentation Mask);
掩码的每个像素点对应类别标签(如不同器官的区域)。
🔑 网络结构总结
TransUNet = CNN + Transformer + U-Net 式解码器
CNN Backbone:提取局部空间特征,提供跳跃连接。
Transformer 编码器:对 patch 序列进行全局建模,获得长程依赖。
Reshape + CUP 解码器:逐层上采样 + 拼接高分辨率特征,恢复细节。
Segmentation Head:输出像素级分割结果。
这张图就是一个很直观的流程图:从左到右就是 输入 → CNN → Transformer → Reshape → 解码器(带跳跃连接) → 输出分割图。
4、存在的重大缺陷
计算和显存开销大
Transformer 编码器需要处理图像 patch 序列,注意力机制的复杂度是 O(N2),其中 NNN 是 patch 数量。
当输入分辨率高时(例如 512×512 图像),序列长度急剧增加,导致 计算量和显存消耗爆炸。
在医学影像场景(CT/MRI 常是高分辨率 3D 数据)下尤其严重,难以在普通 GPU 上训练和推理
对数据依赖过强
Transformer 本身参数量大,需要 大量训练数据 才能发挥优势。
医学图像分割的数据标注极其昂贵(需要专业医生手动标注),数据量往往有限。
如果训练数据不足,TransUNet 容易出现 过拟合 或 泛化能力下降 的问题。
Transformer 特征缺乏空间精细度
尽管加入了 U-Net 式跳跃连接,但 Transformer 部分还是基于 patch 表示,导致:
对 小器官(如胆囊、胰腺) 的边界恢复不够细腻;
在复杂形态或低对比度区域(如肿瘤、血管分叉)容易出现 边界模糊或定位误差。
本质上,Transformer 在分割任务上仍不如 CNN 对 局部空间细节 的捕捉自然。
模型训练和部署成本高
需要 预训练模型(ImageNet 上的 CNN+ViT backbone) 来初始化,否则效果显著下降。
对医疗机构而言,这样的模型对硬件和软件环境要求较高,不利于快速部署。
推理速度较慢,不适合 实时临床应用(比如术中导航)。
三维扩展的局限性
TransUNet 主要在 2D 切片(CT/MRI 横断面)上做实验。
医学影像往往是 3D 体数据,需要在三维空间中捕捉结构关系。
将 TransUNet 扩展到 3D 时,显存需求和计算成本会进一步放大,导致在实际临床场景下 难以应用。
🔑 总结:TransUNet 的重大缺陷
计算复杂度高 → 注意力机制在高分辨率下难以扩展。
依赖大规模标注数据 → 医学数据稀缺时表现不稳定。
细粒度空间建模不足 → 小器官或复杂边界的分割仍存在偏差。
训练与部署成本高 → 需要预训练和高端硬件支持。
3D 场景适应性差 → 在完整体数据上的可扩展性有限。
5、后续基于此改进创新的模型
A. 纯 Transformer 的 U 形结构
Swin-UNet(2021) 用分层 Swin Transformer(移位窗口注意力) 做成完整 U 形的编解码器;不再依赖 CNN 主干,靠层级 token 金字塔实现局部--全局建模与高效性。适合2D,多器官/心脏数据上表现突出。 arXiv
B. 3D 体数据方向(体素级分割)
UNETR(2021/2022) 直接用 Transformer 编码器连接到解码器的多个尺度跳连,从设计上就是为 3D 体数据 而生,显著提升 BTCV/MSD 等 3D 任务;可视作把 TransUNet 的"Transformer+U-Net 跳连"思想系统化到 3D。 arXiv+1
Swin-UNETR(2022) 在 UNETR 框架里把编码器换成 Swin Transformer 的分层/移位窗口版本,解码仍为 FCN,并在多尺度处做跳连;对 3D 脑肿瘤等任务很强。后续还有 Swin-UNETR-V2 加强版。 arXiv+2Google Colab+2
Swin-Unet3D(2023) 进一步把 Swin-UNet 思路做成 三维纯 Transformer U 形结构,并尝试并行 CNN+ViT 特征以兼顾局部与全局。 BioMed Central
C. 轻量高效/混合式改进
LeViT-UNet(2021) 用更高效的 LeViT 作为编码器,在 速度-精度权衡 上优于常规 ViT;仍保持 U-Net 跳连,面向部署友好。 arXiv+1
UTNet(2021) 典型的 CNN×Transformer 混合:在 U-Net 中引入自注意力并做 空间降采样注意力 以降复杂度,延续 TransUNet "融合局部+全局"的思路但更注重效率。 ACM Digital Library+2miccai2021.org+2
D. 编码器/模块层面的 Transformer 强化
MISSFormer(2021/2023 TMI) 设计 增强型 Transformer Block 与 上下文桥(Context Bridge),同时强化长程依赖与本地上下文,强调 从零训练也能强 的鲁棒性。可看作把 TransUNet 的"全局+局部"做得更精细。 arXiv+2PubMed+2
E. 特定任务的混合框架
TransBTS(2021,脑肿瘤) 3D CNN 编码器 + Transformer 全局建模 + 解码器恢复 的混合范式,在 BraTS 上验证;与 TransUNet 同宗同源,但更聚焦多模态 3D 脑肿瘤。 arXiv+2SpringerLink+2
本文来源:k学长的深度学习宝库,点击查看源码&详细教程。深度学习,从入门到进阶,你想要的,都在这里。包含学习专栏、视频课程、论文源码、实战项目、云盘资源等。