本方案具备一定的局限性。
是以brainstorm的默认图谱为依据来进行构建的。进行的是按照brainstorm的标准的皮层标签来重构的。
首先,你需要配置matlab的brainstorm这个插件,然后,对于默认的Default anatomy,是有比较多的模板和皮层分区的。其支持的内容如下:
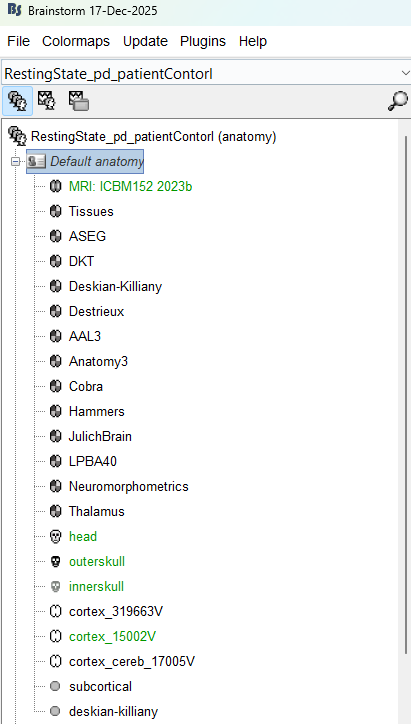
你可以使用对应的空间标准。
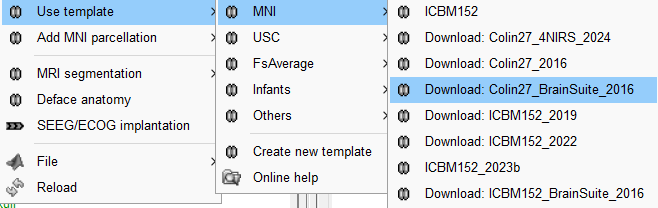
添加MNI为标准的Parcellation。
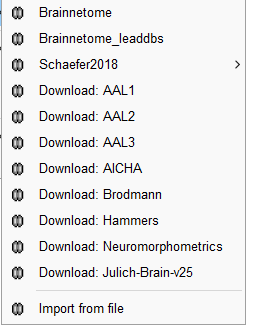
有些下载可能需要VPN。
可以和大多数标准进行统一。
如果研究只使用就是皮层标签级别的对齐的话。
包括ebrains的Julich-Brains(细胞类型的图谱)的siibra。其大脑模板有Colin27的模板。
siibra具备一定的图谱搜索和对应的功能,但是其支持的图谱相对有限。
Ebrains支持的脑模板标准。
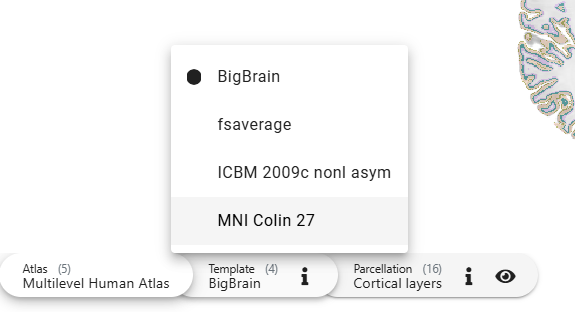
操作过程
在默认的Default anatomy
我们首先要确定使用的MNI的某个模板。
下载,如我使用的是BrainICBM152
第二步骤,添加你需要的进行转换的Parcellation。

第三步,使用CAT12,或者下面的可以生成Cortex的都可以。当然freesurfer可以用于个体的模板生成,且功能强大。但是这个操作界面和流程较为简单。
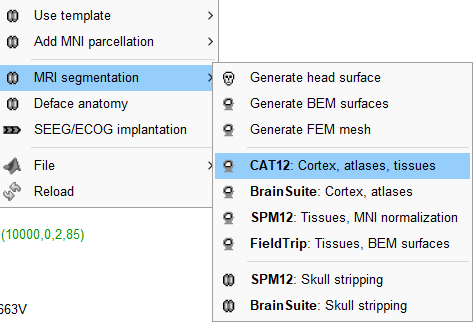
当我们下载了Parcellation之后,就可以生成
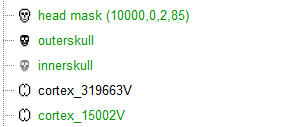
我们主要使用较小的cortex_15002V。
当我们把这个文件导出到matlab时,我们就会得到一个结构为下面的文件。
注意如果直接导出格式是mesh,什么的就只会有mesh和vertices.
导出到matlab。
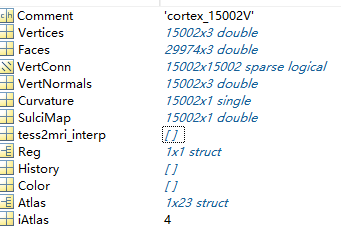
我们可以看到有下面的结构。
上面的Verices是和Faces是可视化支持的。
Atlas,可以来获取对应的Scouts
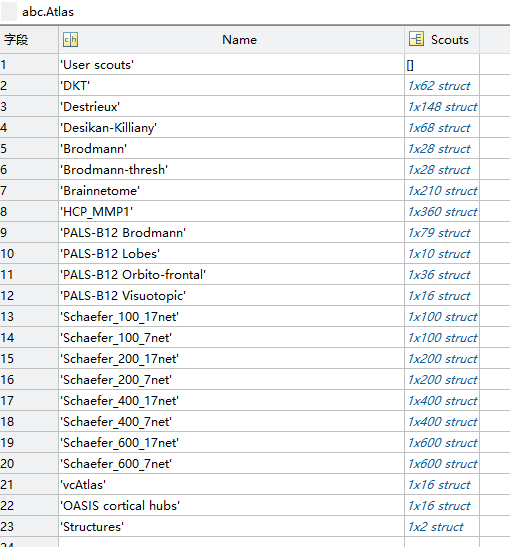
此处我下载的有上述这些模板的
通过下面的程序,我们可以对这个不同模板进行图谱到功能网络的加权概率映射.
首先直观的看,肯定使用较为细分的图谱Schaefer_600_7net进行功能网络映射具备较高精度。
我们使用Vertics的匹配范围并判定其label的所属来判断最终属于什么功能网络分区。
需要三个函数
matlab
function map = atlas_to_yeo7(atlas_scouts, schaefer600_7net)
% 将任意 atlas 映射到 Yeo 7-network
% 通过 Schaefer_600_7net 的顶点重叠实现
%
% atlas_scouts : 任意 atlas 的 Scouts
% schaefer600_7net : Schaefer_600_7net Scouts
%
% map(i) 包含:
% roi_index
% roi_label
% roi_region
% network_names
% network_id
% network_weights
nROI = length(atlas_scouts);
nSCH = length(schaefer600_7net);
% ---------- 提取 Schaefer 顶点 ----------
SCH_vertices = cell(nSCH,1);
SCH_netname = cell(nSCH,1);
SCH_netid = zeros(nSCH,1);
for j = 1:nSCH
SCH_vertices{j} = schaefer600_7net(j).Vertices(:);
[SCH_netname{j}, SCH_netid(j)] = parse_yeo7_label(schaefer600_7net(j).Label);
end
% ---------- 主循环 ----------
map = struct([]);
for i = 1:nROI
vA = atlas_scouts(i).Vertices(:);
rA = atlas_scouts(i).Region;
overlap_net = zeros(7,1);
for j = 1:nSCH
% 左右半球约束(非常重要)
if rA(1) ~= schaefer600_7net(j).Region(1)
continue;
end
% 顶点重叠
ov = numel(intersect(vA, SCH_vertices{j}));
if ov > 0
overlap_net(SCH_netid(j)) = overlap_net(SCH_netid(j)) + ov;
end
end
% 归一化
if sum(overlap_net) > 0
overlap_net = overlap_net / sum(overlap_net);
end
idx = find(overlap_net > 0.05); % 5% 阈值(可调)
map(i).roi_index = i;
map(i).roi_label = atlas_scouts(i).Label;
map(i).roi_region = atlas_scouts(i).Region;
map(i).network_id = idx;
map(i).network_names = yeo7_id_to_name(idx);
map(i).network_weights = overlap_net(idx);
end
end
matlab
function [name, id] = parse_yeo7_label(label)
% 从 Schaefer Label 中解析 Yeo 7-network
if contains(label, 'Vis')
name = 'Vis'; id = 1;
elseif contains(label, 'SomMot')
name = 'SomMot'; id = 2;
elseif contains(label, 'DorsAttn')
name = 'DorsAttn'; id = 3;
elseif contains(label, 'SalVentAttn')
name = 'SalVentAttn';id = 4;
elseif contains(label, 'Limbic')
name = 'Limbic'; id = 5;
elseif contains(label, 'Cont')
name = 'Cont'; id = 6;
elseif contains(label, 'Default')
name = 'Default'; id = 7;
else
error('Unknown Yeo network in label: %s', label);
end
end
matlab
function names = yeo7_id_to_name(ids)
dict = {'Vis','SomMot','DorsAttn','SalVentAttn','Limbic','Cont','Default'};
names = dict(ids);
end调用
matlab
%加载模板数据atlas_struct.mat
%加载格式如下面的
% 最简单的方式 - 将数据加载到工作区
load('filename.mat')
% 指定完整路径
load('C:\path\to\your\data\filename.mat')
% 路径的别称aaaa
Desikan_Killiany = aaaa.Atlas(4).Scouts;
Brainnetome = aaaa.Atlas(7).Scouts;
Schaefer_600_7net = aaaa.Atlas(20).Scouts;
map_Brainnetome_yeo7 = atlas_to_yeo7(Brainnetome, Schaefer_600_7net);可以得到加权roi映射到功能网络的数据