基于 2025 年 8 月发表于《npj Digital Medicine》的该论文(DOI: 10.1038/s41746-025-01955-x),以下从 10 个核心维度详细拆解内容:
一、研究问题
许多罕见遗传病具有可识别的面部表型(如颅缝早闭综合征的下颌前突、眼窝浅等特征),这些表型是临床诊断的关键线索。然而,当前大语言模型(LLMs)在该领域的应用存在显著局限,无法有效支撑罕见遗传病的精准诊断,具体问题聚焦于:
- 如何利用面部表型与基因、疾病的关联关系,提升 LLMs 对罕见遗传病的诊断能力;
- 如何解决 LLMs 在专业医疗场景中 "幻觉生成错误信息" 和 "缺乏领域知识" 的核心问题,确保诊断结果的准确性与可靠性。
二、面临挑战
论文明确指出 LLMs 在面部表型相关罕见遗传病应用中的三大核心挑战:
- LLMs 的幻觉问题(Hallucination):LLMs 可能生成看似合理但实则错误或无依据的信息(如将 A 遗传病的面部表型错误归因于 B 遗传病),在医疗诊断中可能导致严重后果。
- 领域知识缺失(Lack of specialized domain knowledge):LLMs 虽擅长自然语言理解与生成,但缺乏对面部表型、基因、罕见遗传病三者复杂关联的深度认知(如无法识别 "基因 - 变异 - 疾病 - 表型" 的多跳关联),难以处理专业查询。
- 现有检索增强方法的局限 :传统检索增强生成(RAG)依赖 dense vector 相似性检索,对复杂查询(如多实体关联查询)处理能力不足,且易因多源相似信息导致冗余回复;同时,缺乏面部表型相关的领域知识图谱(KG),无法为 LLMs 提供结构化、高精度的专业知识支撑。
【
Dense Vector(稠密向量)相似性检索,是将数据(文本、图像、音频等)转化为高维稠密向量后,通过计算向量间相似度来找到 "内容相似数据" 的技术,核心解决 "从海量数据中快速定位相似信息" 的问题,广泛用于推荐系统、搜索引擎、图像检索等场景。
1. 核心概念:什么是 Dense Vector(稠密向量)?
- 定义 :与 "稀疏向量"(仅少数维度有值,如 One-Hot 编码)相反,稠密向量的每个维度都有非零值 ,且每个维度都承载数据的 "语义 / 特征信息"。例:用模型将文本 "猫" 转化为稠密向量
[0.21, -0.56, 0.89, ..., 0.12](假设 128 维),其中每个数值都对应 "猫的生物属性、外形特征、常见场景" 等隐含信息。 - 生成方式:依赖深度学习模型(如 NLP 中的 BERT、图像中的 ResNet),通过模型对原始数据的 "语义 / 特征理解",自动映射为固定维度的稠密向量(即 "Embedding 嵌入" 过程)。
2. 核心逻辑:如何通过 "向量相似" 判断 "内容相似"?
核心假设是:内容相似的数据,其对应的稠密向量在高维空间中的 "距离" 更近。通过计算向量间的 "相似度指标" 量化这种 "距离",常用指标有 3 种:
| 相似度指标 | 计算逻辑 | 特点 |
|---|---|---|
| 余弦相似度(Cosine Similarity) | 计算两向量夹角的余弦值,范围 [-1,1],值越接近 1 则方向越一致 | 忽略向量 "长度"(如文本长短),更适合语义相似判断 |
| 欧氏距离(Euclidean Distance) | 计算两向量在高维空间中的直线距离,值越小越相似 | 受向量 "长度" 影响,适合特征维度数值范围一致的场景(如图像像素) |
| 曼哈顿距离(Manhattan Distance) | 计算两向量各维度差值的绝对值之和,值越小越相似 | 对异常值更稳健,适合低维或特征分布较离散的场景 |
3. 关键挑战与解决思路:从 "暴力计算" 到 "高效检索"
若直接对海量向量(如百万 / 亿级)逐一计算相似度(暴力检索),会因 "高维 + 海量" 导致速度极慢,因此需通过向量索引技术优化:
- 核心目标:在 "检索精度" 和 "检索速度" 间找平衡,实现 "近似但高效" 的相似性检索(Approximate Nearest Neighbor, ANN)。
- 主流索引方案 :
- 聚类索引(如 K-Means):先将所有向量聚类成多个 "簇",检索时先找到查询向量所属的簇,再在簇内计算相似度,减少计算范围。
- 树结构索引(如 KD-Tree、Ball-Tree):通过递归划分高维空间构建树,检索时沿树路径快速定位可能的相似向量,适合低维数据(高维下易 "维度灾难")。
- 哈希索引(如 LSH):将高维向量映射到低维 "哈希桶",相似向量大概率落在同一桶中,通过桶内检索提速,适合对精度要求不极致、追求超高速的场景。
- 图结构索引(如 FAISS-IVF、HNSW):将向量构建成图(节点 = 向量,边 = 相似关系),检索时从起始节点出发,沿相似边迭代找到最相似向量,兼顾精度与速度,是当前主流方案(如 FAISS、Milvus 等工具的核心)。
4. 典型应用场景
- 文本领域:语义搜索(如 "苹果手机" 与 "iPhone" 向量相似,可召回相关结果)、问答系统(匹配用户问题与历史答案向量)。
- 图像领域:以图搜图(如上传一张猫的图片,检索相似猫图)、面部识别(人脸特征向量的相似匹配)。
- 推荐系统:用户兴趣向量与商品向量的相似匹配(如用户喜欢 "科幻电影",推荐向量相似的其他科幻片)。
- 医疗领域:如你提供的 "面部表型相关罕见病检索",可将患者面部特征转化为向量,与已知病例向量比对,辅助疾病诊断。
】
三、创新点
- 构建首个面部表型领域知识图谱(FPKG):基于人类表型本体(HPO),整合面部表型、基因、疾病、变异、患者、文献 6 类实体(共 6143 个节点)及 7 类关联关系(共 19282 条边),填补该领域结构化知识的空白。
- 提出两种图检索增强(Graph RAG)方案 :
- Cypher RAG :利用 LLMs 生成图数据库查询语言(Cypher),精准检索 KG 中的相关子图,适用于结构明确的查询;
- Vector RAG :结合生物医学命名实体识别(NER)与图嵌入算法(GLEE 最优),通过向量相似性检索模糊关联信息,解决 LLMs Cypher 生成能力弱的问题。
- 建立四维度评估框架:首次从 "领域知识问答、诊断测试、一致性评估、温度敏感性分析" 四个维度,系统评估 RAG-LLMs 在罕见遗传病领域的性能,覆盖准确性、稳定性、鲁棒性等核心需求。
四、研究贡献
- 知识图谱层面:构建 FPKG,包含 6143 个节点和 19282 条边,为面部表型相关罕见遗传病提供结构化、可动态更新的知识底座(支持纳入最新研究成果,无需重新训练 LLMs)。
- 数据集层面 :创建 3 个基准数据集(共 500 个问题),涵盖 "面部表型 - 疾病 / 基因关联查询、同义词查询、临床诊断案例",标准化该领域 LLMs 评估流程,数据集已开源(https://github.com/zhelishisongjie/Facial-Phenotype-RAG)。
- 方法层面 :验证 Graph RAG 对 LLMs 的优化效果 ------ 相比传统 LLMs,RAG-LLMs 在诊断准确率(平均提升 11.10%-33.10%)、回复一致性(GPT-4o+Cypher RAG 达 99.0%)、温度诱导变异性(降低 53.94%)上均有显著提升。
- 实践层面 :明确不同 RAG 方案的适用场景 ------Cypher RAG 适合结构简单的精准查询,Vector RAG 在模糊查询、KG 未覆盖实体(如 GMDB 私有数据)场景中更灵活,为临床应用提供选择依据。
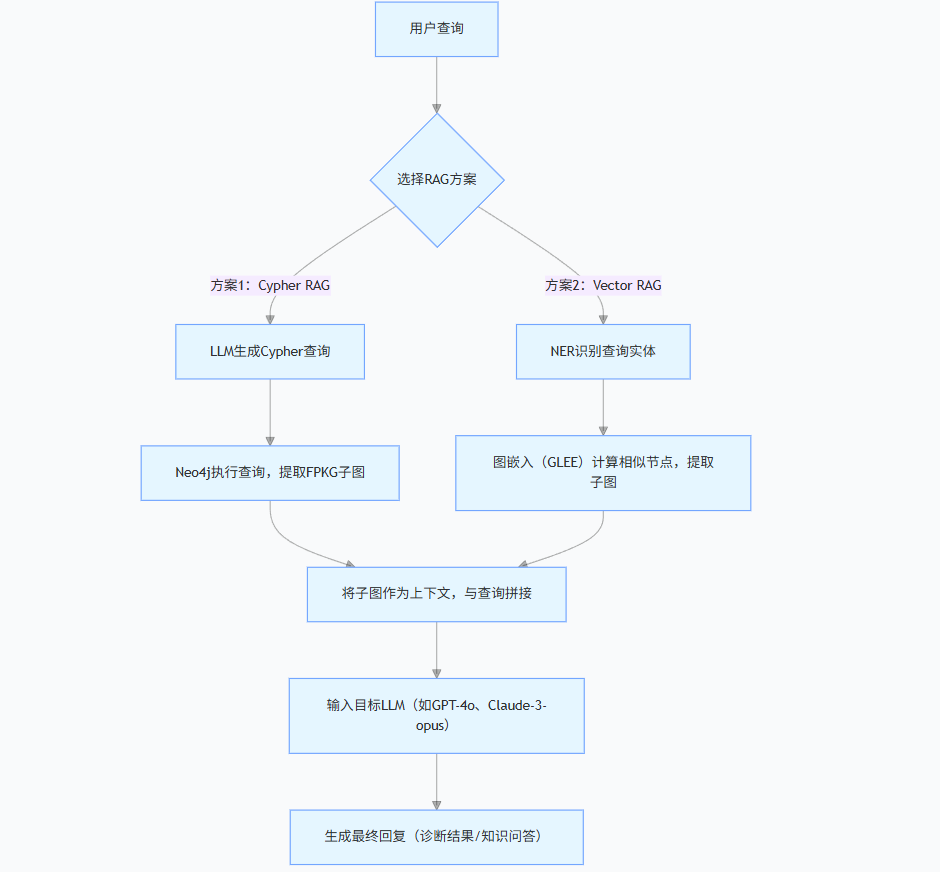
五、提出的方法
论文核心方法为 "FPKG 构建 + 双 Graph RAG + 四维度评估",具体流程如下:
1. 面部表型知识图谱(FPKG)构建
-
数据来源:从 PubMed 检索 25568 篇文献,经去重、筛选(排除综述、动物实验等)后保留 509 篇核心文献,结合 GestaltMatcher Database(GMDB)的患者数据。
-
实体与关系定义 :
实体类型 包含内容(示例) 关系类型 关联逻辑(示例) 面部表型 下斜睑裂、厚下唇、扁鼻梁等 Affect 基因→影响→面部表型(ARID1B→厚下唇) 基因 FAM20C7、KDM6A、CHD7 等 Cause 基因变异→导致→疾病(CHD7 变异→CHARGE 综合征) 疾病 Crouzon 综合征、Coffin-Siris 综合征 Exhibit 疾病→表现出→面部表型(Crouzon 综合征→眼窝浅) 患者 年龄、性别、基因型等 demographic 数据 From 患者数据→来源于→文献 / GMDB 文献 / 变异 文献 ID、基因突变位点(如 c.3479G>T) Have/with_FP 患者→具有→面部表型 -
存储与可视化:使用 Neo4j 图数据库存储结构化数据,通过 GraphXR 可视化图谱,便于直观理解实体关联。
2. 双 Graph RAG 实现
(1)Cypher RAG 流程
- 接收用户查询(如 "Crouzon 综合征相关面部表型有哪些?");
- 调用 LLMs 生成 Cypher 查询语句(如MATCH (d:Disease {name:"Crouzon综合征"})-[:Exhibit]->(fp:FacialPhenotype) RETURN fp);
- 执行 Cypher 查询,从 FPKG 中提取相关子图作为上下文;
- 将上下文与用户查询结合,输入 LLMs 生成最终回复。
(2)Vector RAG 流程
- 用自定义 NER 模型(基于 Bert-base 训练,覆盖 "患者信息、基因变异、面部表型" 三类实体)识别查询中的实体(如 "基因 FAM20C7");
- 将识别的实体作为新节点加入 FPKG,使用图嵌入算法(GLEE 最优,经节点分类任务验证 F1 最高)将图谱转化为向量表示;
- 计算查询向量与 KG 节点的余弦相似度,取 Top-K 相似节点及邻居构成子图作为上下文;
- 结合上下文与查询,输入 LLMs 生成回复。
3. 四维度评估方法
- 领域知识问答 :评估 LLMs 对 "面部表型 - 疾病 / 基因关联、同义词" 的理解,采用 BertScore(语义相似度)和 Coverage(回复覆盖参考答案的比例)作为指标,参考答案基于 HPO 制定。
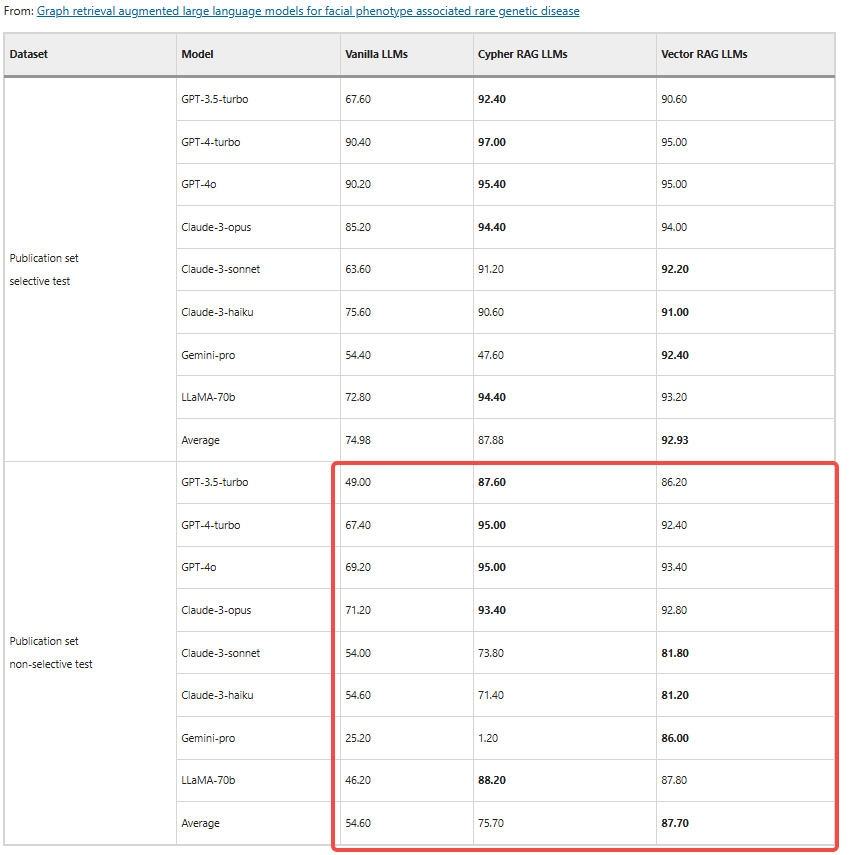
- 诊断测试 :使用 "Publication 集(文献案例)" 和 "GMDB 集(私有临床数据)",分 "选择性测试(提供选项)" 和 "非选择性测试(自主生成答案)",计算 5 次重复查询的平均准确率。
- 一致性评估:对同一问题重复查询 5 次,定义 "一致性 = 最频繁回复出现次数 / 5",衡量 LLMs 输出的稳定性。
- 温度分析:设置温度参数 T=[0.0,0.2,0.4,0.6,0.8,1.0],计算不同温度下诊断准确率的标准差(σ),评估 RAG 对 LLMs 随机性的抑制效果。
六、评估指标
论文针对不同实验任务设计了多维度量化指标,具体如下:
| 评估维度 | 核心指标 | 计算逻辑与意义 | 关键结果(示例) |
|---|---|---|---|
| 领域知识问答 | BertScore | 基于 BERT 计算 LLM 回复与参考答案的语义相似度(0-1),越高越准确 | Vector RAG-LLMs 平均 BertScore 比 Vanilla LLMs 高 0.23 |
| Coverage | (LLM 回复中匹配参考答案的要点数 / 参考答案总要点数)×100%,衡量信息完整性 | 面部表型 - 疾病关联查询中,RAG-LLMs Coverage 比 Vanilla LLMs 高 25.32% | |
| 诊断测试 | 平均诊断准确率 | 5 次重复查询中正确诊断的次数 / 5×100%,衡量诊断能力 | Vector RAG 在 GMDB 非选择性测试中准确率提升 21.83% |
| 一致性评估 | 回复一致性 | (最频繁回复出现次数 / 5)×100%,衡量稳定性 | Cypher RAG-GPT-4o 在 Publication 集选择性测试中达 99.0% |
| 温度分析 | 准确率标准差(σ) | 不同温度下准确率的波动程度,σ 越小越稳定 | RAG-LLMs 平均 σ 比 Vanilla LLMs 降低 53.94% |
| RAG σ Reduction | (Vanilla σ - RAG σ)/Vanilla σ ×100%,衡量变异性降低幅度 | GPT-4-turbo+Vector RAG 的 σ Reduction 达 74.29% |
七、模型结构
论文未提出新的 LLMs 架构,而是基于现有 8 种主流 LLMs(GPT-3.5-turbo、GPT-4-turbo、GPT-4o、Claude-3-opus/sonnet/haiku、Gemini-pro、LLaMA-70b),通过 "Graph RAG 插件" 优化输入上下文,核心结构如下:
关键组件说明:
- NER 模型:基于 Bert-base 训练,训练数据包含 "患者信息、基因变异、面部表型" 三类实体,验证 F1 值达 89.7%( Supplementary Table 3 ),权重已开源至 Hugging Face。
- 图嵌入算法:对比 Node2Vec、DeepWalk、GLEE 等 6 种算法,通过 "节点分类任务"(以实体类型为标签,随机森林预测)验证 ------GLEE 的 F1 值最高(87.2%),故作为 Vector RAG 的默认嵌入方案。
八、研究结论
- Graph RAG 显著优化 LLMs 性能:相比传统 LLMs,RAG-LLMs 在面部表型相关罕见遗传病的 "准确性、一致性、稳定性" 上均有突破 ------ 诊断准确率平均提升 11.10%-33.10%,回复一致性最高达 99.0%,温度诱导变异性降低 53.94%。
- 不同 RAG 方案各有优劣 :
- Cypher RAG:在结构简单的精准查询中表现更优(如 "某基因关联的疾病"),但依赖 LLMs 的 Cypher 生成能力(Gemini-pro 因生成错误 Cypher 导致准确率下降);
- Vector RAG:在模糊查询、KG 未覆盖实体(如 GMDB 私有数据)场景中更鲁棒,平均诊断准确率比 Cypher RAG 高 7.53%-15.33%(GMDB 集)。
- LLMs 选型建议:GPT-4-turbo、GPT-4o、Claude-3-opus 在该领域表现最优,适合作为临床辅助工具;Gemini-pro 因 Cypher 生成能力弱,需优先选择 Vector RAG 方案。
- FPKG 的价值验证:FPKG 的动态更新特性可保持知识时效性,且即使 KG 未完全覆盖领域知识(如部分罕见变异),RAG-LLMs 仍可通过 "排除法推理"(如排除 CHD7 相关疾病,缩小诊断范围)提升诊断价值。
九、剩余挑战
- Graph RAG 的局限性 :
- Cypher RAG 依赖 LLMs 的 Cypher 生成能力,对低能力 LLMs(如 Gemini-pro)适配性差;
- Vector RAG 虽灵活,但无法像 Cypher RAG 那样精准捕捉实体间的结构化关系(如 "基因 - 变异 - 疾病" 的多跳关联)。
- FPKG 覆盖度不足:当前 FPKG 仅包含 509 篇文献和部分 GMDB 数据,未覆盖全部面部表型与罕见遗传病(如部分新发变异、地区性罕见病),导致 RAG-LLMs 在部分查询中仍需依赖 LLMs 预训练知识,可能产生幻觉。
- 非选择性测试性能短板:所有 LLMs 在非选择性测试(自主生成答案)中的准确率均显著低于选择性测试(提供选项)------Vanilla LLMs 准确率下降 20.38%-29.58%,RAG-LLMs 下降 5.23%-26.65%,表明 LLMs 自主推理能力仍需提升。
- 私有数据适配性:GMDB 包含大量未纳入 LLMs 训练集的私有数据,导致所有 LLMs 在 GMDB 集的准确率均低于 Publication 集,说明 RAG-LLMs 对私有 / 稀缺数据的适配性仍需优化。
十、未来工作
- 扩展 FPKG 规模与维度:纳入更多文献(如非英文文献)、临床数据库(如 OMIM、Orphanet),补充 "治疗方案、预后信息" 等实体,构建更完整的 "面部表型 - 基因 - 疾病 - 治疗" 知识网络。
- 优化检索机制 :探索 "Cypher RAG+Vector RAG" 混合方案,结合两者优势(精准性 + 灵活性);研究多跳检索算法,提升对 "基因 - 变异 - 疾病 - 表型" 复杂关联的捕捉能力。
- 拓展应用场景 :
- 临床端:开发医生辅助工具,支持实时查询 "罕见病面部表型特征",并作为医学教育素材(提升医护人员对罕见病的认知);
- 患者端:提供标准化的疾病科普回复,帮助患者及家属理解病情;
- 科研端:辅助挖掘 "未被发现的面部表型 - 基因关联",加速罕见病机制研究。
- 提升 LLMs 自主推理能力 :结合 "思维链(CoT)" 技术,优化 RAG-LLMs 在非选择性测试中的表现,减少对 "选项提示" 的依赖。
- 隐私保护与数据共享:探索联邦学习等技术,在保护 GMDB 等私有数据隐私的前提下,扩大 FPKG 的数据来源,提升模型泛化能力。
我好奇的问题
1.模型在生成式回答的表现,用到的数据集
The diagnostic test used two datasets. The Publication set and the GMDB set. Each dataset is divided into selective and non-selective subsets.
Publication set non-selective test:
BertScore评估LLM答案与参考答案之间的语义相似度
数据集在这。101条,我无语了,看在他是面部罕见病的份上原谅他吧

2.它的知识图谱检索怎么做的(初步给我的印象是检索方法很粗糙)。
Cypher 的 RAG 利用 LLM 生成结构化图查询的能力,通过 Neo4j 的 Cypher 查询实现对相关子图的精确检索。相比之下,基于向量的 RAG 采用生物医学命名实体识别模型和图嵌入算法进行基于相似性的检索,展现出对 Cypher 生成能力有限的 LLM 的更强鲁棒性。
1.,用大模型生成查询的代码
2.先提取查询的实体,如何把实体的节点嵌入在知识图谱中映射,把所有结点的邻居节点查出来获得子图,如何作为上下文提示。
(首先节点的邻居是几跳无法得知,各个查询节点是否联通也不知道,查询出来的节点和关系很多怎么取舍也没说)
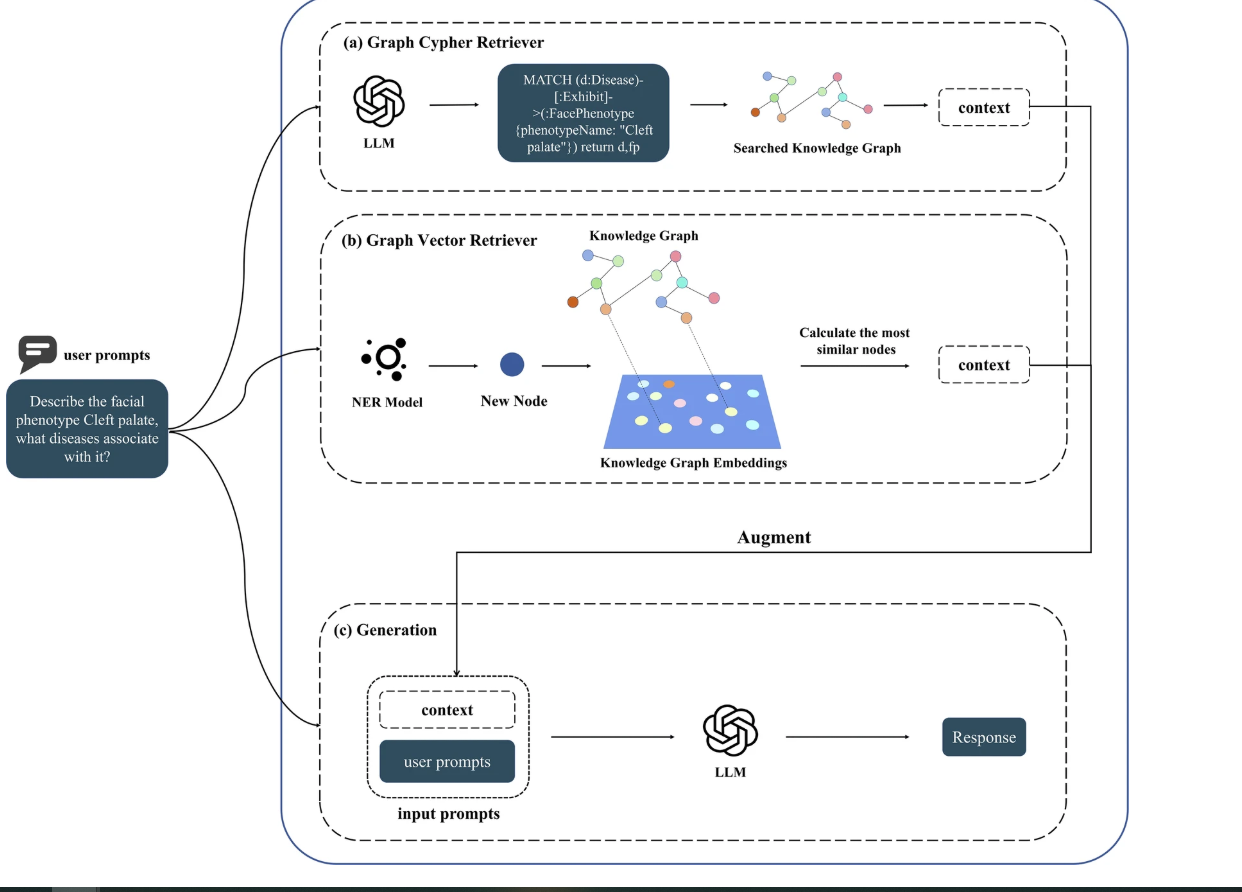
图密码检索器。LLM 生成密码查询,从知识图谱中检索相关信息作为上下文。b . 图向量检索器。训练良好的命名实体识别 (NER) 模型将查询中的实体识别为节点,添加到知识图谱中,并以向量表示嵌入。最后,使用余弦相似度最高的 K 个节点及其邻居作为子图来构建上下文。c **.**生成。将检索到的上下文连同用户提示一起输入 LLM 以获得最终响应。
3.目前的RAG方法和效果如何
然而,目前的RAG依赖于密集向量相似性 搜索作为检索机制。这种方法不足以处理复杂的查询,并且当从多个来源检索相似信息时,还可能遇到冗余问题,导致重复响应<sup> 18,19 </sup>。<sup> 18,19 </sup>。
他们的解决方法是:构造领域知识图谱
4..怎么融合进LLM
看起来是直接搞了提示词

在这里我分析了一下他两个检索的源代码,先看向量检索,方法是:用 LLM 从病例问句抽取实体 → 把新病例挂到一个已有知识图谱上 → 用图嵌入找相似节点 → 取相似节点的邻域子图做 RAG 上下文 → 再让 LLM 输出诊断答案
实体抽取直接明确定义了要哪些实体,这就是只做某种领域疾病的好处,全科关注的实体是不同的
context = [
"{表型} -> {疾病}",
"{疾病} -> {基因}",
"{表型} -> {疾病}",
"{变异} -> {基因}"
]
You are a medical genetics assistant specializing in analyzing facial phenotypes
to identify rare genetic diseases.
Instructions:
The information section provides some knowledge based on the patient's symptoms
and genetic data. You should refer to this knowledge and make the most likely diagnosis.
Information: {context}
Question: {question}
from langchain.chat_models import ChatOpenAI
import os
import openai
import pandas as pd
from tqdm import tqdm
import time
from openai import OpenAI
import pandas as pd
from sklearn.metrics.pairwise import cosine_similarity
from karateclub import NodeSketch, GLEE
import networkx as nx
import matplotlib.pyplot as plt
import json
import numpy as np
# question = "There is a 19 years old female German patient with a facial phenotype of Wide nasal bridge, Broad philtrum. What disease might this patient have?"
# FRONTONASAL DYSPLASIA 1(FND1)
questions = pd.read_csv("./questions/GMDB_patient_question.csv")
questions = questions['Patient_question']
# ==================== QA template ====================
Extraction_TEMPLATE="""
You are a professional biomedical information extraction assistant. Please accurately identify and extract the following entity information from the given sentence:
- age
- gender
- race
- mutation_gene
- mutation_hgvs (mutation description in HGVS format)
- facial_phenotypes
- disease
Requirements:
1. Output MUST be a SINGLE VALID JSON object.
2. DO NOT wrap the output in ```json ``` or any other Markdown.
3. If a field is not found, set it to `null`.
4. Never add comments or text outside the JSON.
Input sentence: {question}
Example output format:
{{
"age": "extracted age information",
"gender": "extracted gender information",
"race": "extracted race information",
"mutation_gene": "extracted gene name",
"mutation_hgvs": "extracted HGVS mutation description",
"facial_phenotypes": ["facial feature 1", "facial feature 2"],
"disease": ["disease 1", "disease 2"]
}}
Usage example:
Input sentence: "There is a 12.0-year-and-0.0-month-old female European patient with a mutation in NFIX, namely, NM_002501.3:c.59T>C, p.(Leu20Pro) and with facial phenotypes of Tall stature, Narrow mouth, Open mouth, Everted lower lip vermilion, Long face, Mandibular prognathia, Triangular face, Anteverted nares, Strabismus, Deeply set eye, Downslanted palpebral fissures, Atypical behavior, Intellectual disability, Hypotonia, Global developmental delay, Motor delay, obsolete Joint laxity, Ventriculomegaly, Chiari malformation, Prominent forehead. What disease might this patient have? Choose from the following options: A.SOTOS SYNDROME; SOTOS B.LATERAL MENINGOCELE SYNDROME; LMNS C.Oculocutaneous albinism D.SPASTIC PARAPLEGIA 50, AUTOSOMAL RECESSIVE; SPG50"
Output:
{{
"age": "12.0",
"gender": "female",
"race": "European",
"mutation_gene": "NFIX",
"mutation_hgvs": "NM_002501.3:c.59T>C, p.(Leu20Pro)",
"facial_phenotypes": ["Tall stature", "Narrow mouth", "Open mouth", "Everted lower lip vermilion", "Long face", "Mandibular prognathia", "Triangular face", "Anteverted nares", "Strabismus", "Deeply set eye", "Downslanted palpebral fissures", "Atypical behavior", "Intellectual disability", "Hypotonia", "Global developmental delay", "Motor delay", "obsolete Joint laxity", "Ventriculomegaly", "Chiari malformation", "Prominent forehead"],
"disease": ["SOTOS SYNDROME; SOTOS", "LATERAL MENINGOCELE SYNDROME; LMNS", "Oculocutaneous albinism", "SPASTIC PARAPLEGIA 50", "AUTOSOMAL RECESSIVE; SPG50"]
}}
"""
# LLMs
LLM_MODEL = 'gpt-4o'
# api key
os.environ["OPENAI_API_BASE"] = "https://api.openai.com/v1/"
os.environ["OPENAI_API_KEY"] = "your api key"
API_KEY = os.environ.get('API_KEY')
API_VERSION = os.environ.get('API_VERSION')
API_BASE = os.environ.get('OPENAI_API_BASE')
openai.api_type = "azure"
openai.api_key = API_KEY
openai.api_base = API_BASE
openai.api_version = API_VERSION
# ==================== LLM ====================
temperature = 0.1
chat_model = ChatOpenAI(model_name = LLM_MODEL, temperature = temperature)
questions = pd.read_csv("./questions/GMDB_patient_question.csv")
questions = questions['Patient_question']
import json
import re
# entities
with open('records_25.05.08.json', encoding='utf-8-sig') as f:
graph_data = json.load(f)
client = OpenAI(
base_url=os.environ.get('OPENAI_API_BASE'),
api_key=os.environ.get("OPENAI_API_KEY"),
)
def filter_fields(node_data):
target_fields = ['labels', 'phenotypeName',
'with_disease', 'sid',
'geneName',
'details', 'variation_gene_name',
'dname']
filtered = {}
for field in target_fields:
if field in node_data:
filtered[field] = node_data[field]
elif 'properties' in node_data and field in node_data['properties']:
filtered[field] = node_data['properties'][field]
return filtered
from fuzzywuzzy import fuzz
def match(str1, str2):
str1 = str1.replace(" ", "")
str2 = str2.replace(" ", "")
match_percentage = fuzz.ratio(str1, str2)
if match_percentage > 80:
return True
else:
return False
def trim_strings_to_max_length(string_list, max_length):
total_length = sum(len(s) for s in string_list)
if total_length > max_length:
excess_length = total_length - max_length
for i in range(len(string_list) - 1, -1, -1):
current_string_length = len(string_list[i])
if current_string_length > excess_length:
string_list[i] = string_list[i][:current_string_length - excess_length]
break
else:
string_list[i] = ""
excess_length -= current_string_length
return string_list
context_list = []
for k in tqdm(range(len(questions))):
question = questions[k]
print(question)
# construct graph
G = nx.Graph()
for i in range(0, len(graph_data)):
info = graph_data[i]['p']
start_node = info['start']
end_node = info['end']
G.add_node(start_node['identity'], labels=start_node['labels'], properties=start_node['properties'])
G.add_node(end_node['identity'], labels=end_node['labels'], properties=end_node['properties'])
relation = info['segments'][0]['relationship']
# G.add_edge (relation['start'], relation['end'], type = relation['type'],properties = relation['properties'])
G.add_edge(relation['start'], relation['end'], type=relation['type'], properties=relation['properties'],
key=relation['identity'])
def graph_sort(G):
index_map = {}
index = 0
for node in G.nodes():
index_map[str(node)] = index
index += 1
# print(index_map)
mapping = {node: i for i, node in enumerate(G.nodes())}
G = nx.relabel_nodes(G, mapping)
# print(G.nodes)
# print(f" lenth: {len(G.nodes())} last node:{ G.nodes[len(G.nodes())-1] }")
return G, index_map
G, index_map = graph_sort(G)
# entity extraction
data = {"question": question}
content = Extraction_TEMPLATE.format(**data)
# Employing an LLM for entity extraction may lead to better results.
max_retries = 5
retries = 0
while retries < max_retries:
try:
chat_completion = client.chat.completions.create(
messages=[
{
"role": "user",
"content": content,
}
],
model=LLM_MODEL,
)
response = chat_completion.choices[0].message.content
entity = json.loads(response)
break
except Exception as e:
retries += 1
print(f"An error occurred: {e}")
print(f"retry:{retries}...")
age = entity.get('age')
gender = entity.get('gender')
race = entity.get('race')
mutation_gene = entity.get('mutation_gene')
mutation_detail = entity.get('mutation_hgvs')
facial_phenotypes = entity.get('facial_phenotypes')
disease = entity.get('disease')
# Add node to KG
new_node_id = len(G.nodes)
G.add_node(len(G.nodes), labels=['New nodes'], properties={})
connection_rules = [
('Variation', 'details', mutation_detail or []),
('FacePhenotype', 'phenotypeName', facial_phenotypes or []),
('Genotype', 'geneName', mutation_gene or []),
('Disease', 'dname', disease or [])
]
for n in G.nodes:
node_labels = G.nodes[n].get('labels', [])
if not node_labels:
continue
node_props = G.nodes[n].get('properties', {})
for label, prop_key, match_value in connection_rules:
if node_labels[0] == label:
prop_value = node_props.get(prop_key, '')
if any(match(prop_value, p) for p in match_value):
G.add_edge(n, new_node_id, type='Connected')
# print(f"Connected {prop_value} to new node")
# Embedding
model_GLEE = GLEE(seed=100)
model_GLEE.fit(G)
embeddings = model_GLEE.get_embedding()
new_node_vector = embeddings[new_node_id]
embeddings = embeddings[:-1, :]
similarities = cosine_similarity(new_node_vector.reshape(1, -1), embeddings)
most_similar_idx = similarities[0].argsort()[::-1]
neo4j_index = [list(index_map.keys())[idx] for idx in most_similar_idx]
top_similar_nodes = most_similar_idx[0:5]
# for topnode in top_similar_nodes:
# print(similarities[0][topnode], G.nodes[topnode])
# subgraph of top similar nodes
context = []
for topnode in top_similar_nodes:
neighbors = list(G.neighbors(topnode))[:-1]
for neighbor in neighbors:
edge_data = G.get_edge_data(topnode, neighbor)
subgraph = str(filter_fields(G.nodes[topnode])) + " " + edge_data['type'] + " " + str(
filter_fields(G.nodes[neighbor]))
# print(f"***********Retrieved subgraph: {subgraph}")
context.append(subgraph)
context = trim_strings_to_max_length(context, 10000)
print(context)
context_list.append(context)
# ==================== QA template ====================
QA_TEMPLATE="""
You are a medical genetics assistant specializing in analyzing facial phenotypes to identify rare genetic diseases, and interpret the relationships between genes, facial features, and associated diseases.
Instructions:
The information section provides some knowledge based on the patient's symptoms and genetic data. You should refer to this knowledge and make the most likely diagnosis.
Make the answer sound as a response to the question.
Do not mention that you got this result based on the information provided, but ensure the explanation is medically sound and justifiable.
If the information provided is empty, answer the question normally using medical reasoning based on typical symptoms and known genetic associations.
Information: {context}
Question: {question}
"""
# LLMs
#LLM_MODEL = 'gpt-3.5-turbo'
#LLM_MODEL = 'gpt-4-turbo-2024-04-09'
LLM_MODEL = 'gpt-4o'
#LLM_MODEL = 'claude-3-opus-20240229'
#LLM_MODEL = 'claude-3-sonnet-20240229'
#LLM_MODEL = 'claude-3-haiku-20240307'
#LLM_MODEL = 'gemini-1.0-pro-latest'
filename = "Vector-" + LLM_MODEL + "-1"
API_KEY = os.environ.get('API_KEY')
API_VERSION = os.environ.get('API_VERSION')
API_BASE = os.environ.get('OPENAI_API_BASE')
openai.api_type = "azure"
openai.api_key = API_KEY
openai.api_base = API_BASE
openai.api_version = API_VERSION
# ==================== LLM ====================
temperature = 0.1
chat_model = ChatOpenAI(model_name = LLM_MODEL, temperature = temperature)
questions = pd.read_csv("./questions/GMDB_patient_question_withoutchoices.csv")
questions = questions['Patient_question']
# ============================================================ openai ============================================================
client = OpenAI(
base_url=os.environ.get('OPENAI_API_BASE'),
api_key=os.environ.get("OPENAI_API_KEY"),
)
max_retries = 5
response_rag_list = []
for i in tqdm(range(len(response_rag_list), len(questions))):
if LLM_MODEL.startswith('claude'):
time.sleep(12)
time.sleep(1)
question = questions[i]
context = context_list[i]
data = {
"context": context,
"question": question
}
content = QA_TEMPLATE.format(**data)
retries = 0
while retries < max_retries:
try:
chat_completion = client.chat.completions.create(
messages=[
{
"role": "user",
"content": content,
}
],
model=LLM_MODEL,
)
response = chat_completion.choices[0].message.content
break
except Exception as e:
retries += 1
print(f"An error occurred: {e}")
print(f"retry:{retries}...")
# print(response)
response_rag_list.append(response)
# ============================================================ openai ============================================================