论文题目:Machine Learning-Guided Protein Engineering
基于决策变量分类的动态多目标优化算法(Petr Kouba,# Pavel Kohout,# Faraneh Haddadi,# Anton Bushuiev, Raman Samusevich, Jiri Sedlar, Jiri Damborsky, Tomas Pluskal,* Josef Sivic,* and Stanislav Mazurenko*)
个人总结:
机器学习可以解决蛋白质工程的 实验慢 成本高 筛选难度大的问题,首先就是使用机器学习的模型 从大量的数据里面找规律筛选出合适的蛋白质,所以前提是需要把蛋白质变成计算机能理解的数据格式。
现阶段主要存在的问题:高质量的数据少,大多数的模型是黑箱模式,不知道为什么这么预测。
PLM(蛋白质语言模型,处理酶的序列),MSA(多序列对比,把同源的蛋白质序列对齐,找保守位点。)
监督学习用标注数据训练,无监督学习用海量未标注的序列找规律,主动学习筛选有价值的样本做实验。
总目标是去找高活性高稳定的酶突变体,但是酶的整个序列空间很大,所以采用计算机来模拟。先用机器学习的模型做预判,预判完了以后采用进化算法进行迭代,通过迭代出的好解再做实验验证。
白箱优化:使用ML模型去学习他的规律,就是知道怎么进化能有更好的分数,
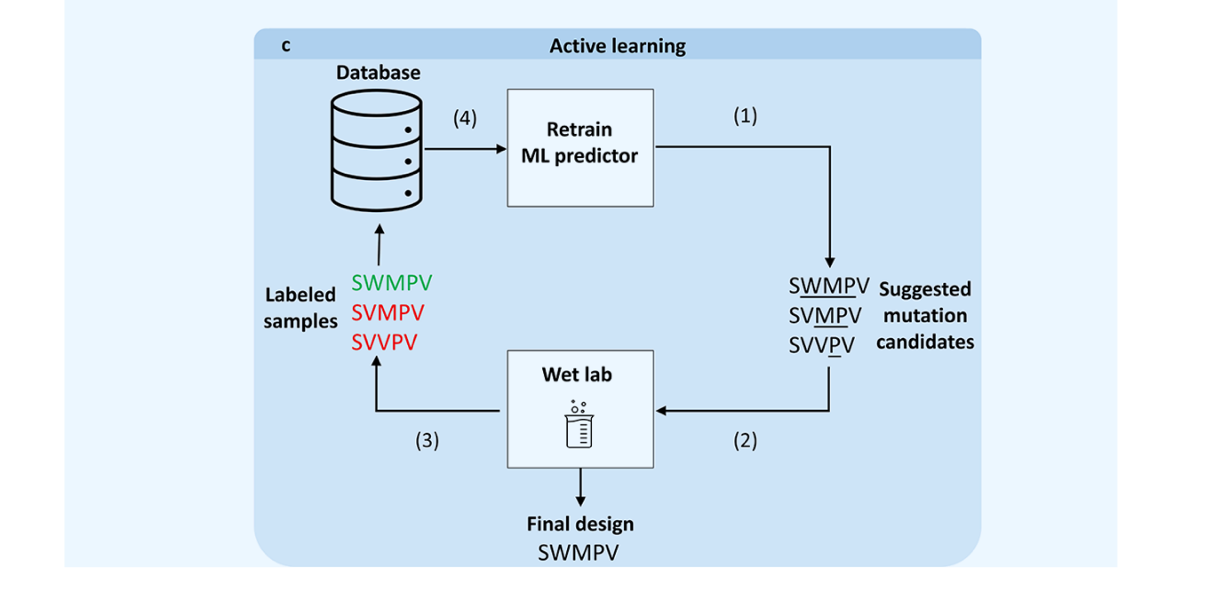
第三个就是主动学习的循环:首先使用黑盒或白盒得到一批预测解,然后做实验进行更新真是数据,得到真实数据以后再放回原始数据里面进行进一步的迭代提高预测的准确率,进行一个正向循环。
虽然计算机模拟优化方法支持迭代生成有前景设计,但它们完全依赖预测变量正确评分突变景观中任一点的能力,这可能是个不切实际的强假设
摘要
- 近年来,在极具前景的生物催化剂工程上,机器学习方法的推进越来越多。这些方法利用现有的实验和模拟数据,帮助发现和注释有潜力的酶,并提出有益突变以改进已知靶点。
- 蛋白质工程机器学习领域正逐渐获得动力,这得益于近期的成功案例以及其他领域的显著进展。它已经涵盖了诸如理解和预测蛋白质结构与功能、催化效率、对镜选择性、蛋白质动力学、稳定性、溶解度、聚集等雄心勃勃的任务。尽管如此,该领域仍在不断发展,面临许多挑战和需要解决的问题。
- 在本视角中,我们概述了该领域的持续趋势,重点介绍了近期案例研究,并探讨基于机器学习的方法目前的局限性。我们强调在使用比率前对新兴模型进行彻底实验验证的重要性
现状
生物催化是一个充满前景的领域,为各行业创造可持续且环保的解决方案提供了多样可能性。其潜力源于利用经过数百万年进化的细胞和酶,模拟并利用自然力量,高效执行特定化学反应。这使得能够选择性且高效地转化化合物成为可能,为传统化学催化提供了一种替代方案,传统催化往往需要恶劣的条件和有毒化学品。因此,生物催化剂在精细化学品、药品和食品成分的生产以及能源和材料生产的可持续工艺开发中具有价值。此外, 生物催化是一个令人振奋的研究与开发领域,未来充满希望,因为它有可能通过提供绿色替代传统化学工艺、新能源以及提升工业流程整体效率或生物去除难废物的工具,开启应对多样挑战的新方案。它也是一个高度跨学科的研究领域,大量运用先进的实验技术,计算方法。
许多研究领域正逐步从几乎完全依赖实验工作,向融合计算模拟和数据驱动方法的混合方法转变。过去,研究人员会积累单个实验的观测数据,并利用所得数据制定基本规则。随后,他们基于这些规则创建了模拟,以更好地理解正在调查的系统。随着计算能力的提升,研究人员能够转向依赖机器学习(ML,表1中加粗术语的术语表)算法的数据驱动方法,直接从数据中推导规则。这一转变使得高效且全面地分析通常由高通量技术生成的大型复杂数据集成为可能。特别是,非常强大的深度学习算法在生命科学领域获得了广泛的应用,本文将在该视角中详细讨论。虽然实验科学和计算模拟依然发挥着重要作用,但随着技术和数据收集方法的进一步发展,数据驱动方法的趋势很可能会持续。
这一范式转变体现在描述蛋白质工程中的机器学习(见图1)。数据驱动方法的趋势预计将继续,因为技术进步使我们能够更有效地积累、存储和再利用生物和生化数据。这一目标得益于诸如FAIR原则等倡议,该原则促进数据的可查找性、可访问性、互作性和可复用性,以及欧洲开放科学云,旨在推广数据处理的最佳实践。这些大规模倡议预计将加速数据驱动方法的采用,使研究人员更容易访问, 使用并共享现有数据,并确保这些数据质量高。
本文重点
本观点聚焦于机器学习在蛋白质工程中的应用,即通过分子生物学技术优化生物催化剂的序列和三级结构,提升其性能。在时间上,我们将主要涵盖自2019年发表的同主题综述以来的时期。至于具体领域,我们将主要关注通过突变已知蛋白质来实现工程化的应用,而非全新设计蛋白质。对于对新生设计特别感兴趣的读者,本观点也可能有所帮助,因为我们涵盖了多种蛋白质设计任务中常见的技术,但关于新生设计的具体细节,如深度生成建模,请参阅其他综述。 我们还将介绍机器学习的高层概念,帮助读者熟悉更广泛的背景,不会深入探讨各种神经的具体技术细节网络架构。我们推荐读者参考几篇关于这些主题的最新优秀评测。 我们将从用户视角考虑新方法。我们认为这一点很重要,因为研究论文中提出的方法虽然令人兴奋且富有创新性,但如果更广泛的社区无法快速轻松地采纳,往往影响有限。此外,我们还会从其他领域汲取灵感,因为我们相信识别不同领域任务之间的相似点可以加速更强大、更实用方法的发展。
机器学习原理
涵盖基于机器学习的管道和术语基础知识,突出蛋白质工程与其他领域的相似之处,概述蛋白质数据与其他机器学习常用数据类型的主要区别,并总结用蛋白质数据进行机器学习的挑战
A.机器学习基础
机器学习基础。机器学习通常被视为人工智能(AI)的一个子类别。其主要目的是直接从可用数据中学习模式,并利用所学模式生成新数据的预测。它与其他建模系统行为的方法(如量子力学计算)的主要区别在于,机器学习不依赖硬编码规则来进行预测 。相反,机器学习模型是依赖于通用参数的数学函数,这些参数的值通过利用可用数据和优化准则(即所谓的损失函数)进行优化获得(学习)。
由于最终模型是从输入数据中推导出来的,仔细的数据收集对机器学习至关重要 。特别是,任何偏差、测量噪声和不平衡都必须被识别和考虑。此外,由于机器学习基于数学函数,数据集中的每个数据点通常都需要用通常称为特征的数向量表示。特征可以通过对原始数据进行简单编码获得,例如端到端学习和单热编码,但它们也可能代表从原始数据中推导出的更复杂的量。例如,在预测蛋白质序列的溶解度时,特征可能是简单的氨基酸计数、不同残基形成二级结构的倾向、蛋白质的守恒评分,或代表聚合的物理化学性质的变量。选择能提供数据中潜在模式相关信息的有用且有辨别性的特征在机器学习中至关重要,因为这些特征是算法唯一能提供的数据特征在训练和基于未来输入进行预测时加以利用。
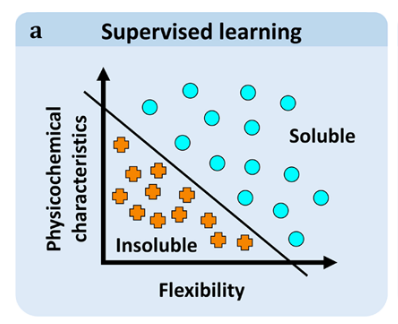
存在几类不同类型的机器学习问题。在监督学习问题中,目标是为每个数据点预测一个特定的属性(称为标签)(图2A)。例如,如果我们试图预测蛋白质的溶解度,每个数据点都可以根据实验结果被标记为"可溶性"或"不溶性"。数据点可以有多个标签,因此一个蛋白质可能有"可溶性"、"来自嗜热生物体"和"球状"等标签。标签可以组成一组类,或落在数值范围内,导致监督学习问题有两个子类型:涉及无固有顺序标签的分类问题(如"可解"或"不可解"),以及涉及数值标签(如蛋白质产率)的回归问题。
相比之下,无监督学习问题的目标是识别未标记数据中的模式 。无监督学习技术包括聚类算法 以及数据压缩或投影方法,如主成分分析(见图2B)
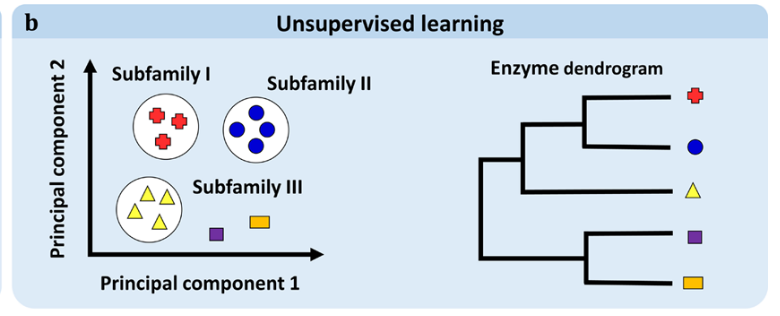
半监督学习问题是指标记数据数量有限但存在大量未标记数据的情况。未标记的数据用于学习数据的一般分布,帮助学习监督模型。例如,所有数据都可以通过无监督算法进行聚类,未标记样本则可以根据集群中的标签自动标记,从而增强了监督学习数据集,尽管标注质量较低,但这对其性能有帮助。
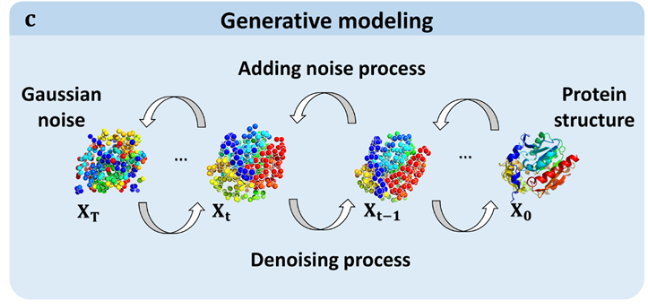
监督机器学习与非监督机器学习的界限因能够合成标签的方法的出现而变得模糊。例如,在数据压缩方法中,标签可能就是输入本身,算法可能会施加约束(例如架构中的瓶颈),迫使模型学习更紧凑的方式来表示数据及其分布。旨在捕捉数据分布以生成新样本的算法属于一类机器学习模型,称为生成模型。该类最新的例子包括扩散模型,近年来被用于生成蛋白质主链结构并预测柔性配体与蛋白质的结合情况(见图2C)。在扩散模型中,合成训练数据通过逐步对真实数据(X0)进行噪声生成,最终获得最大噪声样本(XT)。噪声逐渐增加的数据序列被反转,用于训练模型通过让(噪声较小的)采样 Xt‐1 作为后续(噪声更小的)采样 Xt 的"标签"来进行逐步去噪。有关扩散生成模型及其在生物信息学中的应用,请参见最近的综述。
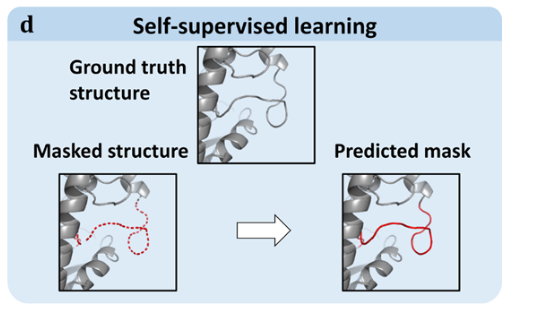
或者,我们可以通过遮蔽输入的一部分,例如蛋白质序列或结构中的残基,,并训练一个预测遮蔽部分的模型来避免标记。换句话说,原始数据(例如被掩蔽的氨基酸)被视为对应数据点的标签。这些方法属于自导学习方法(见图2D),目前因其在大型语言模型中的巨大成功而受到广泛关注;事实证明,这种"自监督"方法使算法能够学习数据的有用特征,比如自然语言模型中的语法和语义。以下章节介绍了自监督学习在酶学领域的一些应用。
监督学习中,欠拟合和过拟合是两个必须始终考虑的重要概念。欠拟合指的是所选模型类别不足以近似可用数据中的模式、正则化过强,或训练过程的参数(如训练时长或学习率)不合适的情况。因此,模型无法捕捉输入与输出之间的关系,且训练误差较高。相反,当模型拥有过多的自由度时,就会发生过拟合,导致其在训练过程中开始在训练数据中拟合噪声。这导致推广力差,模型应用于新输入时性能显著下降。因此,对训练模型进行稳健评估对于机器学习至关重要,以获得训练过程反馈并开发改进的训练方案或模型超参数。
机器学习的最佳实践是将可用数据拆分为三个不相交的子集:训练集、验证集和测试集。模型通过将参数拟合到训练集,学习数据中的潜在模式。验证集在训练的某些阶段提供给模型进行基础评估,这些评估结果用于选择模型的超参数。最后,测试集用于获得模型性能的真实估计,因此仅在训练完成并设定超参数最终值后使用。由于模型在训练过程中未看到测试集,当模型应用于该"新"数据集时,如果测试集准确代表研究数据的一般分布,其性能应与测试集的表现相当。
评估指标的选择取决于当前任务。分类问题主要基于模型准确性评估,即正确预测数与总预测数的比值。回归问题通常基于预测标签与真实值标签之间的差异来评估,因此常用的指标包括这些标签之间的相关性、均方误差(MSE)以及相关指标,如均方根误差(RMSE)。更复杂的问题通常需要定制的度量。例如,在蛋白质结构预测任务中,可以使用Cα原子预测位置与实际位置之间的MSE,这可以用固定坐标系(全局比对)或每个残基的局部坐标(局部比对)来表示。这两个指标都表明预测结构与实际结构的高度匹配程度
B.生物催化中机器学习任务与其他领域的相似之处。
机器学习的一个优势是其通用性,因为用于蛋白质工程任务的算法与其他领域相似。因此,研究蛋白质数据的科学家可以重复利用并基于自然语言处理、计算机视觉和网络分析等其他领域的现有解决方案。
自然语言处理(NLP)是计算机科学的一个领域,旨在教授计算机如何理解和处理自然语言。近年来,新的机器学习技术推动了该领域的重大进展。例如,NLP中的一个常见任务是生成语义和语法正确的句子。在蛋白质工程中,代表一级序列的氨基酸串可以被视为由二十个字母组成的字母表组成的词,这些字母代表典型氨基酸。这些词语可以代表二级结构或其他意象,这些意象可以以有意义的方式组合,形成蛋白质结构语言中对应功能性蛋白质的句子。NLP中的另一个常见任务是为单个词语(例如预测词汇类别或识别相关信息)或短语(例如情感分析)赋予标签。该数据结构类似于带有表示蛋白质稳定性、结合亲和力、特异性或其他特征的注释蛋白数据集。此外,蛋白质序列、结构和功能之间的复杂关系类似于人类语言,促使研究人员将NLP中使用的基于变换器的大型语言模型应用于蛋白质工程任务。
计算机视觉,近年来也因机器学习的进步而受益匪浅,相关技术也在蛋白质结构研究中得到了应用。例如,蛋白质结构可以通过应用离散网格转换为体素(3D像素)阵列。由此产生的表示类似于体积三维图像,使得机器学习架构(如卷积神经网络)能够应用,这些架构最初设计用于处理图像数据。这些网络通过卷积和层级聚合学习表示,近年来被用于预测蛋白质突变景观、 蛋白-配体结合亲和力以及蛋白质与水分子的相互作用。去噪扩散概率模型是另一类应用于蛋白质结构预测的计算机视觉模型。它们被训练去噪现有采样,通过变换随机噪声生成新采样。这带来了图像生成的重大突破,并催生了极为成功的模型,如DALL-E 238或稳定扩散。在蛋白质科学中,这些模型被用于快速实现蛋白质-配体结合,成新的小分子配体和连接子,以及对大型蛋白质进行全新设计。
通过适应视频分析开发的技术,预测蛋白质动态,图像与蛋白质结构之间的相似性还可进一步加以利用。例如,分子动力学模拟中生成的轨迹可以被视为一组三维图像的时间序列。这使得蛋白质动力学分析类似于视频处理,并意味着事件检测视频方法可以应用于分子动力学轨迹,以检测如隧道开启等事件。视频处理技术因此可以调整,通过分析单个原子或原子群在蛋白质结构中的运动,类似于物体或物体群的运动,从而澄清蛋白质的功能。一个视频。此外,视频合成ML技术取得了显著进展,可能启发捕捉和合成蛋白质动力学的新方法。
最后,还有一个与蛋白质工程相关的领域:网络分析,涉及研究相互关联元素的性质和结构。网络分析技术已被成功应用于研究多样化的社会和生物网络,包括对社交网络和蛋白质-蛋白质相互作用网络的新冠相关情感的发展。 蛋白质之间或蛋白质与配体之间的相互作用可以用网络(图)表示,其中节点对应蛋白质和配体,边对应它们之间的生物关系。一旦定义了这样的网络,就可以应用链路预测或群落检测方法。 或者,蛋白质结构也可以表示为一个网络,其中节点对应残基或单个原子,边缘对应残基间相互作用或原子间键。这使得基于图的机器学习算法能够用于预测蛋白质功能、溶解度或毒性等任务。此外,关于蛋白质相互作用组和小分子结构的数据也被用来推动基于图的机器学习理论研究:最成熟的图学习基准标准OGB包含了多个此类生化数据集。
C.机器学习在蛋白质数据中的挑战
如上所述,蛋白质工程任务与其他机器学习领域(包括自然语言处理、计算机视觉和网络分析)之间存在显著相似之处。然而,蛋白质数据也面临着与蛋白质表示、标记数据集构建以及建立稳健训练协议相关的独特挑战。
蛋白质表示的选择是所有与蛋白质相关的计算任务中的关键步骤 。蛋白质可以以不同细节层次表示,从离散且准确的氨基酸序列一维表示 ,到连续且精度较低的三维表示每个原子位置(包括或排除化学键)。所选表示决定了计算模型可用的信息类型和数量,以及适用的模型架构范围。
蛋白质序列的常用表示是用20个氨基酸(字母)字母表构建的字符串(单词)。字符串的长度等于残基数,第n个字符编码蛋白质序列中第n位的氨基酸。 在计算机模拟中,氨基酸通常通过单热编码表示。当可用于训练的数据不足以实现端到端学习时,序列的单热编码可以进一步转换为对应氨基酸特定物理化学特性的值,例如使用AA指数。这些指标为管道提供了额外信息和可解释性,尽管在某些任务中它们的性能与随机向量相当。 另一种富集蛋白质序列表示的策略是通过多序列比对(MSA)而非单一蛋白质序列来包含进化信息。这些进化信息在多种任务中非常有价值,尤其是在结构预测中,因为序列中不同残基位置的协方差可以与残基的空间接近度相关。
表示蛋白质结构的选项更为丰富;表示可能包括所有原子,仅包含部分化学元素(例如,除氢原子外的所有原子),或仅包含残基的关键成分(例如,α碳)。此外,它们可能包含关于这些原子和/或残基的不同类型的信息。理想情况下,为了实现数据高效的模型训练,结构表示应对旋转、平移和反射保持不变。然而,基于残基(原子)三维坐标的简单表示缺乏这一性质。因此,通常使用残基间或原子间距离矩阵来表示蛋白质结构 ,其中每一行和列对应蛋白质的特定残基(或原子),每个基质条目的值等于对应残基(原子)之间的距离。这样的矩阵必然是对称的;因此,通常会将上部(或下部)三角形部分转换为一维矢量进行处理,例如通过神经网络进行处理。虽然该表示在旋转和平移上不变,但本质上也是冗余的,因为其空间复杂度与残基(原子)数量成平方。
基于图的蛋白质表示近年来引起了广泛关注。 图由一组节点通过一组边连接组成。节点通常代表残基、原子或空间上相近的原子群,而边通常对应化学键、节点间的空间接近度(接触点)或两者兼有。基于图的蛋白质表示非常灵活,因为节点和边的定义可以针对特定任务进行定制,并且可以等变于旋转, 平移和反射。对节点和边的方便定义还可以引入归纳偏置,从而提升模型性能。例如,对应化学键的边可以引导模型更快或更少的数据学习化学知识。图神经网络(GNN)架构近年来在多种蛋白质相关任务中达到了最先进的性能,例如DeepFRI和HIGHPPI预测蛋白质功能和蛋白质相互作用的方法。基于图的特殊蛋白质表示,如点云(无边)或完整图(完整边集),对于使用强大的变换器模型进行处理尤其方便。
一种更通用的蛋白质表示方法是通过深度学习模型直接学习该表示,这一方向目前在生物学领域正逐渐兴起。其目标是通过从现有数据中推断表征参数,消除人类做出选择的次优性。此外,通常可以通过自监督训练学习这种表示,即无需注释数据。这些表示可以从序列数据中获得,例如由ESM("进化尺度建模")语言模型实现,也可以从大型结构数据集中获得,例如GearNet。越来越多的模型结合了这两种数据源,例如ESM-GearNet
在应用机器学习于酶工程时,获取适当标记的数据集可能具有挑战性,因为在选择实验数据获取方法时,数据质量与数量之间常常存在权衡。大多数可靠的生化方法使用专门设计的仪器只能提供少量蛋白质变异的数据,因此通常不足以代表性地抽样庞大的突变空间。相反,像深度突变扫描(DMS)这样的高通量方法容易出现数据质量问题,并且在筛选速度明显慢于测序的酶时,会面临吞吐瓶颈。
从多个来源汇总新数据集的过程可能因蛋白质研究惯例的不一致而变得复杂。这些不一致包括各种实验工具的偏差差异、数据归一化时使用的不同分布,以及稳定性、溶解度和酶活性等量的定义不一致。所有这些都可能在构建的数据集中引入错误,例如导致同一蛋白质上出现矛盾标记。蛋白质数据也可能包含设计策略引入的偏差。例如,由于广泛使用的丙氨酸扫描技术,青氨酸在突变数据中往往被过度代表。在构建数据集和解释模型输出时,考虑这些偏差非常重要,因为训练集的组成显著影响模型探索的模式空间。
多年来,收集了大量蛋白质数据。然而,这些数据大多属于专有,学术界无法访问。此外,公开数据集通常以非结构化方式发布,限制了其可用性。虽然像GPT-3,、GPT-4,或BioGPT这样的大型语言模型在大规模总结文本方面非常有效,但从出版物中挖掘相关数据仍然需要大量人力。
一些机器学习软件包,如TorchProtein,为各种蛋白质科学任务提供预处理数据集,使蛋白质研究对其他领域的机器学习专家更为便捷。 其他软件包如PyPEF则为整合更简单的机器学习模型提供了框架,并结合了从AAindex氨基酸物理化学和生化性质数据库中衍生的特殊编码。 尽管取得了这些进展,开发蛋白质数据的机器学习模型仍需一定的生物化学领域知识,以应对机器学习其他应用领域缺乏的蛋白质数据细节。这些具体内容包括蛋白质之间的进化关系和结构相似性。缺乏这些专业知识的模型可能因数据处理错误而缺乏实际用途。
一种常见的数据处理错误类型是数据拆分之间的数据泄漏 。训练、验证和测试集不应共享相同(或接近相同)的数据点,因为这种重叠可能导致高估模型在新数据上的表现,从而影响模型的评估。在某些数据集中,所有数据点都足够不同,因此将可用数据随机拆分为不相交集是一种可行的策略。然而,处理蛋白质数据时,通常需要更复杂的拆分策略以避免进化数据泄露等问题。 处理蛋白质数据时,考虑多个层次的分离也很重要,例如由突变及其影响组成的层次。例如,可能需要确保训练集和测试集中不会出现相同的替换、位置或蛋白质。定义蛋白质相似性也是一项具有挑战性的任务,存在多种策略。许多策略涉及根据序列身份或相似度阈值对蛋白质进行聚类,然后确保在拆分数据时,给定簇的所有成员都被分配到同一集合。该策略对于构建带标记的蛋白质结构数据集尤为有用,因为此类数据集主要来源于大型冗余数据库,如PDB78(见表S2)。然而,在某些情况下,序列空间中的聚类可能不足。例如,远亲蛋白即使序列同源性较低,活性位点几何可能非常相似。聚类也可以在结构表示层面进行。虽然此类策略过去很少被使用,但随着Foldseek等新蛋白质结构搜索工具的出现,可能会变得更加普及。
机器学习解决的蛋白质工程任务
A.蛋白质的功能注释
了解蛋白质功能是蛋白质工程流程的基础。例如,在蛋白质适应度优化中,科学家从具有一定预期功能的已描述的野生型序列出发。 同样,在生物催化中,需要了解酶的功能,才能从合理的酶反应中组装出生物合成途径。
传统上,科学家们通过繁琐、耗时且昂贵的湿实验室实验来描述蛋白质的功能。然而,由于高通量DNA测序技术的发展,蛋白质序列的指数级增长数量已远超实验功能注释的能力范围;例如,Big Fantastic Database(BFD,表S2)迄今已包含25亿条序列 。功能注释对酶尤为重要。通过序列同源性和蛋白质结构域基序搜索,可以相对容易地实现宽层次注释(如酶族),但对酶底物和产物进行详细注释目前仍需实验表征。为了加速这一过程,近期有大量工作致力于开发函数注释的新颖计算方法。
开发的计算方法大量依赖先前描述的酶功能数据集进行训练。 例如,蛋白质家族和结构域的信息通常来源于Pfam、 SUPERFAMILY,或CATH数据库 。酶活性数据通常来自Rhea、BRENDA、SABIO-RK、91 PathBank、92 ATLAS、93和MetaNetX.94等数据库 。Expasy基础设施下的酶数据库提供酶委员会**(EC)编号,这是酶功能最常用的命名法。** EC编号是一种分层分类系统,将酶促反应分为四个细节层级,其中第四层最为详细。EC编号将具有相同酶活性的蛋白质归类,无论反应机制如何。上述数据库的数据也经过后处理并整理成ECREACT97或EnzymeMap等数据集,这将进一步促进计算模型的发展。
酶活性预测模型通常以酶氨基酸序列作为输入 ,目的是直接注释高通量DNA测序的输出。结构输入的纳入,得益于近期结构预测的突破,社区仍有待进一步探索。这些模型的输出通常分为三类 ,基于预测的分辨率。首先,最一般的模型预测蛋白质家族和结构域 。其次,EC类预测模型提供了更详细的酶活性估计 。最后,最全面的酶活性图景需要预测酶底物及其相关产物的模型 。一些近期的深度学习模型预测蛋白质家族和结构域。尽管这些模型对蛋白质研究至关重要,但它们在酶活性预测中的适用性有限,因为单个蛋白家族可以结合催化不同反应的酶。 为了满足蛋白质工程的需求,预测EC数值的模型似乎更具相关性, 因为它们能够捕捉催化活性。
多年来,社区尝试利用多重序列比对和位置特定评分矩阵(PSSM)或隐马尔可夫模型(HMM)配置、个基于k的最近邻分类器、支持向量机(SVM)以及深度学习来预测EC数。大多数方法将EC数的预测视为分类问题,导致在代表性不足的EC类别中表现不佳。最近的基于深度学习的方法通过对比学习方法来预测EC数,在预训练的蛋白质语言模型的序列嵌入基础上训练暹罗神经网络。由此产生的预测算法CLEAN能够更好地识别属于任何EC类别的酶序列,包括代表性不足的序列。CLEAN在计算机测绘中实现了电谱数预测的先进性能,并通过高效液相色谱-质谱结合酶动力学分析,对一组先前错误注释的卤素酶序列进行了体外实验验证。
EC类预测使得逆生物合成等后续应用成为可能。 例如,生物合成的规划通过利用基于变压器的神经网络预测底物的化学结构和所需酶EC数来实现。 其他已发表的深度学习模型也希望基于酶反应产物估算酶反应的底物。此类模型可用于根据酶类或底物/产物对优先选择酶。然而,将特定酶序列(即不仅仅是EC类)归入目标反应仍是未来开发的挑战。
最近,首批预测单个酶与底物/产物相互作用的通用模型已发表。这些基于DL的模型将蛋白质序列和小分子的成对作为输入,预测它们可能的相互作用。遗憾的是,所有一般的底物-酶相互作用模型均未在湿实验室实验中得到验证。此外,Kroll等人承认该模型对样本外底物的推广性较差。此外,酶家族特异模型在预测酶-底物相互作用方面优于一般模型。总之,通用酶-底物相互作用模型的实际适用性尚待确定。
B.监督学习以预测突变的影响
预测突变对各种蛋白质特性的影响,如溶解度、稳定性、聚集、功能和对映选择性,是蛋白质工程的另一个理想目标。从机器学习的角度来看,这意味着要有一个模型,以参考蛋白及其变体为输入,并预测所研究性质的变化作为输出。直观地说,这可以通过先对野生型蛋白的标记数据集进行监督学习(例如预测某蛋白的溶解度评分、结合能或熔点) ,然后应用训练好的模型独立预测参考蛋白及其变异体的标记,然后取两个预测分数的差值来实现。该策略的吸引力来自于可供训练的野生型蛋白大量注释数据集。例如,蛋白质结构倡议生成了庞大的数据集TargetTrack,常用于蛋白质溶解度预测。此外,通过液相色谱串联质谱获得的较新的蛋白质稳定性Meltome Atlas也被用于预测熔点。我们持续通过蛋白质序列预测高价值熔点的努力,促成了TmProt软件工具(https://Loschmidt.chemi.muni. CZ/TMPROT/)。大型标记数据集通常能提供足够的训练数据,支持强大的端到端深度学习。 然而,当训练数据集不包含突变时,少量替换通常会得到相似的预测标签(例如溶解度评分),这与实验中常见的剧烈变化形成鲜明对比。因此,将参考蛋白预测标记与其变异体之间的差值取的策略通常无法产生可靠的突变效应预测因子。
更有前景的方法是使用带标签的突变数据集进行训练。这种策略存在自身局限性,因为此类数据集不仅稀缺,而且在被探测的突变景观范围上也很稀疏(序列空间随着突变残基数量呈指数增长),并且偏向于多个过度代表的蛋白质。这些障碍严重阻碍了机器学习的使用,而机器学习高度依赖于高质量且覆盖关注领域的数据。因此,通常需要额外的数据整理和处理、训练方案的调整以及更全面且关键的数据评估。这些努力将是建立可靠机器学习流程以预测突变效应的关键第一步。
最丰富且多样化的突变数据来自大多数蛋白质工程研究中常规进行的一般生物物理特征分析,包括蛋白质表达性、溶解度和稳定性的测量。使用此类数据的主要挑战在于收集和整理:测量数据分散在文献中,且常被临时报告,因为它们通常是研究主要结果的补充。 这凸显了建立和维护带有蛋白质注释的数据库的重要性,以促进数据的发现和再利用。 例如,我们最近发布了SoluProtMutDB,目前已有近33,000条关于突变对100多种蛋白质溶解度和表达影响的标记条目。该数据库包含了最近用于开发溶解度预测器的所有数据点(见表2),这些预测器实现了约70%的正确预测比。
蛋白质稳定性测量是另一种广泛可用的生物物理数据,可用于机器学习。蛋白质稳定性通常以熔点(Tm)或折叠态与未折叠态之间的吉布斯自由能差(ΔΔG)来量化。存在多个蛋白质稳定性数据库,包括FireProtDB,158、ThermoMutDB,138和ProThermDB,135,这些数据常被用来训练机器学习预测变量,在应用于独立测试集时,达到了皮尔逊相关系数高达0.6,RMSE值达到1.5 kcal/mol。有趣的是,这些数据在过去十年几乎没有变化, 表明,可能需要定性范式转变以推动基于机器学习预测突变诱导蛋白质稳定性变化的应用。132 大量新数据集可能提供必要的推动力,一些令人振奋的研究正在收集此类数据:cDNA显示蛋白水解最近被用于测量约85万个单点突变体和选定的双点突变体的热力学稳定性,这些突变体涵盖354个自然结构域和188个新设计的蛋白质结构域长度分别为40和72个氨基酸。
突变后催化活性的变化也引起了机器学习研究者的关注。由于酶的机制极为多样,预测突变对酶活性的影响比预测蛋白质的稳定性和溶解度更具挑战性。其中一个丰富的突变数据来源是大规模深度突变扫描。 这些实验结合了高通量筛选和测序,通常通过比较蛋白质变异在应用特定选择前后的丰度来评分。这些数据集提供了各种酶局部突变景观的全面概述,因其不偏的突变覆盖,对机器学习具有重要价值。已有多个团队收集了多种深度突变扫描(DMS)数据集用于基准效应预测变量,我们预计随着更多数据集的出现,这一趋势将持续。此类重要研究包括最近发表的磷酸酶活性图谱,二氢叶酸还原酶,DNA聚合酶,以及棕榈酰乙醇酰胺转移酶。
由于其在选择过程中高度灵活,DMS可应用于多种酶功能。然而,其高通量也以限制研究中可使用蛋白质靶点数量为代价;通常只审查一个案例。同时靶向多种酶的愿望促使创建了另一个著名的酶活性变化数据库:D3DistalMutation。 该数据库包含来自UniProt注释的数据,代表了2130种酶中超过9万次突变效应。然而,其在机器学习中的潜力尚未被充分探索。其他蛋白质特性也可以作为蛋白质工程和机器学习的靶点。这些靶点通常基于感兴趣的酶选择,可能包括重要的功能性状,如底物特异性、酶对抗选择性、动力学常数、温度敏感性、或温度最优状态。此外,VariBench基准数据集中还包含若干可用于基于机器学习工具的突变数据集,这些工具关注蛋白质折叠速率、结合和聚集。近期精选示例工具列于表2。表S2对所描述数据库和数据集的概述。
C.设计变异的方法
虽然近年来预测突变效应的工具日益先进,但它们的简单形式只能为特定置换提供标签。然而,蛋白质工程流程的期望结果是拥有一份有前景的蛋白质变异样本清单,用于实验验证。因此,即使有可靠的机器学习工具用于预测替代效应,也必须解决提出有前景假设设计的问题。这个问题可能成为一个主要瓶颈,因为即使预测单点或多点突变效应很快,评估所有可能的突变组合仍然不可行。因此,对能够同时预测突变影响和缩小搜索空间的工具需求日益增长,这也是本小节的重点。
开发此类工具的一个主要挑战是找到高效减少多点突变体空间的方法。在对九个案例研究的分析中,Milton及其合著者发现,由于对应的单点突变的了解,无法预测一半多点突变影响酶性质的影响,相关复杂性则源于残基间的直接相互作用,以及在其他情况下的长程相互作用 这种常见的非加性行为, 这种现象被称为认知,促使开发了机器学习模型和组合优化算法,能够通过设计对多点突变体进行评分或搜索。以下已提出几种方法以克服这一挑战,相关内容可在综述中查阅。
其中一种方法是利用可靠的物理和进化工具制作一个用于筛选的变异库。即使是耗时的有潜力热点预选,也能大幅缩短下游机器学习评分和搜索的计算时间。例如,HotSpot Wizard 3.0通过使用多种基于序列和结构的过滤器识别可变残基,从而实现了热点的稳健选择,然后利用成熟的Rosetta和FoldX工具量化突变效应。另一个例子是FuncLib Web服务器,它利用进化守恒分析和基于Rosetta的稳定性计算计算有潜力的单点活性位点突变。 该工具对每种突变体组合进行了详尽建模,并按能量对它们进行排序。进化信息也可以被基于机器学习的模型捕捉,用来提出有前景的替代,例如条件似然高于野生型的氨基酸。
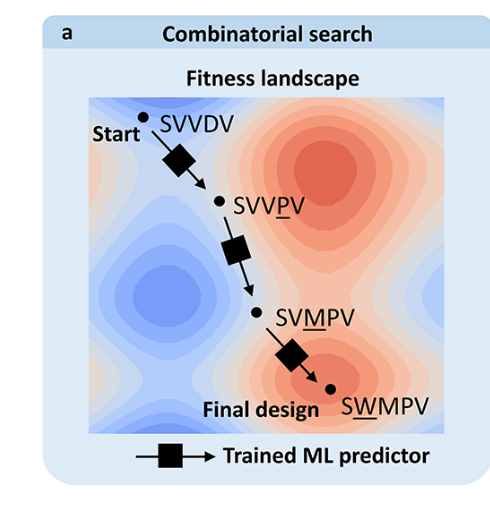
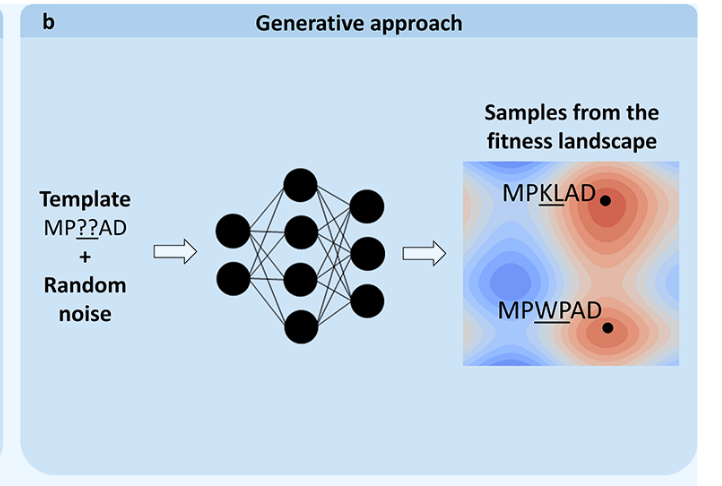
数学优化方法可以通过基于现有机器学习评分数据迭代生成新设计,在计算机模拟中生成有前景的蛋白质序列候选物(见图3)。其中一类方法利用机器学习预测器作为黑箱预言机来评估现有候选方案。该评估随后用于近似序列的"适应度",进而用于导航序列景观,并利用进化算法或模拟退火等工具生成新的候选序列。 然而,使用表示估计且简化分布的预言机来近似复杂的突变景观,可能会损害优化过程,阻碍最优解的发现。设计空间的自适应采样可以使用相反,为了获得更好的结果。其他替代方案是使用生成模型,或依赖所谓的白箱优化,即利用预测变量内部工作的知识来寻找最优解。例如,线性回归系数可以用来建议对输入的修改(突变),从而改变相应的特征朝预期方向发展。白箱方法将在可解释人工智能的背景下,第6.1节进一步讨论。
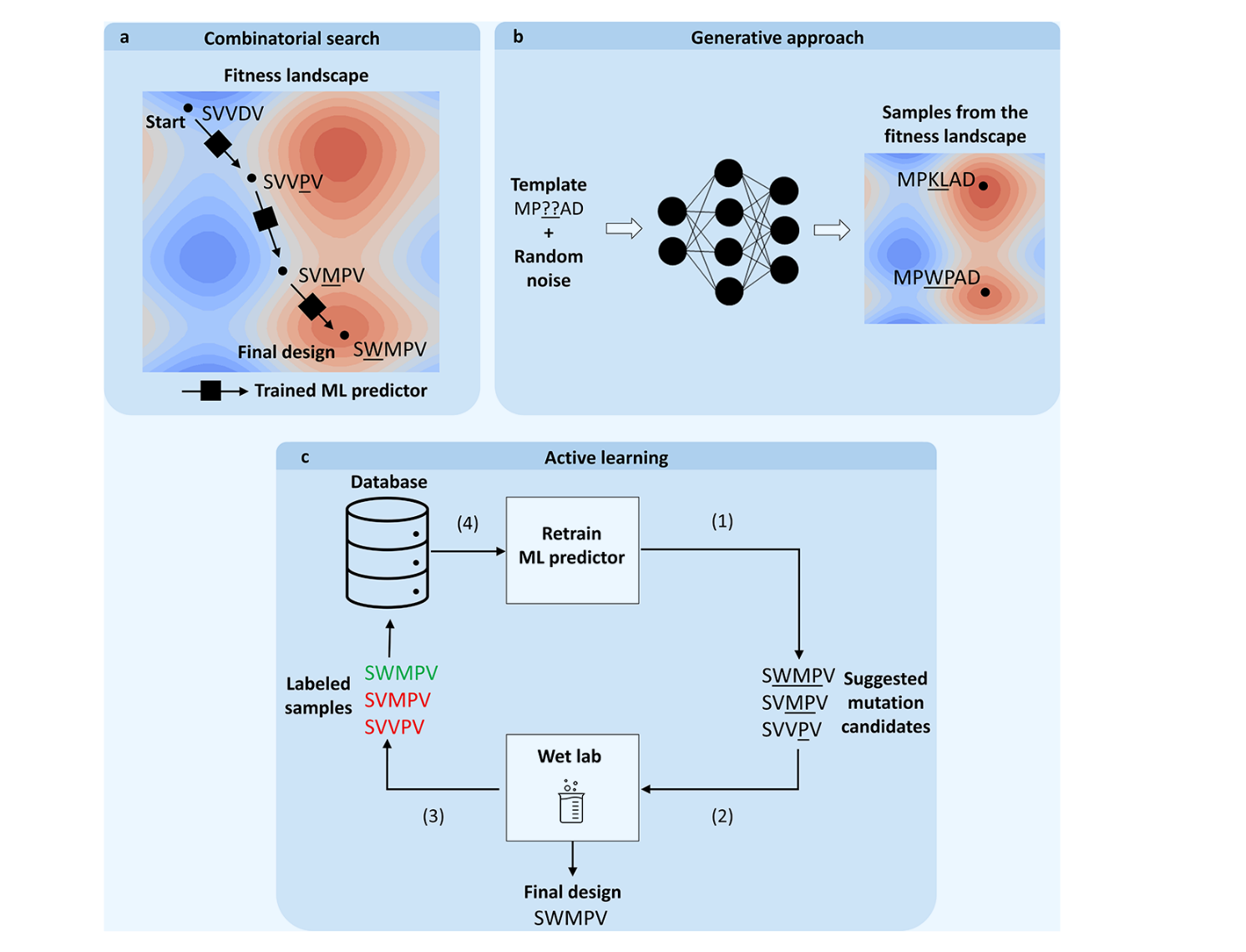
这与我之前的方向相关,简单讲一下我的理解: 总目标是去找高活性高稳定的酶突变体,但是酶的整个序列空间很大,所以采用计算机来模拟。先用机器学习的模型做预判,预判完了以后采用进化算法进行迭代,通过迭代出的好解再做实验验证。
黑箱优化就是模型只负责给一个适应度值,然后使用进化算法进行筛选,留下的突变再打分再筛选,最后得到理想解并进行验证。但是缺点就是,他是近似打分可能会出现局部最优的情况,错过真正的解。

白箱优化:使用ML模型去学习他的规律,就是知道怎么进化能有更好的分数,

第三个就是主动学习的循环:首先使用黑盒或白盒得到一批预测解,然后做实验进行更新真是数据,得到真实数据以后再放回原始数据里面进行进一步的迭代提高预测的准确率,进行一个正向循环。
虽然计算机模拟优化方法支持迭代生成有前景设计,但它们完全依赖预测变量正确评分突变景观中任一点的能力,这可能是个不切实际的强假设(非常认可这个观点 。另一种可能的替代方案(尽管成本更高且吞吐量更低)是直接将实验验证纳入优化循环。这种实验输入可以引导搜索算法找到突变景观中更有前景的部分,类似于定向进化。虽然此类高级搜索方法此前已被用于改进传统定向进化,但它们并未充分发挥实验特征描述中间变异的潜力。基于机器学习的主动学习方法通过迭代提取所有特征变体中的知识并挑选最有前景的变体,加速定向进化。依赖新的实验数据,只能预期有限数量的训练样本,这使得机器学习模型的选择限制在参数数较少的模型中,如多层感知器(MLP)仅有两层。主动学习领域的最新进展是GFlowNets的开发,该网络旨在在机器-专家循环中推荐多样且准确的候选方案,加速科学发现。使用此类网络的研究已证明其在设计小分子方面的潜力;然而,蛋白质设计的实用性仍需可靠验证。
用于探索酶及其他蛋白质变异的强大设计技术,通常不仅依赖实验和计算机模拟技术的结合,还结合多种计算机模拟方法,有时还会结合不同的机器学习技术。例如,聚焦训练机器学习辅助定向进化(ftMLDE)结合了无监督和监督训练方法,利用无监督聚类构建监督分类器的训练集。这些分类器随后用CLADE 2.0.等工具筛选有潜力的突变体。同样,对数百万序列进行无监督学习获得了名为UniRep的蛋白质表示,进一步无监督调优,获得了针对目标序列相关蛋白质的eUniRep(evotuned UniRep),从而为蛋白质整体及目标特定对象提供了丰富的特征集。这种表示方式使得数据高效的监督式学习成为可能,以指导计算机演化。 另一种方法是使用无监督的"概率密度模型"生成"进化密度评分,这一特征随后用于补充少量标记数据点,这些数据点上以监督方式训练光模型。有趣的是,这种方法被证明优于以无监督方式预训练的概率密度模型的监督微调。另一种方法将自监督的大蛋白语言模型与监督式结构到序列预测器结合起来,采用一种新的更通用框架------LM设计,声称能推动预测起始主链结构对应蛋白质序列的预测技术,有时称为"逆折叠"。虽然逆折叠并不显式地搜索突变全景,但可以通过输入现有的蛋白质结构和部分掩蔽序列,并利用逆折叠工具为掩蔽部分提出氨基酸,从而识别有前景的突变。
D.利用无标签数据集对突变进行评分
在过去十年里,大型语言模型(LLM)已成为解决从语言翻译到情感分析等自然语言处理问题的流行工具。这一重大范式转变源于认识到即使是未标记的数据也包含有用信息:分配假说认为,可以通过分析词语在不同文本中出现的频率和伴词组合来推断词语的含义。 类似地,在生物学中,我们可以将蛋白质视为基于"生命语言语法"的序列,这意味着氨基酸在特定位置的分布可以提供有价值的见解,帮助预测物质的影响对蛋白质功能的分析,从而减少对外部数据源的依赖。例如,Elnaggar 等人证明,LLM 生成的嵌入作为输入特征时,可以有效促进小型监督模型的开发,其预测能力可与依赖 MSA 进化信息的先进方法媲美。 此外,蛋白质语言模型 ESM-2 最近在 UniRef 数据库中的蛋白质序列上训练,预测了给定序列中 15% 被掩蔽氨基酸的预测。 这使得无需补充 MSA 即可直接利用序列信息大幅提升 B 细胞表位预测。ESM-2固有的注意力机制也可用于促进蛋白质结构预测。6我们在表3中提供了更多示例。此外,从语言模型中获得的嵌入,如ESM-1b,65 ESM-2,67 ProtT5,197和ProtTrans,已成为表示顺序数据的流行方式,使预训练ESM模型成为迁移学习的常用对象。这些模型的知识转移通过微调预训练权重(197,200)通过仅在已学习嵌入之上训练模型(保持预训练权重固定),以及在训练层之间引入适配器模块201实现参数高效微调来实现。学习到的嵌入的强大功能在完全无监督的环境中也能见证。更具体地说,ESM-1v66模型仅依靠蛋白质语言模型在预训练中学到的野生型氨基酸概率,无需后续微调,能够准确评分蛋白质变异。
大型深度学习模型在学习一般蛋白质性质方面表现出色。对于需要对特定蛋白质或蛋白质家族的详细理解,而非跨不同蛋白家族的通用模式的情况下,基于序列的模型可以从单一MSA学习分布模式。MSA已被证明是丰富的进化信息来源,例如识别功能重要的保守区域、插入或缺失,以澄清导致序列分歧的机制。
进化数据分析及突变型与野生型序列分配概率的比较也表明,基于机器学习的模型在深度突变扫描实验中比基于进化数据的既有方法更准确地预测突变的影响。例如,通过将MSA输入与先进的变换器架构结合,实现了出色的蛋白质结构预测性能。此外,Xie等人利用最大熵(MaxEnt)原理推断同源序列的统计能量,发现活性位点残基的统计能量与酶活性显著相关,而远离活性位点的残基则与蛋白质稳定性相关。Hsu等人观察到,结合基于MSA的机器学习模型进化密度分数与标记单热编码蛋白序列数据集的混合线性回归模型,在一系列蛋白质适应度预测任务中表现出优异表现,即使标记数据集大小在48−240个数据点范围内。 Illig等人报告了类似效应,针对50−250个小数据点。217 Ding等人证明变分自编码器可以捕捉系统发育关系 i在MSA潜空间的几何结构中,进一步证明了潜模型赋予序列的自由能量可用于预测突变引起的稳定性变化。基于这些发现,潜空间的几何结构最近被用来指导盐烯烷脱卤酶的设计。219 此外,通过探索序列信息背后的潜流形,我们可以发现原始潜空间嵌入中不易察觉的依赖关系。
尽管MSA有其优势,但也存在一些缺点。首先,创建包含足够进化相关序列以在关键氨基酸位置建立强模式的MSA可能很困难。其次,MSA的创建通常被视为一种手工而非系统化的过程,比对过程可能受到用户选择的敏感,包括替换矩阵的选择、间隙惩罚和迭代次数。错误的选择可能导致MSA残基比对不当,原因是比对过程中迭代次数过少,或在比对开始和结束时引入了大量缺口,这需要额外的预处理和MSA的修剪以达到最佳结果。此外,将新序列比对到MSA是一项具有挑战性的工作,需要仔细考虑比对参数和序列属性。尽管面临这些挑战,MSA仍然是研究生物序列关系和进化历史的重要信息来源。
近期研究还探讨了在蛋白质序列空间中两个截然不同尺度上研究模式的机会:庞大蛋白质序列宇宙中的一般模式和特定蛋白家族中序列模式的局部分布。这些工作将大型预训练模型与轻量级、易于重新训练的组件结合,实现了高效的家族特异性适应。例如,Sevgen等人设计了ProtT-VAE,这是一种融合了对4600万条UniRef50序列预训练的变换器和一种实现对感兴趣序列库进行微调的自编码器。213 在微调苯丙氨酸羟化酶(PAH)序列家族模型后,ProtT-VAE被用来预测具有多达100个突变的变异,使催化活性比人类野生型提高了2.5×。同样,Luo等人将预训练语言模型与MSA特异性直接耦合方法结合,捕获了一般蛋白质语法和蛋白质特异性上位作用。214 所得模型ECNet被用于工程化具有改进氨苄青霉素耐药性的TEM-1 β内酰胺酶变体。
在半监督环境中,未标记数据的利用已被尝试,例如在孤儿序列的二级结构预测或蛋白质序列的结构相似性预测中。 这两种方法都采用伪标记或定制相似度指标,使监督学习任务能够从大量初始未标记数据中获益。在某些情况下,即使是自监督方法也被视为半监督,因为它们从未标记的数据中学习到强大的表示,然后可以用小型标记数据集进行微调。然而,与半监督方法的区别在于,自监督方法建立在已有的监督学习方法论基础上,利用从大量未标记数据集自动生成的合成标签来构建监督代理任务。这种自监督方法推动了本节开头讨论的蛋白质语言模型的最新成功,且总体上比传统的半监督方法更为成功。
与序列数据库的爆炸式增长同步,蛋白质数据库在2023年4月达到了20万条条的里程碑,为自我监督的蛋白质结构学习提供了丰富的实验结构数据来源。许多深度学习模型已被拟合到PDB数据的大部分子集,以利用蛋白质结构的自然多样性。例如,MutCompute 在一个非冗余的 19K 蛋白质样本上训练,基于局部三维环境预测人工掩蔽的残基,使模型能够成功捕捉与蛋白质稳定性相关的表型景观。31 此外,Zhang 等人设计了多个通用任务用于图神经网络蛋白质结构的自监督预训练,从而提升了多种下游任务的性能。68 蛋白质上的自监督学习结构还被用来建议特定主链的蛋白质序列,并生成全新蛋白质结构。
E.利用蛋白质动力学比较突变
前述部分重点分析静态数据。然而,蛋白质是复杂且动态的系统,其构象变化和运动常常为仅靠静态结构研究无法获得的反应机制提供关键见解。捕捉蛋白质动力学的一种方法是研究其结构系群,这些集合在许多情况下可从现有的蛋白质结构数据库中获得。因此,有人提出基于结构的机器学习方法,例如基于此类数据的AlphaFold2,可能为蛋白质动力学提供见解。基于这一假设,Brotzakis及其合著者提出了利用AlphaFold2预测和FoldingDiff框架的加权程序27,用于生成无序肽的结构集合。在某些情况下,此类程序可能是计算量高的模拟的有用替代方案。然而,由于依赖MSA进化信息的AlphaFold2预测对单点突变不敏感,可能无法准确捕捉密切相关蛋白变异间的细微差异,因此它们不太可能用于比较密切相关的蛋白质变异的结构集合。
另一个选择是研究分子动力学(MD)轨迹。MD数据通过进行模拟,应用物理定律计算蛋白质中每个原子在特定时间步后基于当前时间点的三维结构的未来位置获得。最终的轨迹由一系列快照组成,捕捉了连续时间点的蛋白质配置。由于每个快照包含模拟系统中所有原子的坐标,模拟提供了大量数据,即使是一般大小的蛋白质。在已有系统知识的情况下,可以使用粗粒度模型来降低系统的维度,并可能捕捉主要的关注运动。227 然而,在大多数情况下,找到有效描述系统动态行为的所谓集体变量(CV)并不容易。有人提出,无监督学习方法可以帮助解决识别CV的问题,因为它们可以从原始MD轨迹中学习,而无需对所寻找的CV做出假设。一般来说,这类方法试图寻找足够丰富的低维表示,以重建原始(或时间偏移)输入。228 由此产生的低维投影可用于构建蛋白质动力学的简化模型。这通常通过马尔可夫态模型(MSM)实现,MSM可以将模拟分子的构象空间聚类为可处理数量的簇(态)。MSM假设这些状态之间的转变是马尔可夫的,即下一时间步的状态仅依赖于当前状态,无论之前的轨迹如何。该方法与端到端深度学习模型VAMPnet结合应用,用于为所研究系统寻找最优预测。 最近,CoVAMPnet框架230通过扩展VAMPnet方法,增加了可解释性功能和一种MSM对齐方法,便于两组模拟中MSM的比较而创建。如果应用于某一蛋白质的不同变异,CoVAMPnet有望用于评估判别突变对蛋白质动态的影响。然而,这一应用尚未被进一步探讨。
一般来说,识别与蛋白质变异之间生化差异相关的动态特征,比为单一蛋白质寻找低维表示要困难得多。解决这一问题的一种方法是DiffNets框架,该框架中监督自编码器被训练在MD轨迹上,以识别蛋白质变异间两对残基距离的最显著差异。另一种比较变异轨迹动力学变化的方法是直接分析由变分自编码器获得的构型低维投影分布。在这种方法中,相似的空间配置往往聚集在学习的低维空间的某些子空间中。例如,这种聚类可用于分析不同底物存在下催化位点的MD模拟,以识别驱动底物偏好的结构差异。尽管有这些有前景的研究,系统比较不同蛋白质变异MD轨迹的问题仍然大多未被探索,这为机器学习应用提供了引人注目的未来方向。
近期成功案例与经验教训
基于序列,基于结构,基于小数据集
这些例子值得注意,因为虽然很少有团队具备深度学习所需的专业知识或基础设施,但更简单且易于获取的机器学习方法仍可用于推动传统蛋白质工程流程的发展。同时,我们预计深度学习工具将逐渐变得更易接近和易于使用。
技术水平的重大缺口
2023年的 这里我就没看了
尽管上述应用令人振奋且案例有前景,但仍有若干重大知识空白待解决,以推动蛋白质工程迈向新高度。首先,许多蛋白质工程任务尚未受益于机器学习,包括预测插入物和非自然氨基酸的影响、创建同时针对多个靶点的预测器,以及预测突变对蛋白质相互作用的影响。其次,分子动力学与机器学习在蛋白质工程应用上的重大进展保持隔阂;在训练当前最先进的预测器时,动力学信息几乎从未被使用。第三,迫切需要建立黄金标准蛋白质数据集,因为这些基准显著加速了机器学习在其他领域的进展。最后,基于机器学习的工具影响力往往局限于一小部分方法开发者,因此有必要向更广泛的生物化学家和合成生物学家社区伸出援手。下面我们将更详细地讨论这些空白。
未来机遇
可信度与可解释的ai
在许多领域,包括医学和金融,机器学习系统必须具备可解释性和可解释性,以建立对算法的信任。理解预测背后的机制还能使工具进行更严谨的验证,或为后续决策提供线索。在此语境下,可解释模型是指能够洞察其预测和决策原因的模型,例如通过展示输入中哪些部分对预测影响最大。相反,可解释模型是指其内部预测过程能够被人类轻松理解的模型。这可以通过在决策树中明确的决策路径或在简单线性方程中设置易于理解的特征权重来实现。如果机器学习算法在数学上不复杂,它们通常本质上是可解释和可解释的。然而,随着算法的复杂,解读和解释他们的预测变得更加具有挑战性。这就是为什么像深度神经网络这样的模型常被称为黑匣子。可解释人工智能(XAI)领域试图通过分析、显著性图或文字来创造解释,帮助人类理解机器学习算法为何做出某些决策或预测。
近年来提出了多种XAI方法(见图4),并在两篇近期综合综述中进行了更详细的讨论。XAI的两种简单策略是使用自解释的白箱方法,以及检查变化输入如何影响黑盒网络的输出。特征重要性方法是一类著名的白箱方法,通过基于模型参数(如权重和系数)识别关键特征来实现可解释性。例如,在线性回归算法中,科学家可以根据相关系数的大小来确定每个特征的重要性。基于传播的方法是另一类白箱方法,常用于深度学习的类似目的。该类近期方法之一是分层相关性传播,利用在神经网络每个节点评估的传播规则,将预测从输出传播到输入。316 由于该方法依赖简单公式,不需要计算量大的采样,且在训练过程中对噪声和其他伪影具有相对的鲁棒性。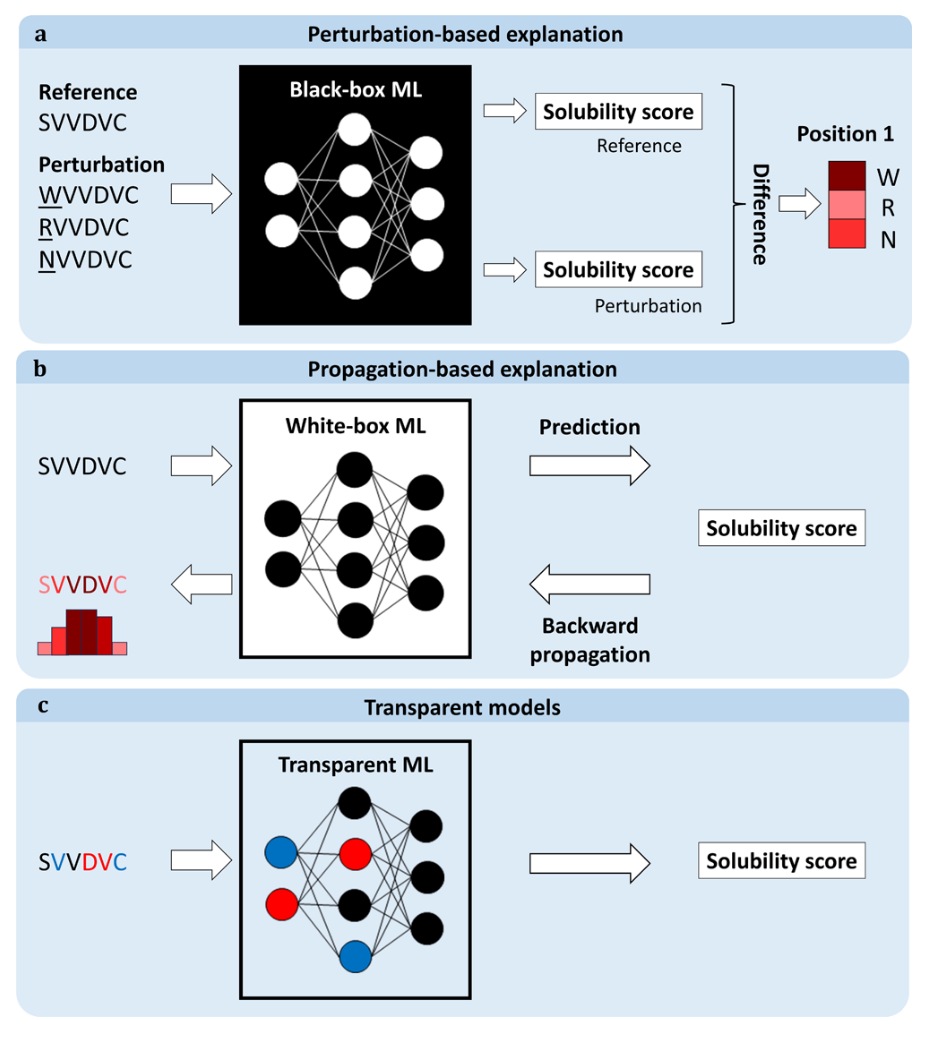
对于黑箱模型,通常通过分析输入输出行为来实现可解释性和可解释性。例如,Shapley值通过评估每个特征对输出的边际影响来估算其贡献。不幸的是,这种方法需要大量计算资源,且随着特征数量增加而扩展性较差。 另一种策略是用更易解释的类似物近似黑箱模型。这种方法以LIME方法为代表,它通过线性回归或决策树对决策边界进行局部近似,以可解释且忠实的方式模拟任何分类器的预测。另一种黑箱方法通过扰动输入数据并分析实际输出与受扰数据集输出之间的差异来解释模型预测。如果输入的某一特定扰动产生了输出差异较大,则数据集中被更改的部分标记为预测的关键部分。
另一种将可解释性融入机器学习管道的方法是在流程开发过程中追求可解释性,而非在预测器发布时,例如使用透明神经网络。320 此类神经网络设计使人类更易理解和理解。先验知识,如现有的生物学知识和实验数据,可以帮助科学家开发此类模型。例如,卷积神经网络可以设计用来学习对应已知蛋白质结构基序的滤波器。321 或者,也可以使用知识引发的神经网络,其中节点对应基于先验知识连接的蛋白质。
越来越多地应用于蛋白质工程的机器学习方法通常复杂且难以解释。由于机器学习设计的蛋白质和酶的湿实验室验证成本高且耗时,加强人工智能辅助蛋白质工程的信誉至关重要,并确保实验者能够确信这些方法能产出有良好成功率的设计。XAI通过提供对蛋白质工程中机器学习模型决策过程的洞见,可以增强公信力。323 它还能帮助科学家通过揭示错误和偏差来改进算法。因此,近年来利用XAI解释深度学习网络,吸引了药物发现、化学和蛋白质工程等领域的关注,包括主动配体搜索,325 预测酶EC数,326,并识别显示GPCR受体活跃与非活性状态转变的残基。
在蛋白质工程中,可解释人工智能主要用于机器学习模型的预测分析,旨在获得新的生物化学知识。具体来说,谭等人。 提出了ExplainableFold,328,这一概念旨在通过残基删除和残基替换,提升对基于深度学习的蛋白质结构预测模型(如AlphaFold)的理解。本质上,ExplainableFold的核心目标是揭示那些负责维持或改变折叠蛋白质结构的重要残基。更广泛地说,还提出应用如exBERT329等最初用于可视化变换子内部表示的工具,可用于蛋白质训练型变形子,突出氨基酸之间的关系。最终,可解释人工智能在蛋白质设计中的应用仍处于早期阶段,我们尚未看到其在蛋白质工程流程中的主要应用。
识别隐藏偏见
机器学习模型可能受到训练中使用的数据偏倚,这可能降低其整体准确性,或导致其性能在数据输入空间(例如蛋白质序列空间或蛋白质结构空间)中显著差异。在监督学习中,必须特别关注班级失衡问题,即当某些班级的训练样本多于其他班级时,就会出现这种情况。在这种情况下,模型可能会高估主要类别。在无监督学习中,这些偏差可能更难发现和量化。例如,在自然语言处理中,大型自监督语言模型通常训练于大量文本,这些文本往往来自互联网,缺乏全面的数据整理。如果不加以控制,这些模型据报道可能产生不公正或压迫性的语言,从而促进歧视、排斥和毒性。335 使用有偏的训练数据损害模型性能的风险,在酶工程中与其他领域同样重要。31 然而,与自然语言不同,酶学模型可能采用哪些不利偏见尚不明显, 因为没有快速简单的方法直接验证输出。因此,应保持责任和谨慎,尤其是在不同数据来源合并时,例如与被报告存在种族偏见的人类基因组数据库数据结合时。
酶数据中偏倚的一个典型例子出现在预测突变稳定性的任务中,预测变量输入原始且突变序列时,应报告对应预测的ΔΔG的单一数字。一般来说,天然蛋白质的随机突变更可能造成不稳定而非稳定,337,这种偏差常常传播到稳定性数据集,导致类似类失衡问题,导致不稳定突变被过度预测。为应对此类效应,利用突变稳定性的反对称性已成为标准,该原理由以下方程描述:338 ΔΔG(WT → Mut) = −ΔΔG(Mut → WT)。也就是说,突变残基时ΔG的变化等于假设逆突变引起的ΔG负变化。这一特性使得通过将突变序列(Mut)的人工逆突变添加到野生型序列(WT)中,来增强训练数据集,从而获得平衡的数据集。反对称性也可以被设计纳入,确保即使使用不平衡的训练集,该性质也能得到强化。339 有趣的是,解决突变后蛋白质稳定性变化预测问题的努力推动了其他敏感性研究的进展,例如使用约化化氨基酸字母表来解释训练数据中突变表示的偏差,131 结构敏感性,340 或在集合中使用晶体结构而非AlphaFold模型的"支架偏差"。276 另一个报告数据集偏倚的任务是基于结构的虚拟筛选。最近,作者通过基于机器学习的评分函数解决了这个问题,并特别注重特征重要性与人类知识的一致性;这迫使模型无论当前偏差如何都要学习相关特征,从而提高了推广性。
然而,在许多情况下,即使对研究任务有详细理解,也无法揭示和修正偏差的直接路径,因为问题往往源于研究者未察觉的数据点之间复杂的相互依赖关系。为解决此类问题,提出了多重校准的概念。342 多重校准的目标是使预测变量在不同数据子类间表现更为统一。然而,所提方法的复杂度相对于训练数据的可能子集数量呈线性,因此对训练样本数量呈指数级。为缓解这种高计算成本,其他研究提出了利用多校准和多准确性方法进行低度多重校准。343,344 这些概念目前主要在算法公平性领域研究,345,该领域主要关注基于机器学习的预测器处理人类个人数据。有趣的是,这些概念在酶学领域是否有用,比如通过确保不同蛋白质家族的预测变量表现相似,会有所帮助。