生物信息学数据库是生信分析的核心基础,不同数据库聚焦核酸 / 蛋白序列、基因组、基因表达、结构、通路、表观遗传、代谢组等不同方向,且多数经典数据库为国际公共库、数据免费开放。以下按功能分类整理最常用、最核心的生信数据库,兼顾通用性和实用性,适配零基础入门的生信分析需求,核心数据库做加粗标注,同时说明适用场景和特点:
一、核酸序列数据库(基础核心,存储核酸原始 / 注释序列)
这类数据库是核酸序列分析(如引物设计、序列比对、基因查找)的基础,三大国际库数据实时同步,是序列提交和查询的权威来源。
- GenBank(NCBI):美国 NCBI 旗下,最常用的核酸序列数据库,涵盖所有物种的 DNA/RNA 序列(基因组、cDNA、mRNA、miRNA 等),支持 BLAST 比对、序列下载。
- EMBL-EBI(欧洲分子生物学实验室):欧洲权威核酸库,与 GenBank、DDBJ 组成国际核酸序列数据库联盟,数据完全同步,配套的 ENA(European Nucleotide Archive)是其升级版本,支持更灵活的序列检索和批量下载。
- DDBJ(日本 DNA 数据库):亚洲核心核酸库,与前两者同步,无地域使用差异。
- RefSeq(NCBI):经过人工注释和验证的参考序列数据库,比 GenBank 的原始提交序列更准确、无冗余,是生信分析(如基因组比对、基因注释)的首选序列库,涵盖基因组、转录本、蛋白的参考序列。
- miRBase:microRNA 专属数据库,存储所有物种的 miRNA 前体、成熟体序列,以及 miRNA 的靶基因预测信息,是小 RNA 测序分析的核心库。
- piRNAbank:piRNA 专属数据库,聚焦小非编码 RNA piRNA 的序列和注释。
二、蛋白质序列数据库(存储蛋白序列 + 功能注释,适配蛋白分析)
核心数据库经过手动 / 自动注释,区分高质量注释库和海量无冗余库,是蛋白序列比对、功能预测的基础。
-
UniProt:全球最权威、最常用的蛋白质序列数据库(EBI/EMBL/ 瑞士生物信息学研究所联合维护),分三个子库,优先级:Swiss-Prot(手动注释,高质量、无冗余,核心首选) > TrEMBL(自动注释,海量序列,覆盖未手动注释的蛋白) > UniParc(所有蛋白序列的综合库,去重)。支持蛋白功能、结构、互作、疾病关联等多维度注释查询。
-
NCBI Protein:NCBI 旗下,与 GenBank 关联,蛋白序列与核酸序列一一对应,支持 BLASTp 比对,适合快速联动核酸和蛋白数据。
三、基因组数据库(存储完整基因组序列 + 全维度注释,适配基因组分析)
这类数据库整合了物种的完整基因组组装序列、基因结构、注释信息(启动子 / 外显子 / 内含子)、变异位点等,部分配套可视化浏览器,是基因组测序、基因定位、变异分析的核心。
通用全物种基因组库
- NCBI Genomes:涵盖原核、真核、病毒的所有参考基因组,注释信息全面,支持按物种、基因组版本检索下载。
- Ensembl(EBI/Sanger):可视化体验最佳的基因组数据库,核心覆盖脊椎动物,也拓展至植物 / 微生物,提供基因组浏览器、基因表达、变异数据的一站式分析,支持批量数据下载和在线工具(如 BLAST、Variant Effect Predictor)。
- Ensembl Genomes:Ensembl 的拓展版,专门针对非脊椎动物、植物、真菌、原核生物,弥补了 Ensembl 对非脊椎的覆盖不足。
- UCSC Genome Browser(加州大学圣克鲁兹):生信分析最常用的基因组可视化工具,不仅是数据库,更是分析平台 ------ 支持基因组比对、基因定位、表观遗传数据叠加,内置 BLAT(快速序列比对)、Table Browser(批量数据提取)等核心工具,人类 / 小鼠 / 果蝇等模式生物的注释最精细。
物种特异性基因组库(常用)
- TAIR:拟南芥专属基因组数据库,植物研究的经典参考。
- Rice Genome Annotation Project:水稻基因组核心库。
- IMG:微生物(原核 + 真菌)基因组专属库,涵盖宏基因组拼接的基因组。
四、基因表达数据库(存储基因表达原始数据,适配转录组分析)
这类数据库收录了芯片、RNA-seq、单细胞 RNA-seq的原始表达数据和标准化结果,支持数据挖掘、差异基因验证、表达模式分析,是转录组研究的重要数据来源。
- GEO(NCBI Gene Expression Omnibus):最常用的基因表达数据库,覆盖所有物种,收录芯片、RNA-seq、单细胞、甲基化等多组学数据,数据以Series(研究系列) 和Sample(样本) 组织,支持按基因、物种、疾病检索,可下载原始数据(SRA)和标准化表达矩阵。
- ArrayExpress(EBI):与 GEO 类似,是欧洲的基因表达数据库,数据标准更严格,与 EBI 的其他工具(如 limma)联动性好。
- TCGA/GDC(The Cancer Genome Atlas):肿瘤多组学核心数据库,最初为 TCGA,现整合至 GDC(Genomic Data Commons),收录人类 30 + 种癌症的基因组、转录组、蛋白质组、甲基化等多组学数据,是癌症生信分析的黄金数据来源。
- GTEx(Genotype-Tissue Expression):人类正常组织基因表达图谱,收录人体 50 + 种正常组织 / 细胞的基因表达数据,与 TCGA 互补,可用于分析癌症与正常组织的表达差异。
- Single Cell Portal(Broad 研究所):单细胞测序专属数据库,收录 scRNA-seq、scATAC-seq 等单细胞多组学数据。
五、蛋白质结构数据库(存储 / 预测蛋白 3D 结构,适配结构生物学分析)
核心库存储实验验证的 3D 结构,近年的 AI 预测结构库覆盖度极高,成为蛋白结构分析的主流工具,适配蛋白功能预测、分子对接、突变分析。
- PDB(Protein Data Bank):全球唯一的实验验证蛋白 3D 结构数据库,存储 X 射线晶体学、冷冻电镜(Cryo-EM)、NMR 获得的蛋白、核酸、复合物的 3D 结构,常用查询平台为RCSB PDB(最友好)、PDBe(EBI)。
- AlphaFold DB(DeepMind/EBI):AI 蛋白结构预测的核心库,基于 AlphaFold2 算法,预测了几乎所有已知蛋白质的 3D 结构(涵盖 UniProt 的 98% 以上蛋白),包括人类、模式生物、微生物的蛋白,结构精度接近实验水平,是无实验结构蛋白的首选参考。
- SWISS-MODEL:瑞士生物信息学研究所开发,既是同源建模工具(预测蛋白结构),也是结构数据库,收录了大量同源建模的蛋白结构,适合快速预测目标蛋白的 3D 结构。
六、通路与功能注释数据库(标注基因 / 蛋白的功能、通路,适配富集分析)
这类数据库是GO 富集、通路富集、基因功能预测的核心,通过标准化的术语和通路图谱,将基因 / 蛋白与生物学功能关联,是生信分析后结果解读的关键。
- KEGG(Kyoto Encyclopedia of Genes and Genomes):最经典、最常用的通路数据库,涵盖代谢通路、信号通路、生物合成、疾病关联等,同时整合了基因、蛋白、代谢物、酶的注释信息,支持通路可视化和富集分析。唯一不足是更新较慢,但仍是生信分析的首选。
- GO(Gene Ontology):基因功能的标准化注释体系,分为三个维度:分子功能(MF)(如酶活性、结合活性)、细胞组分(CC)(如细胞核、细胞膜)、生物过程(BP)(如细胞增殖、糖代谢),是GO 富集分析的核心库,所有生信工具均支持 GO 注释。
- Reactome:手动注释的通路数据库,更新快、注释更精细,涵盖信号通路、代谢、细胞过程,支持通路的动态可视化,适合补充 KEGG 的不足。
- COG/KOG:直系同源基因簇数据库,COG 针对原核生物,KOG 针对真核生物,通过同源基因聚类预测基因的功能,适合新基因 / 未知基因的功能注释。
- STRING:蛋白 - 蛋白相互作用(PPI)数据库,存储实验验证和预测的蛋白互作关系,支持构建 PPI 网络,同时整合了基因功能、通路注释,是互作分析的核心库。
七、表观遗传学数据库(存储 DNA 甲基化、组蛋白修饰等数据,适配表观分析)
聚焦DNA 甲基化、染色质开放状态、组蛋白修饰、非编码 RNA 调控等表观遗传信息,支持表观组学数据挖掘和验证。
- ENCODE(Encyclopedia of DNA Elements):人类 / 小鼠表观基因组核心数据库,收录染色质开放区、DNA 甲基化、组蛋白修饰、转录因子结合位点等数据,解析基因组的功能元件(如启动子、增强子)。
- Roadmap Epigenomics:表观基因组图谱数据库,补充 ENCODE 的不足,涵盖人类多种细胞系 / 组织的表观数据,支持表观状态的聚类和分析。
- UCSC Epigenome Browser:UCSC 的表观专属可视化工具,叠加 DNA 甲基化、组蛋白修饰、染色质开放数据,适合表观位点的定位和验证。
- MethBank:多物种 DNA 甲基化数据库,收录人类、小鼠、植物等的甲基化位点和甲基化水平数据,支持甲基化位点的检索。
八、代谢组数据库(存储代谢物信息,适配代谢组分析)
整合代谢物的结构、理化性质、浓度、代谢通路、检测数据,是代谢组测序(非靶 / 靶标)分析、代谢物鉴定的核心。
- HMDB(Human Metabolome Database):最全面的人类代谢组数据库,收录超 40 万种人类代谢物,包含结构、分子量、核磁 / 质谱数据、生物浓度、疾病关联,是非靶代谢组鉴定的首选。
- MetaboLights(EBI):代谢组学原始数据数据库,收录非靶 / 靶标代谢组的质谱 / 核磁数据,支持数据挖掘和验证。
- KEGG COMPOUND/Metabolome:KEGG 的代谢物子库,与 KEGG 通路直接关联,适合代谢物的通路富集分析,联动基因和代谢数据。
- LipidMaps:脂质组专属数据库,存储所有脂质分子的结构、分类、理化性质,是脂质组分析的核心库。
九、微生物 / 宏基因组数据库(适配微生物测序、宏基因组分析)
聚焦原核生物、病毒、宏基因组的序列、注释、丰度数据,是 16S rRNA 测序、宏基因组测序分析的核心。
- SILVA:最常用的核糖体 RNA 数据库,收录 16S rRNA、18S rRNA、ITS 的全序列,支持微生物物种分类和多样性分析,是 16S 测序物种注释的首选库。
- Greengenes:16S rRNA 专属数据库,经典的微生物分类库,适配 QIIME 等宏基因组分析工具。
- MGnify(EBI):宏基因组 / 宏转录组数据库,收录环境、人体、动植物相关的宏基因组数据,提供物种丰度、功能注释的标准化结果,支持数据挖掘。
- RDP(Ribosomal Database Project):核糖体 RNA 数据库,支持微生物的物种分类和序列比对,适合 16S 测序的物种注释。
- VIRUSite:病毒专属数据库,存储病毒的基因组、序列、蛋白注释,支持病毒组分析。
十、人类遗传与变异数据库(适配人类基因变异、疾病关联分析)
聚焦人类基因的SNP、Indel、拷贝数变异(CNV) 等变异位点,以及变异与疾病的关联,是人类基因组、肿瘤基因组分析的核心。
- dbSNP(NCBI):最常用的人类 SNP 数据库,收录单核苷酸多态性、小 Indel 等变异位点,标注变异的位置、频率、功能。
- 1000 Genomes Project:人类基因组变异数据库,收录全球 26 个人群的基因组变异数据,标注变异的人群频率,是变异分析的核心参考。
- OMIM(Online Mendelian Inheritance in Man):人类孟德尔遗传数据库,整合基因与人类疾病的关联信息,标注单基因病、复杂疾病的致病基因和突变位点,是疾病生信分析的关键。
- ClinVar(NCBI):临床变异数据库,收录经实验验证的临床相关变异(致病、疑似致病、良性),是肿瘤 / 遗传病变异解读的核心库。
十一、其他高频实用数据库
- PubMed:NCBI 旗下的生物医学文献数据库,虽非生信专属,但生信分析的结果解读、文献验证必用,可联动基因、蛋白、疾病的相关研究。
- GeneCards:人类基因综合数据库,整合了所有人类基因的序列、表达、功能、通路、疾病、药物等信息,是快速查询人类基因多维度注释的首选工具。
- DAVID:基因功能注释和富集分析工具,整合了 GO、KEGG、COG 等多个数据库,支持批量基因的注释和富集分析,适合生信分析的快速结果解读。
核心总结
入门必记的核心数据库:GenBank/RefSeq(核酸)、UniProt(蛋白)、UCSC/Ensembl(基因组)、GEO/TCGA(表达)、KEGG/GO(通路)、PDB/AlphaFold DB(结构)、SILVA(微生物);
生信分析的通用逻辑:从序列库获取参考序列→从表达库挖掘数据→用通路库做功能解读→用可视化数据库验证结果;
多数数据库支持批量下载和在线工具,无需单独下载全库,可通过工具(如 NCBI BLAST、UCSC Table Browser)按需提取数据。
GTDB(Genome Taxonomy Database) 是澳大利亚昆士兰大学主导开发的微生物基因组分类权威数据库,旨在基于全基因组数据构建标准化、系统发育一致的细菌与古菌分类体系,解决传统基于 16S rRNA 分类的局限性,是宏基因组、微生物多样性研究的核心工具。
- 核心定位与优势
全基因组驱动:以细菌 120 个单拷贝标记基因(bac120)、古菌 122 个单拷贝标记基因(ar122)为基础,结合全基因组平均核苷酸一致性(ANI)与相对进化分歧度(RED),实现更客观、可重复的分类,覆盖大量未培养微生物。
系统性分类:提供域、界、门、纲、目、科、属、种的完整层级,包含 6900 + 物种、2000 + 属、600 + 科,覆盖 73 万 + 基因组(Release 10-RS226,2025 年 4 月)。
工具配套:提供GTDB-Tk命令行工具,支持批量基因组分类、系统发育树构建,适配宏基因组组装基因组(MAGs)与分离株基因组分析。 - 关键应用场景
宏基因组测序物种注释(替代传统 16S rRNA 数据库)
微生物新物种分类命名参考
微生物进化关系解析
临床 / 环境微生物组多样性研究 - 访问与资源
官网:https://gtdb.ecogenomic.org/
数据更新:定期发布(如 R214、R226 等版本),同步 NCBI RefSeq/GenBank 基因组数据。
结合微生物研究 + 生物信息学入门的核心需求,同时覆盖生信分析通用基础环节 + 微生物专属分析 + 多组学拓展,以下按分析流程环节分类整理最常用、易安装、高实用性的生信工具,所有工具均适配Linux 服务器(优先 Conda 一键安装,零基础可直接执行),标注核心功能、适用场景、安装 / 使用要点,同时关联此前的微生物分析流程,兼顾通用性 + 微生物针对性,方便按分析需求快速选型。
核心说明
1.所有工具优先推荐 Conda 安装(适配之前搭建的 Linux 环境,无版本冲突);
2.工具分基础通用工具(所有生信分析必备)和微生物专属工具(聚焦基因组 / 宏基因组 / 微生物群落),同时补充转录组 / 可视化拓展工具;
3.标注 **★为入门必学 ** 工具,是你开展微生物生信分析的核心工具包。
一、序列质控与预处理工具(所有生信分析的第一步,★核心基础)
处理高通量测序原始数据(fastq),完成质量检测、低质量序列过滤、接头去除、去重,输出高质量干净序列,适配基因组 / 宏基因组 / 转录组测序数据。

二、序列比对与映射工具(基因组 / 转录组核心,★基础)
将质控后的干净序列比对到参考基因组 / 参考序列,生成比对结果(bam/sam),用于变异分析、基因定量、基因组组装验证等。

三、基因组组装与质控工具(微生物分离株 / MAGs 核心,★重点)
将比对后的序列 / 质控后原始序列组装为完整 / 部分基因组序列(contig/scaffold),并完成组装结果的质量评估(完整性 / 污染率),是微生物基因组分析的核心。
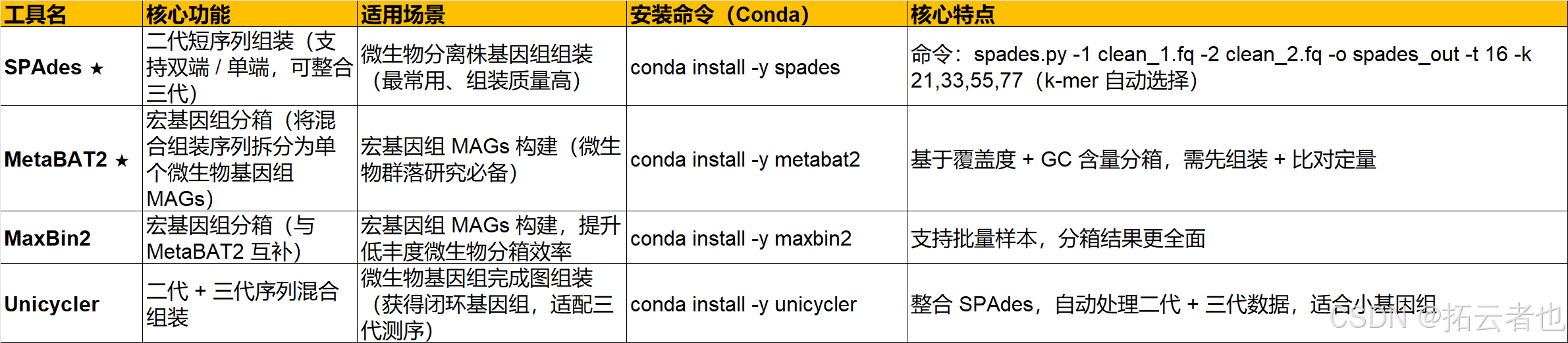
3.2 组装质量质控工具

四、微生物物种分类与注释工具(★核心,你的研究重点)
完成微生物基因组 / MAGs / 宏基因组的物种分类(域 / 门 / 纲 / 目 / 科 / 属 / 种)和群落物种丰度定量,是微生物多样性研究的核心,均为你此前流程中用到的工具。
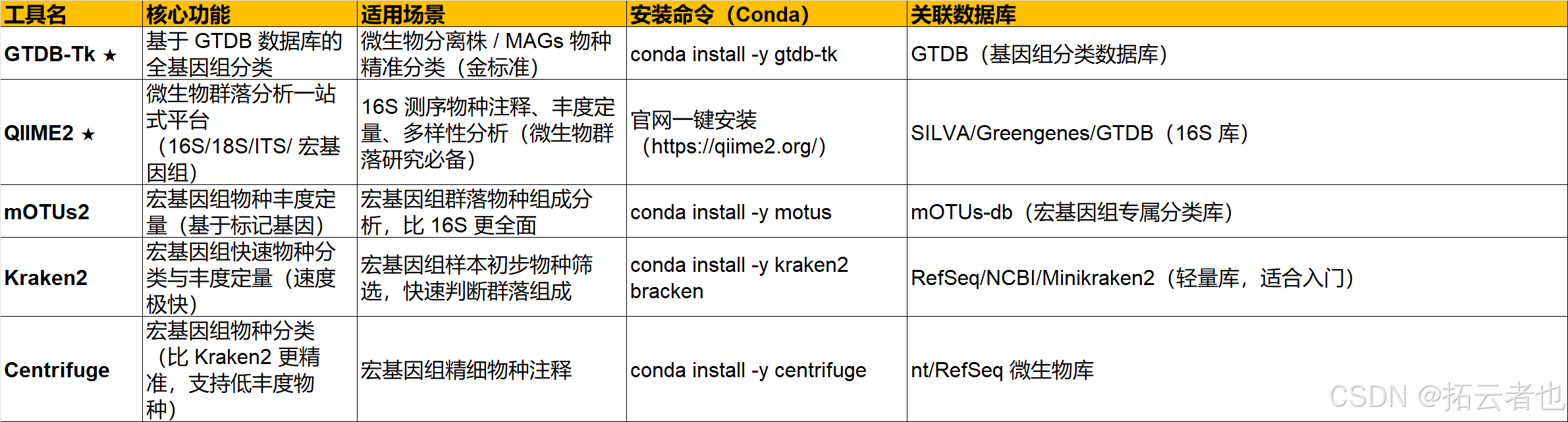
五、基因预测与功能注释工具(★核心,生信分析结果解读)
从微生物基因组序列中预测编码基因 / 非编码 RNA,并完成基因的功能注释(COG/GO/KEGG/CAZy),是解读微生物代谢潜力 / 生态功能的关键。
5.1 基因预测工具
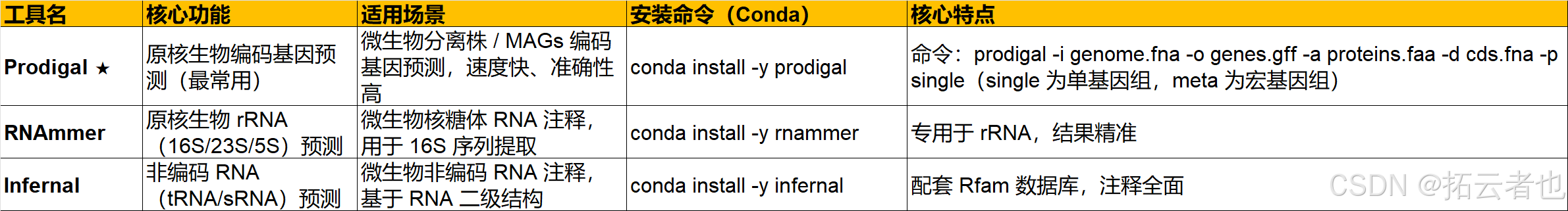
5.2 功能注释工具(均为你此前流程核心工具)

六、微生物专项分析工具(聚焦抗性 / 噬菌体 / 代谢通路,你的研究拓展)
针对微生物研究的专项需求(抗性 / 噬菌体 / 代谢通路 / 水平基因转移)开发的工具,是深入解析微生物生存策略 / 进化机制的关键。
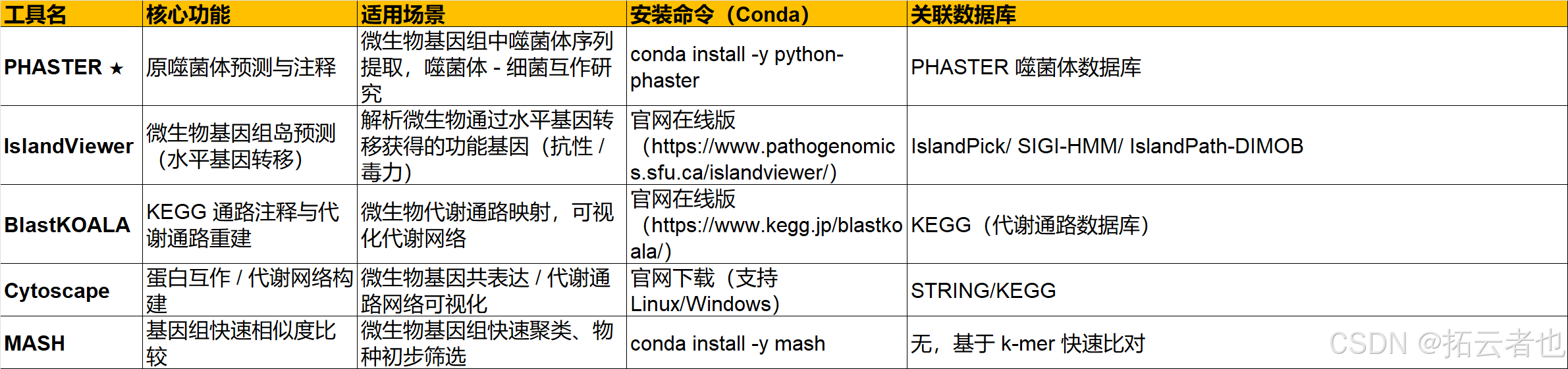
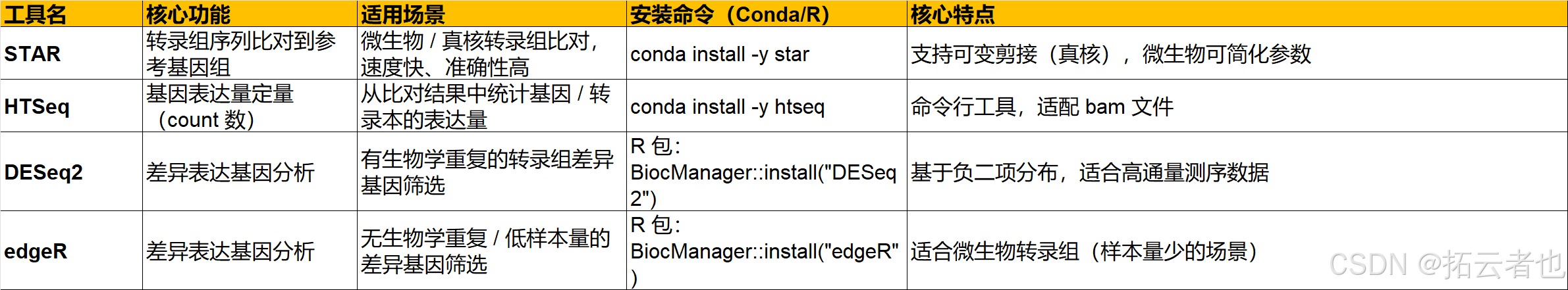
七、转录组分析工具(拓展,适配微生物转录组 / RNA-seq)
若你开展微生物转录组测序(RNA-seq) 研究,以下为核心工具,完成基因表达定量、差异基因分析。
八、可视化与数据挖掘工具(★零基础友好,结果展示核心)
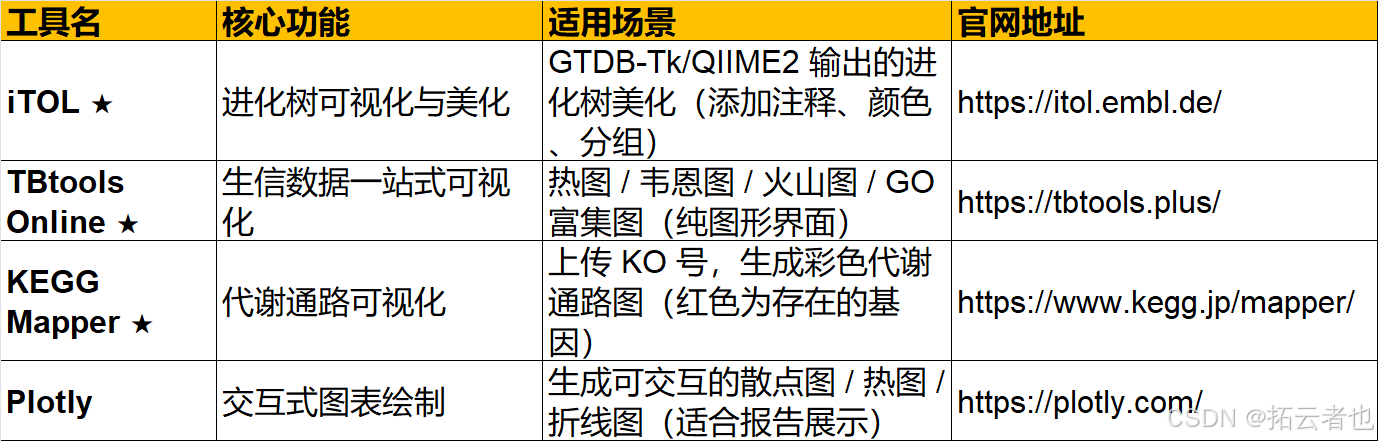
将生信分析的纯文本结果转化为可视化图表(进化树 / 热图 / 气泡图 / 通路图),是论文 / 报告结果展示的关键,分在线工具(零基础)和本地工具(Linux/Windows)。
8.1 在线可视化工具(★零基础必用,无需代码)
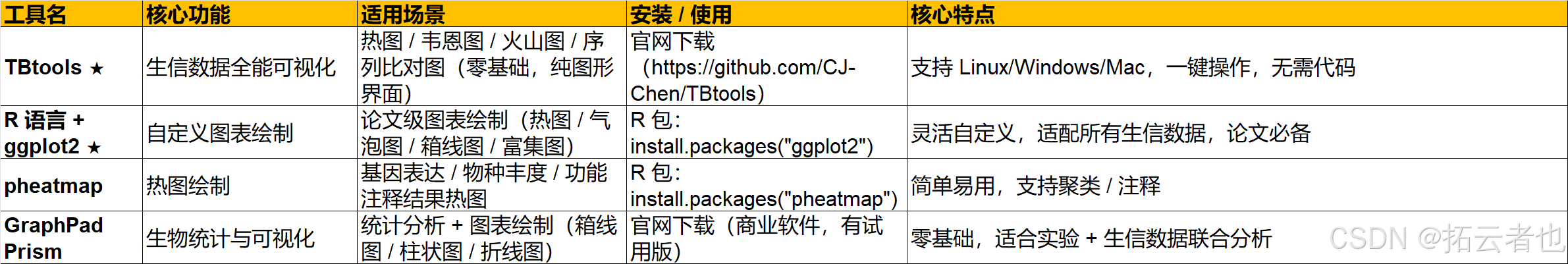
8.2 本地可视化工具(Linux/Windows,适配批量数据)
九、批量分析与流程管理工具(Linux 服务器必备,提升效率)
当样本量较大时(如 10 + 基因组 / 宏基因组样本),用于批量执行分析流程、管理工具依赖、自动化分析,避免逐个样本运行命令,提升效率。

零基础学习建议
- 先掌握 Conda 环境管理:所有工具的安装 / 激活是基础,熟练使用conda create/activate/install/remove;
- 从单样本入手:先拿 1 个微生物分离株基因组练手,完成质控→组装→分类→注释的完整流程,再拓展批量样本;
- 优先掌握命令行基础:Linux 常用命令(cd/ls/cp/mkdir/awk/grep)是生信分析的前提,熟练后能大幅提升效率;
- 可视化工具优先用 TBtools/iTOL:零基础友好,无需代码,快速出图,满足论文 / 报告展示需求。