2026.2.4
该研究提出了MedLAM,一个利用少量模板扫描实现3D医学图像中任何解剖结构精准定位的自监督基础模型,并将其与SAM整合为MedLSAM,显著减少了3D医学图像分割的标注需求,同时保持了与全监督模型相当的性能。
Title 题目
01
MedLSAM: Localize and segment anything model for 3D CT images
3D CT图像的MedLSAM:定位并分割任何模型
文献速递介绍
02
现有基础模型在通用计算机视觉任务中表现出色,但在医疗影像领域,特别是3D医学图像定位方面,仍存在显著空白。尽管SAM及其医学变体在分割方面有潜力,但其对手动提示(如点或边界框)的依赖导致3D数据标注成本高昂。因此,本文旨在开发一个仅需少量用户输入即可定位任何目标结构的3D医学定位基础模型,并将其与SAM结合以实现自动分割。
Aastract摘要
02
基础模型在医学图像分析中展现出巨大潜力,但专门用于医学图像定位的模型仍存在空白。为解决此问题,我们引入了MedLAM,一个3D医学定位基础模型,它仅需少量模板扫描即可准确识别身体内的任何解剖部位。MedLAM在一个包含14,012张CT扫描的综合数据集上,采用统一解剖映射(UAM)和多尺度相似性(MSS)两项自监督任务进行训练。此外,我们通过将MedLAM与"分割一切模型"(SAM)集成,开发了MedLSAM。这一创新框架仅需在几个模板上提供三个方向的极点标注,即可使MedLAM定位图像中的目标解剖结构,并由SAM执行分割,显著减少了3D医学成像场景中SAM所需的手动标注量。我们在涵盖38个不同器官的两个3D数据集上进行了广泛实验。研究结果表明:(1)MedLAM仅使用少量模板扫描即可直接定位解剖结构,达到与全监督模型相当的性能;(2)MedLSAM在手动提示下,性能与SAM及其专用医学适应模型接近,同时最大程度地减少了整个数据集上对大量点标注的需求。此外,MedLAM有潜力与未来的3D SAM模型无缝集成,为增强分割性能铺平道路。我们的代码已公开。
Method 方法
03
本文提出了MedLSAM框架,它包含两个核心组件:自动定位算法MedLAM和自动分割算法SAM。MedLAM是一个3D定位基础模型,通过统一解剖映射(UAM)和多尺度相似性(MSS)两个自监督任务进行训练,将不同个体的扫描图像映射到共享的3D隐式解剖坐标系,并细化像素级局部特征的相似性,以实现任何解剖结构的精准定位。MedLSAM的推理过程首先由MedLAM识别目标结构的六个极点来生成3D边界框,然后可以采用全补丁定位(WPL)或子补丁定位(SPL)策略生成2D边界框提示,最后由SAM或MedSAM进行精确分割。
Discussion讨论
04
本研究提出了MedLAM,一个用于3D医学图像中识别任何解剖结构的基础定位模型,并结合SAM构建了MedLSAM自动化分割框架,以减少医疗图像分割的标注工作量。尽管MedLSAM在减少标注方面表现出潜力,但其性能尚未完全达到全监督模型(如nnU-Net)的水平,尤其在处理具有显著解剖异常的器官时仍存在局限性。未来的工作应致力于增强MedLAM的通用性,并将其与新兴的通用医学人工智能(GMAI)应用结合,以缩小与全监督模型之间的性能差距,并提升医疗影像分析的效率和灵活性。
Conclusion结论
05
MedLAM是首个3D医学图像定位基础模型,能够以少量支持样本实现与全监督模型相当或更优的定位性能。与SAM结合的MedLSAM框架在显著减少手动标注需求的同时,在38个不同器官上展示了有竞争力的分割性能。该研究为3D医学图像分析中自动化基础分割模型的发展铺平了道路,并通过公开代码和模型促进了医学影像领域基础模型的研究。
Results结果
06
MedLAM在StructSeg HaN和WORD两个数据集上的landmark定位和器官定位任务中,表现优于DetCo、Mask R-CNN等方法,尤其在StructSeg HaN数据集上显著超越了全监督模型。对于小器官,MedLAM的IoU分数相对较低,但通过扩展3D边界框仍可有效进行后续分割。MedLSAM在多数器官上接近手动提示的分割性能,并优于UniSeg和nnDetection提示方法。尽管与全监督模型(如nnU-Net)仍有差距,但SPL策略显著提升了具有形态变异器官(如脑干和脊髓)的DSC分数。MedLAM在FLARE2023数据集上对正常和异常器官的定位表现稳定,但在肾脏等肿瘤体积较大的器官上效果有所下降。
Figure 图
07
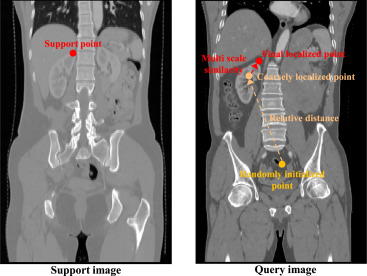
图1. 医学定位任意模型(MedLAM)推理阶段的结构。该过程涉及将一个代理从查询图像中的随机初始化位置向目标地标移动,由3D相对距离和多尺度特征向量引导。
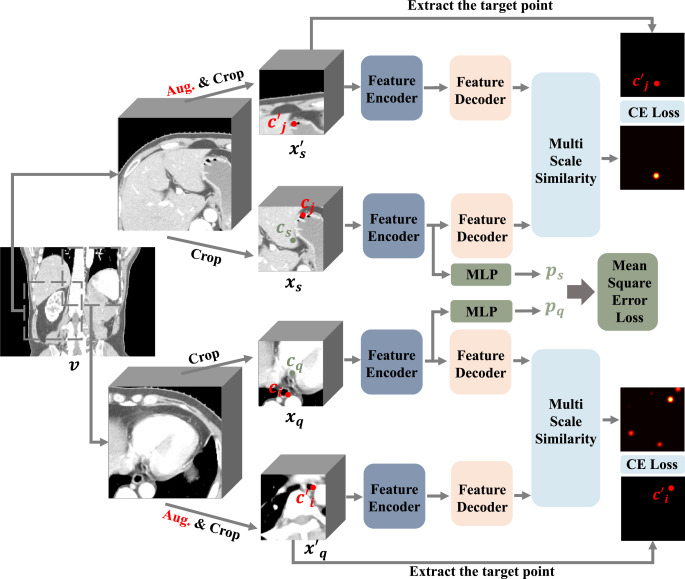
图2. MedLAM的学习过程。我们首先从扫描中随机提取两个大的图像块。然后,我们从这两个大的图像块及其增强版本中随机裁剪一个小的图像块,生成两对图像块,即原始图像块对xq,xs和增强图像块对xq′,xs′。所有这些图像块都通过MedLAM,训练目标包括两方面:(1)统一解剖映射(UAM):通过预测原始图像块xq和xs之间的相对距离,MedLAM将不同个体的图像投影到一个共享的解剖坐标空间。(2)多尺度相似性(MSS):确保在原始图像块xq,xs和其增强对应物xq′,xs′中,对应相同区域的特征之间的相似性最大化。
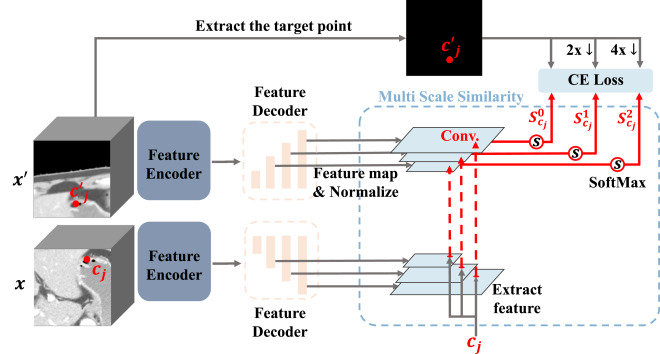
图3. 多尺度相似性(MSS)的详细信息。原始小图像块x和增强图像块x′都经过特征编码器和特征解码器,以获得归一化的多尺度特征图。然后,我们提取点cj的多尺度特征向量,计算其与x′对应特征图的相似性,并在每个相似性图中应用Softmax操作以获得最终的概率图集合{Scj0,Scj1,Scj2}。最后,概率图受交叉熵损失的约束。
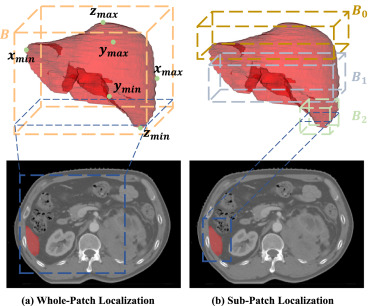
图4. 全补丁定位(WPL)和子补丁定位(SPL)策略的比较。(a) WPL通过识别目标解剖结构的六个极点,生成一个单一的最小3D边界框(B)。(b) SPL将目标结构划分为多个片段,每个片段都有自己的局部3D边界框(Bi),从而在每个切片上获得更准确的表示。
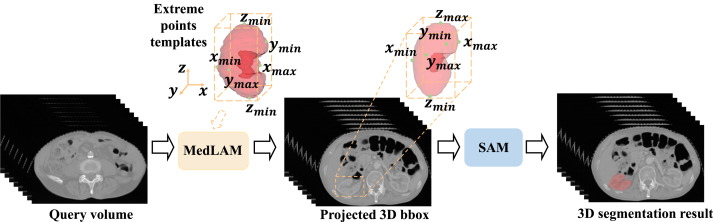
图5. MedLSAM的整体分割流程如下。给定任意大小的数据集,MedLSAM首先应用MedLAM来识别任何感兴趣解剖结构的六个极点(在z、x和y方向)。这个过程会生成一个包含目标器官或结构的3D边界框。随后,对于这个3D边界框内的每个切片,会导出一个2D边界框,表示3D边界框在该特定切片上的投影。这些2D边界框随后被"分割一切模型"(SAM)用于执行目标解剖结构的精确分割,从而实现整个分割过程的自动化。
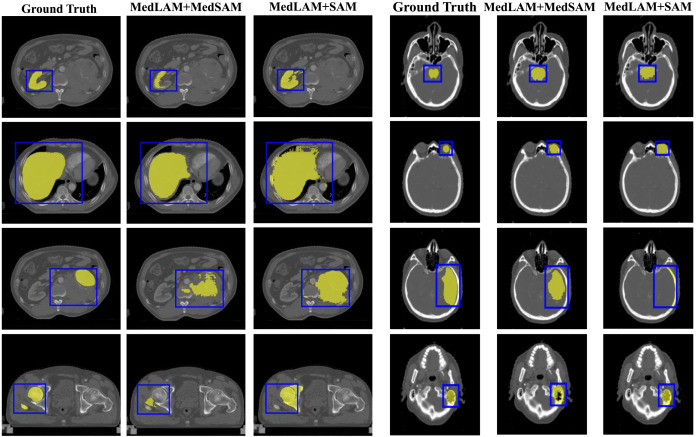
图6. 使用预训练的MedSAM和SAM,在经过MedLAM地标定位后,在WORD和StructSeg头颈部数据集上进行分割结果的可视化示例。