2026.2.13
该研究提出一种结合解剖学引导提示和跨模态自对齐预训练的全身PET-CT乳腺癌分割框架,通过生成器官伪标签和特征对齐,显著提升了原发性和转移性病灶的分割精度,优于现有多种SOTA方法,尤其在小而分散的转移性病变分割上表现出色。
Title 题目
01
Anatomy-guided prompting with cross-modal self-alignment for whole-body PET-CT breast cancer segmentation
解剖学引导的全身PET-CT乳腺癌分割与跨模态自对齐
文献速递介绍
02
乳腺癌是全球女性癌症相关死亡的主要原因,转移进展是患者预后的主要决定因素。PET-CT结合了代谢和解剖信息,是转移性乳腺癌成像的基石。然而,准确分割PET-CT图像中的转移性乳腺病灶面临多重挑战:(i) 转移性病灶小而分散,外观异质,代谢活动多样;(ii) PET和CT模态间分辨率、对比机制和噪声特性差异,有效融合困难;(iii) 缺乏专家标注的器官分割数据,限制了利用解剖上下文改进病灶检测。现有深度学习方法,如CNN和Transformer,在处理长距离依赖和计算成本方面存在局限。生理性高摄取区(如脑、心、膀胱)的存在也导致PET-CT成像中的歧义。为解决这些问题,本文提出了一种结合解剖学引导提示和跨模态对齐预训练的新颖框架,通过知识蒸馏生成器官伪标签,并利用自对齐跨模态预训练增强PET和CT图像的特征整合,旨在更精确地分割小而分散的转移性病灶。
Aastract摘要
02
准确分割PET-CT图像中的乳腺癌对于精确分期、监测治疗反应和指导个性化治疗至关重要。然而,转移性病灶体积小、分布分散,加上注释数据稀缺和模态间异质性阻碍了有效信息融合,使这项任务极具挑战。本文提出一种新颖的解剖学引导跨模态学习框架来解决这些问题。该方法首先通过教师-学生学习范式生成器官伪标签,作为解剖学提示指导癌症分割。接着引入一种自对齐跨模态预训练方法,通过掩蔽3D补丁重建在共享潜在空间中对齐PET和CT特征,实现有效的跨模态特征融合。最后,利用预训练编码器权重初始化分割网络的编码器,并通过基于Mamba的提示编码器和Hypernet控制的交叉注意力机制整合器官标签,实现动态解剖特征提取和融合。值得注意的是,该方法在两个包含原发性乳腺癌、转移性乳腺癌及其他类型癌症分割任务的数据集上,均优于八种最先进的方法,包括基于CNN、Transformer和Mamba的方法。
Method 方法
03
本文提出的方法包含三个主要阶段:(1) 生成器官特异性伪标签作为癌症分割的解剖学提示。利用教师-学生学习框架,通过知识蒸馏从不同器官特异性数据集生成伪标签,涵盖原发(脑、肺、乳腺、肝脏、肾上腺、脊柱、骨盆)和次要(心脏、肾脏、膀胱、脾脏)器官,解决了注释数据有限的问题。 (2) 进行自对齐跨模态预训练,以获取用于整合PET和CT模态信息的编码器权重。该阶段采用3D U-Net基于掩蔽自编码器(MAE)的策略,对PET和CT图像进行独立掩蔽(75%掩蔽率),通过重建丢失信息,在共享潜在空间中学习模态不变特征,从而弥合PET-CT域差距。 (3) 将解剖学提示整合到分割网络中,并加载预训练的编码器权重,以增强模型利用跨模态信息进行更准确癌症分割的能力。具体来说,Mamba 기반提示编码器将三维器官标签token化并利用双向扫描策略捕获全局体积依赖性,生成器官聚焦表示。Hypernet控制的交叉注意力(HyperCA)机制动态地将这些解剖学提示与3D U-Net骨干网的深层特征融合,通过超网络生成注意力参数,使其能够适应性地聚焦于最相关的解剖区域。
Discussion讨论
04
本文提出的框架通过结合解剖学引导提示和跨模态自对齐预训练,显著提升了全身PET-CT乳腺癌分割的准确性,尤其对于小而分散的转移性病灶。器官伪标签生成策略有效解决了标注数据稀缺的问题,并作为强大的语义过滤器,通过Grad-CAM可视化证实,能有效抑制PET图像中生理性高摄取区域(如膀胱、大脑)的干扰,将注意力精准导向病理病灶。自对齐跨模态预训练则学习了模态不变特征,弥合了PET和CT之间功能与解剖信息差异。Mamba 기반提示编码器和HyperCA机制动态融合了解剖学特征和图像特征,克服了标准Transformer处理三维医学数据计算量大的问题,并实现了内容自适应的特征融合,有效避免了过分割和欠分割。消融研究进一步验证了各个模块的互补性及有效性。此外,与通用基础模型(如SAM-Med3D)的比较突显了本方法在特定医学图像分割任务中的专业化优势。尽管本文方法表现优异,但未来的工作将进一步细化病灶与解剖结构的关系建模,并探索额外的外部提示以扩展框架在其他癌症类型和成像模态上的适用性。
Conclusion结论
05
本文提出了一种用于PET-CT成像中乳腺癌分割的新型解剖学引导跨模态学习框架。该方法结合了解剖学引导、跨模态自对齐特征对齐和动态提示整合,显著提高了原发性和转移性癌症的分割准确性。通过教师-学生框架生成的器官伪标签作为解剖学提示,解决了注释数据有限的问题。自对齐跨模态预训练通过掩蔽3D补丁重建使PET和CT特征在共享潜在空间中对齐,确保了模态不变特征的学习。Mamba 기반提示编码器和HyperCA机制则实现了动态解剖特征提取和融合,提高了对小而分散转移性病灶的分割精度,并缓解了过分割和欠分割问题。实验结果表明,该方法在挑战性转移性病灶分割方面达到了71.88%的DSC,优于最佳竞争模型1.96%,并且在消融研究中也证明了预训练和解剖学引导提示的互补优势,比基线提高了15%。未来的工作将继续优化病灶-解剖关系建模,并探索更多外部提示以扩大框架的应用范围。
Results结果
06
在器官伪标签生成方面,学生模型在多个器官上取得了高Dice系数(DSC),例如肺部98.70%,肝脏97.05%,乳腺88.23%,表明其能有效学习分割各类器官。在癌症分割任务上,提出的方法在SYSU-Breast数据集上取得了最佳性能。对于原发性乳腺病灶,DSC为89.27%,IoU为80.62%,优于第二名SwinUNETRV2超过1.11%和1.79%。对于转移性病灶,DSC为71.88%,IoU为60.86%,优于第二名STU-Net 1.96%和1.54%。在AutoPET III数据集上,该方法也表现强劲,DSC为69.33%,IoU为58.94%,优于最佳模型STU-Net约5%和2%。所有结果的p值均小于0.01,表明统计学显著性。定性比较显示,本文方法在SYSU-Breast数据集上避免了其他SOTA方法的过分割问题,在AutoPET III数据集上解决了欠分割问题,提供了更精确的边界定义。模型复杂性分析表明,本方法在DSC提升15%的情况下,参数量和FLOPs仅略有增加,优于STU-Net。消融研究证实了"解剖学引导提示"模块带来4.42%的DSC提升(推理时间增加0.25s),"预训练"模块带来8.69%的提升(无额外推理成本),且Mamba 기반编码器的双向扫描策略效果最佳。对不同输入模态的分析强调了PET和CT融合的必要性。与SAM-Med3D等基础模型相比,本文方法在处理小而分散的转移性病灶和多样化癌症类型方面表现出显著优势。
Figure 图
07
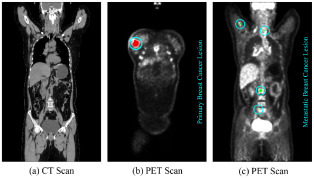
图1. 合作医院核医学科乳腺癌患者PET-CT扫描的2D冠状切片病例示例,包含专家标注。红色突出显示原发性乳腺癌,绿色表示转移性病灶。CT图像重采样以匹配PET图像分辨率,CT窗宽(Hounsfield单位,HU)为[-1000, 2671],PET窗宽(标准化摄取值,SUV)为[0, 21]。
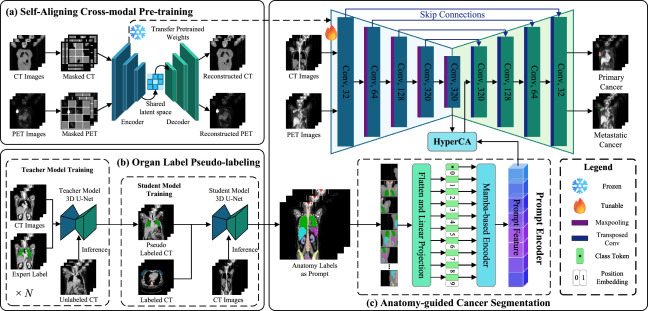
图2. 所提出方法的概述。我们对PET-CT图像中原发性和转移性乳腺癌分割的方法包含三个步骤。(a) 自对齐跨模态预训练:使用3D U-Net重建掩蔽的PET和CT图像,使编码器能够学习模态不变特征并弥合PET-CT域差距,增强特征泛化能力。预训练的编码器权重随后用于初始化分割编码器。(b) 器官标签伪标签化:教师-学生框架将器官分割知识从不同数据集转移到学生模型。学生模型分割多个器官,为病灶分割提供解剖学提示。(c) 解剖学引导的癌症分割:最后,我们引入了一个提示驱动的癌症分割模型。骨干网是3D U-Net,与(a)中相同,其编码器用(a)中的预训练权重初始化。同时,(b)中的器官标签通过基于Mamba的提示编码器编码为提示特征,并通过Hypernet控制的交叉注意力(HyperCA)机制在瓶颈处整合,以引入解剖学先验进行准确的癌症分割。最后,解码器输出预测标签。
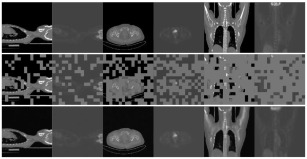
图3. 跨模态掩蔽自编码器重建。第一行:原始图像。第二行:掩蔽图像,其中掩蔽区域用灰色着色。第三行:从未掩蔽补丁重建的图像。每两列显示不同掩蔽比率(从左到右分别为25%、50%、75%)下的CT和PET结果,其中每对的第一列显示CT,第二列显示PET。
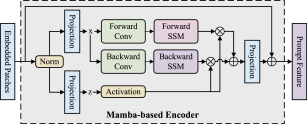
图4. 基于Mamba的编码器架构。该编码器结合了前向和后向卷积与选择性状态模型(SSM),以高效捕获多尺度解剖模式。
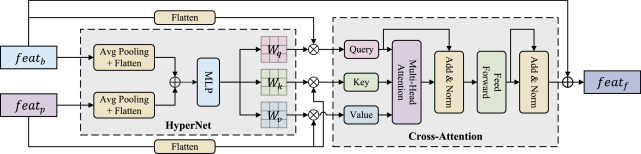
图5. HyperCA机制概述。HyperCA机制包含一个超网络(HyperNet)和一个交叉注意力(Cross-Attention)模块,实现内容自适应的特征融合。超网络根据3D U-Net编码器处理的PET和CT图像提取的池化瓶颈特征(featb)以及提示编码器编码的器官标签提示特征(featp)动态生成注意力参数(Wq, Wk, Wv)。然后,交叉注意力模块利用这些动态生成的参数将输入特征转换为查询、键和值表示,接着进行多头注意力操作和残差连接。此过程产生融合特征featf。
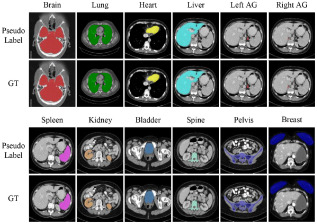
图6. 器官分割结果比较。不同颜色表示不同的解剖结构。AG:肾上腺;GT:真实值。
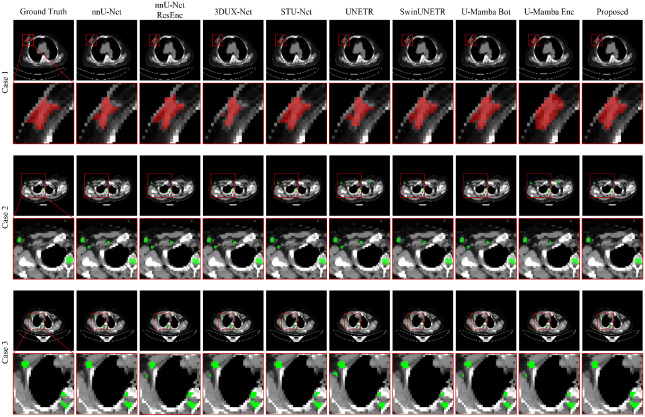
图7. SYSU-Breast数据集上的癌症分割可视化。案例1展示了原发性乳腺癌,病灶区域用红色突出显示;案例2和案例3描绘了转移性乳腺癌,病灶用绿色标记。为了增强清晰度并便于详细比较,红色框包围的区域被放大。
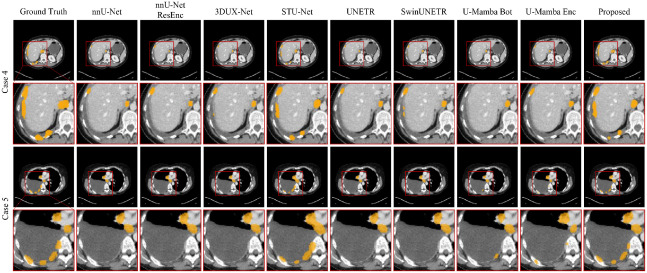
图8. AutoPET III数据集上的癌症分割可视化,病灶区域以橙色显示。红色框包围的区域被放大以提高可见性并支持全面比较。
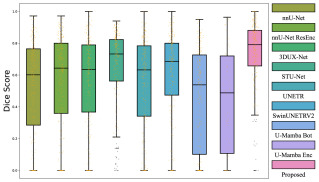
图9. 转移性乳腺癌分割的DSC箱线图,比较了不同方法。黑点表示异常值,小橙点表示单个样本。
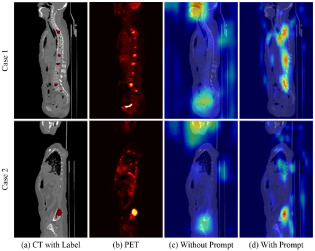
图10. 注意力图的Grad-CAM可视化。行显示脊柱(案例1)和髂骨/肺(案例2)转移。列:(a) 带有器官标签的CT;(b) PET输入;(c) 无提示(受脑/膀胱生理性摄取干扰);(d) 有提示(聚焦于真实病灶)。